- 方法gydF4y2Ba
- 188滚球软件
- 发表:gydF4y2Ba
主持的褶皱变化和分散估计与DESeq2 RNA-seq数据gydF4y2Ba
基因组生物学gydF4y2Ba体积gydF4y2Ba15gydF4y2Ba文章编号:gydF4y2Ba550年gydF4y2Ba(gydF4y2Ba2014年gydF4y2Ba)gydF4y2Ba
文摘gydF4y2Ba
比较高通量测序分析,基本任务是统计数据的分析,比如读计数每个基因在RNA-seq,系统在实验条件变化的证据。小复制数字、离散性、大动态范围和异常值的存在需要一个合适的统计方法。我们提出gydF4y2BaDESeq2gydF4y2Ba微分计算数据的分析方法,使用收缩估计对分散体和褶皱的变化提高估计的稳定性和可解释性。这允许一个更定量分析关注的力量,而不是仅仅的微分表达式。的gydF4y2BaDESeq2gydF4y2Ba包是可以在gydF4y2Bahttp://www.bioconductor.org/packages/release/bioc/html/DESeq2.htmlgydF4y2Ba。gydF4y2Ba
背景gydF4y2Ba
高通量测序(高温超导)技术的快速应用基因组研究已导致需要统计方法定量评估实验之间的区别。一项重要任务是RNA序列(RNA-seq)数据的分析,目的是寻找差异表达的基因在样品组。这个任务是通用:方法通常也适用于其他比较高温超导化验,包括染色质免疫沉淀反应测序,染色体构象捕获,或者计数观察类群在宏基因组研究。gydF4y2Ba
除了需要考虑具体的统计数据,如non-normality和依赖的方差的意思是,一个核心的挑战是小数量的样品在典型的高温超导实验——通常只有两个或三个复制/条件。推论方法,对待每个基因分别遭受从缺乏动力,由于不确定性较高的类内方差估计。在高通量分析,可以克服这个限制在基因池信息,具体来说,利用假设方差不同基因的相似性测量在同一实验(gydF4y2Ba1gydF4y2Ba]。gydF4y2Ba
许多方法等微分表达式分析RNA-seq数据执行信息共享在基因方差估计(或者等价,色散)。gydF4y2Ba刨边机gydF4y2Ba(gydF4y2Ba2gydF4y2Ba]、[gydF4y2Ba3gydF4y2Ba]温和派每个基因朝着一个共同的分散估计估计在所有基因,或向当地估计从基因表达强度相似,使用加权条件的可能性。我们的gydF4y2BaDESeqgydF4y2Ba方法(gydF4y2Ba4gydF4y2Ba)检测和纠正色散估计太低通过建模的依赖色散对所有样本的平均表达强度。gydF4y2BaBBSeqgydF4y2Ba(gydF4y2Ba5gydF4y2Ba)模型上的分散的意思是,与分散的平均绝对偏差估计用来减少离群值的影响。gydF4y2BaDSSgydF4y2Ba(gydF4y2Ba6gydF4y2Ba]使用贝叶斯方法提供了一个估计分散的单个基因异质性的色散值不同的基因。gydF4y2BabaySeqgydF4y2Ba(gydF4y2Ba7gydF4y2Ba),gydF4y2BaShrinkBayesgydF4y2Ba(gydF4y2Ba8gydF4y2Ba]估计先验贝叶斯模型对所有基因,然后提供后验概率或错误发现率(罗斯福)微分表达式。gydF4y2Ba
的转录组数据的比较分析中最常见的方法是测试的零假设之间的对数褶皱变化(利物浦)治疗和控制基因的表达是零,即,基因并不是治疗的影响。常微分分析的目标是产生一个基因列表传递多个测试调整,排名gydF4y2BaPgydF4y2Ba价值。然而,小的变化,即使统计上非常重要,可能不是最有趣的候选人进一步调查。排名由褶皱变化,另一方面,是复杂的吵闹利物浦估计较低的基因数量。此外,称为显著差异表达基因的数量取决于样本的大小和其他方面的实验设计是生物学的实验——和well-powered实验常常生成一个压倒性的长串的支安打(gydF4y2Ba9gydF4y2Ba]。因此,我们开发了一种统计框架基于稳定促进基因的排名和可视化效果大小的估计(利物浦),以及测试的微分表达式对用户定义的阈值的生物意义。gydF4y2Ba
在这里,我们现在gydF4y2BaDESeq2gydF4y2Ba,我们的继任者gydF4y2BaDESeqgydF4y2Ba方法(gydF4y2Ba4gydF4y2Ba]。gydF4y2BaDESeq2gydF4y2Ba集成方法论进步与一些新颖的特点,以促进一个更定量分析比较使用收缩估计RNA-seq数据分散和褶皱的变化。我们展示的优势gydF4y2BaDESeq2gydF4y2Ba通过描述许多应用程序的新功能可能与萎缩褶皱变化和标准误差的估计,包括改进的基因排序和可视化,假设测试高于和低于一个阈值,和正规化的对数转换质量评估和overdispersed计数数据的聚类。我们另外比较gydF4y2BaDESeq2gydF4y2Ba的统计力量与现有的工具,显示我们的方法具有较高的灵敏度和精度,同时控制假阳性率。gydF4y2BaDESeq2gydF4y2Ba是可用的(gydF4y2Ba10gydF4y2Ba)作为R / Bioconductor包(gydF4y2Ba11gydF4y2Ba]。gydF4y2Ba
结果与讨论gydF4y2Ba
模型和标准化gydF4y2Ba
的起点gydF4y2BaDESeq2gydF4y2Ba是一个计算矩阵分析gydF4y2BaKgydF4y2Ba每个基因的一行gydF4y2Ba我gydF4y2Ba并为每个样本一列gydF4y2BajgydF4y2Ba。矩阵的条目gydF4y2BaKgydF4y2BaijgydF4y2Ba显示的顺序读取的数量已经明确地映射到一个基因样本。注意,尽管我们在本文参考项读入基因,本文提供的方法也可以应用到其他类型的高温超导计数数据。对于每一个基因,我们适应一个广义线性模型(GLM) [gydF4y2Ba12gydF4y2Ba)如下。gydF4y2Ba
我们读计数模型gydF4y2BaKgydF4y2BaijgydF4y2Ba后为负二项分布(有时也称为gamma-Poisson分布)的意思gydF4y2BaμgydF4y2BaijgydF4y2Ba和色散gydF4y2BaαgydF4y2Ba我gydF4y2Ba。的意思是作为一个数量gydF4y2Ba问gydF4y2BaijgydF4y2Ba,基因的互补脱氧核糖核酸片段的浓度成比例的样本,按比例缩小的归一化因子gydF4y2Ba年代gydF4y2BaijgydF4y2Ba,也就是说,gydF4y2BaμgydF4y2BaijgydF4y2Ba=gydF4y2Ba年代gydF4y2BaijgydF4y2Ba问gydF4y2BaijgydF4y2Ba。对于许多应用程序,同样的常数gydF4y2Ba年代gydF4y2BajgydF4y2Ba可用于所有基因样本,然后占测序深度样本之间的差异。估计这些gydF4y2Ba大小的因素gydF4y2Ba,gydF4y2BaDESeq2gydF4y2Ba包提供了median-of-ratios方法已经用于gydF4y2BaDESeqgydF4y2Ba(gydF4y2Ba4gydF4y2Ba]。然而,它可以有利于计算gene-specific规范化的因素gydF4y2Ba年代gydF4y2BaijgydF4y2Ba进一步考虑的技术偏见,如不同来源依赖GC含量,基因长度或类似,使用公布的方法(gydF4y2Ba13gydF4y2Ba]、[gydF4y2Ba14gydF4y2Ba),这些可以提供。gydF4y2Ba
我们使用的漠视与对数链接,gydF4y2Ba,设计矩阵元素gydF4y2BaxgydF4y2Ba小gydF4y2Ba和系数gydF4y2BaβgydF4y2Ba红外gydF4y2Ba。在最简单的情况下,两组之间的比较,如治疗和控制样本,设计矩阵元素指示是否一个示例gydF4y2BajgydF4y2Ba是否治疗,和漠视,返回系数表明整体基因的表达强度和日志吗gydF4y2Ba2gydF4y2Ba褶皱的治疗和控制之间的变化。使用线性模型,但是,还提供了灵活性分析更复杂的设计,通常是有用的在基因组研究[gydF4y2Ba15gydF4y2Ba]。gydF4y2Ba
经验贝叶斯收缩分散估计gydF4y2Ba
类内变化。,v一个r我一个b我lity between replicates, is modeled by the dispersion parameterαgydF4y2Ba我gydF4y2Ba用于描述通过计数的方差gydF4y2Ba。色散参数的准确估计gydF4y2BaαgydF4y2Ba我gydF4y2Ba统计推断的微分表达式是至关重要的。研究样本量大,这通常不是一个问题。然而,对于控制实验,样本大小往往是小(实验设计中至少有两个或三个复制是常见的和合理的),导致高度可变色散估计为每一个基因。如果直接使用,这些噪声估计会妥协的微分表达式测试的准确性。gydF4y2Ba
一个合理的解决方案是在基因共享信息。在gydF4y2BaDESeq2gydF4y2Ba,我们假设基因相似的平均表达强度有类似的色散。我们在这里解释的概念方法使用由底部为例的数据集gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba]RNA-seq数据两种不同菌株的老鼠和Pickrell数据集gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba17gydF4y2Ba为人类lymphoblastoid细胞株)与RNA-seq数据。数学细节,看材料和方法。gydF4y2Ba
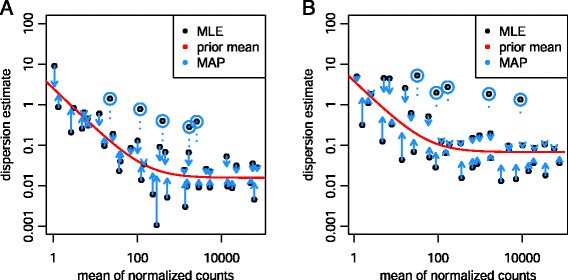
我们第一次分开每个基因治疗,估计gene-wise分散估计(使用最大似然),仅仅依靠数据的每个基因(黑点图gydF4y2Ba1gydF4y2Ba)。接下来,我们确定这些估计的分布的位置参数;允许平均表达强度的依赖,我们适合一个平滑的曲线,红线图如图所示gydF4y2Ba1gydF4y2Ba。这提供了一个准确的估计预期的色散值给定的基因表达强度但不代表个体基因从这个总体趋势的偏差。然后我们缩小gene-wise分散估计对预测的值曲线来获得最终的色散值(蓝色箭头)。我们使用一个经验贝叶斯方法(材料和方法),可以收缩的强度(i)取决于估计距离真正的色散值往往是适合和自由度(ii):随着样本量的增加,收缩强度的减少,最终变得微不足道。我们的方法因此占gene-specific变化在某种程度上,提供这些信息的数据,而艾滋病估计拟合曲线和测试在信息丰富的设置。gydF4y2Ba
收缩估计的色散。gydF4y2Ba情节的分散估计平均强度表达式gydF4y2Ba(一)gydF4y2Ba的底部gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba在两组,]数据集有六个样品gydF4y2Ba(B)gydF4y2Ba5从Pickrell样本gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba17gydF4y2Ba]数据集,配件只有截距项。首先,gene-wise毫升得到只使用各自的基因的数据(黑点)。然后,一条曲线(红色)适合毫升捕捉dispersion-mean依赖的总体趋势。这符合之前被用作意味着第二轮评估,导致最后地图估计色散(箭头)。这可以被理解为一个收缩(蓝色箭头)的噪声gene-wise估计红线所代表的共识。黑色的点检测中被标记为蓝色为分散局外人,而不是缩小朝前(收缩将按照虚线)。为了清楚起见,只显示基因的一个子集,丰富对色散离群值。额外的文件gydF4y2Ba1gydF4y2Ba:图S1显示相同的数据,但分散的基因。地图,最大gydF4y2Ba后验gydF4y2Ba;大中型企业,最大似然估计。gydF4y2Ba
我们所使用的方法是相似的gydF4y2BaDSSgydF4y2Ba(gydF4y2Ba6gydF4y2Ba顺序),两种方法估计的先验分布符合真正的色散值,然后提供最大gydF4y2Ba后验gydF4y2Ba(地图)作为最终的估计。它不同于以前的实现gydF4y2BaDESeqgydF4y2Ba使用最大的拟合曲线和gene-wise分散估计最终估计和倾向于高估了分散体(附加文件gydF4y2Ba1gydF4y2Ba:图S2)。的方法gydF4y2BaDESeq2gydF4y2Ba不同于gydF4y2Ba刨边机gydF4y2Ba(gydF4y2Ba3gydF4y2Ba),gydF4y2BaDESeq2gydF4y2Ba估计的先验分布的宽度数据,因此基于观察到自动控制的收缩量的属性数据。相比之下,默认的步骤gydF4y2Ba刨边机gydF4y2Ba需要一个user-adjustable参数,gydF4y2Ba之前的自由度gydF4y2Ba,重基因估计和个人的贡献gydF4y2Ba刨边机gydF4y2Ba的色散。gydF4y2Ba
注意,在图gydF4y2Ba1gydF4y2Ba许多基因gene-wise分散估计曲线以下最后估计大幅提高。收缩过程从而有助于避免潜在的假阳性,可由于色散的低估。另一方面,如果个体基因的色散远远高于gene-wise分散的分布估计的其他基因,然后收缩会导致最终估计的色散大大减少。我们认为,在许多情况下,异常基因的高度分散的原因是,它不服从我们的建模假设;一些基因可能显示更高的变化比其他生物或技术原因,即使他们有相同的平均表达水平。在这些情况下,推理的基础上缩小的分散估计可能导致不良的假阳性。gydF4y2BaDESeq2gydF4y2Ba处理这些情况下利用gene-wise估计而不是萎缩的估计超过2前时剩余标准差曲线之上。gydF4y2Ba
经验贝叶斯收缩叠化评估gydF4y2Ba
高温超导的常见困难分析数据是利物浦的强有力的方差估计基因读计数较低。我们证明这个问题使用底部的数据集gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba]。可视化,如图gydF4y2Ba2gydF4y2Ba,弱表达基因似乎显示更强的老鼠相比菌株之间的差异比强烈表达基因。这种现象出现在最高温超导的数据集,直接处理的结果gydF4y2Ba数gydF4y2Ba数据,比天生吵着当数很低。这个异方差性(利物浦的方差取决于意味着计数)下游分析和数据解释复杂化,因为它使得尺度效应难以比较数据的动态范围。gydF4y2Ba
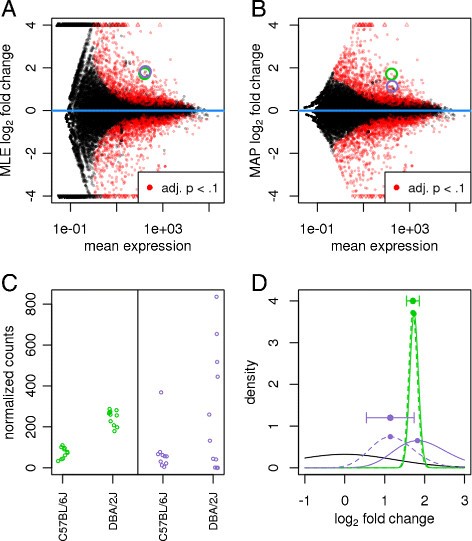
对数褶皱变化收缩估计的影响。gydF4y2Ba情节的gydF4y2Ba(一)gydF4y2Ba大中型企业(即。,no shrinkage) and(B)gydF4y2Ba(即地图估计。,与年代hr我nkage) for the LFCs attributable to mouse strain, over the average expression strength for a ten vs eleven sample comparison of the Bottomlyet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba数据集。小三角形顶部和底部的情节显示点以外的绘图窗口。两个基因具有类似的意思是大中型企业的对数计数和褶皱变化突出显示绿色和紫色的圆圈。gydF4y2Ba(C)gydF4y2Ba数量(按大小归一化的因素gydF4y2Ba年代gydF4y2BajgydF4y2Ba),这些基因揭示低色散的基因基因紫色绿色和高色散。gydF4y2Ba(D)gydF4y2Ba密度的情节可能(实线,按比例缩小的集成1)和后验(虚线)的绿色和紫色基因和前(固体黑线):由于高分散的紫色基因,它可能是更广泛和更少的(少指示信息)达到高峰,背部和前有更多的影响比绿色的基因。曲率越强的绿色后在其最大地图转化为一个较小的标准错误报道利物浦估计(水平误差棒)。调整,调节;利物浦,对数褶皱变化;地图,最大gydF4y2Ba后验gydF4y2Ba;大中型企业,最大似然估计。gydF4y2Ba
DESeq2gydF4y2Ba克服了这一问题,缩小利物浦估计对一分之零的方式,收缩更强时可用的信息基因较低,这可能是因为数量很低,分散高或很少有自由度。我们又雇佣了一个经验贝叶斯过程:首先执行普通GLM适合为利物浦获得最大似然(ml)估计,然后配合zero-centered正态分布毫升的观察到分布在所有的基因。这个分布作为前利物浦在第二轮GLM适合和地图估计作为利物浦的最后估计。此外,每个报告估计的标准误差,这来源于后的曲率最大(有关详细信息,请参阅材料和方法)。这些利物浦萎缩及其标准错误是瓦尔德测试中使用的微分表达式将在下一节中描述。gydF4y2Ba
生成的地图利物浦偏向一分之零方式删除夸张的利物浦低计数的问题。如图gydF4y2Ba2gydF4y2BaB所示,最强的利物浦不再表现出由基因与弱表达。相反,估计更均匀分布在零附近,非常弱表达基因(少于平均每个样本一读),利物浦几乎偏离零,反映出准确的利物浦估计是不可能的。gydF4y2Ba
收缩的力量并不仅仅依靠意味着计数,而是用于褶皱的信息量变化估计(观察费舍尔表示的信息;见材料和方法)。两个基因以同样的表达力,而是不同的分散会经历一个不同数量的收缩(图gydF4y2Ba2gydF4y2BaC, D)。利物浦的收缩估计可以被描述为一个偏见方差的权衡gydF4y2Ba18gydF4y2Ba]:为利物浦估计几乎为零的基因信息,减少强烈的方差购买成本的接受倾向于零,这可能导致一个总体均方误差减少,例如,当比较利物浦估计一个新的数据集。基因为利物浦估计将有高的信息,在我们的方法中,利物浦与低偏差和方差较低。此外,随着自由度的增加,对于利物浦估算和实验提供了更多信息,缩小的估计将收敛于unshrunken估计。我们注意到其他贝叶斯努力缓和RNA-seq褶皱的变化包括层次模型(gydF4y2Ba8gydF4y2Ba]、[gydF4y2Ba19gydF4y2Ba)和gydF4y2BaGFOLDgydF4y2Ba(或广义褶皱变化)工具(gydF4y2Ba20.gydF4y2Ba),它使用一个利物浦的后验分布。gydF4y2Ba
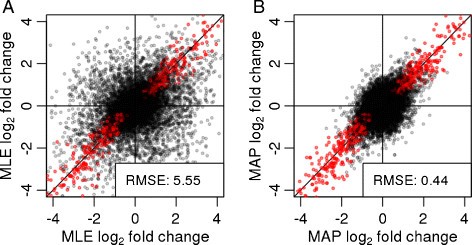
缩小地图利物浦提供了更多可再生的转录大中型企业的利物浦比标准差异的量化。为了证明这一点,我们将底部gydF4y2Baet al。gydF4y2Ba样品同样分成两组,我和二世,这样每组包含一个平衡的菌株的分离,模拟一个场景,一个实验(样品在我组)执行,分析和报告,然后独立复制(样本组II)。在每一组中,我们估计利物浦菌株和第二组之间相比,使用初速利物浦(图gydF4y2Ba3gydF4y2Ba使用地图)和利物浦(图gydF4y2Ba3gydF4y2BaB)。因为收缩移动大利物浦所不支持的数据为零,两个独立样本组之间的协议大大增加。因此,缩小的叠化估计提供了一种更可靠的比正常的ml依据定量的结论。gydF4y2Ba
对数褶皱变化的稳定。gydF4y2BaDESeq2gydF4y2Ba是运行在同样分裂部分的底部的数据吗gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba,半是策划的利物浦。gydF4y2Ba(一)gydF4y2Ba毫升,即。,与out LFC shrinkage.(B)gydF4y2Ba地图的估计,即。,收缩。点左上和右下象限表示基因改变的利物浦的迹象。红点表示基因与调整gydF4y2BaPgydF4y2Ba值< 0.1。传说显示的均方根误差估计在我组在第二组相比。利物浦,对数褶皱变化;地图,最大gydF4y2Ba后验gydF4y2Ba;大中型企业,最大似然估计;均方根误差,均方根误差。gydF4y2Ba
这使得利物浦萎缩也适用于排名的基因,例如,优先为后续实验。例如,如果我们的基因在两个样本图组gydF4y2Ba3gydF4y2Ba由unshrunken利物浦的估计,并考虑最强的100个基因,或下调在我组,我们发现只有21岁的前100年,或衰减基因在第二组。然而,如果我们排名的基因萎缩利物浦估计,重叠提高到81年的100个基因(附加文件gydF4y2Ba1gydF4y2Ba:图S3)。gydF4y2Ba
一个更简单的常用的方法是添加一个固定数量(pseudocount)前各方面形成比率。然而,这只需要一个调优参数的选择和对不确定性的来源之一,低计数,但不要gene-specific分散或样本大小的差异。我们证明这一点gydF4y2Ba基准gydF4y2Ba下面的部分。gydF4y2Ba
假设测试的微分表达式gydF4y2Ba
glm适合每个基因后,你可以测试是否每个模型系数显著不同于零。gydF4y2BaDESeq2gydF4y2Ba报告每个利物浦萎缩的标准误差估计,从曲率系数的获得后(图中虚线gydF4y2Ba2gydF4y2BaD)在其最大值。对于意义的测试,gydF4y2BaDESeq2gydF4y2Ba使用一个瓦尔德测试:利物浦的萎缩估计是除以它的标准误差,导致gydF4y2BazgydF4y2Ba统计,相对于标准正态分布。(有关详细信息,请参阅材料和方法)。瓦尔德测试允许测试个人系数,或对比系数,而不需要适合减少模型与似然比检验,似然比检验虽然也可以作为一个选项gydF4y2BaDESeq2gydF4y2Ba。瓦尔德测试gydF4y2BaPgydF4y2Ba值的子集的基因,通过一个独立的过滤步骤,描述在下一节中,调整为多个测试使用Benjamini和业务的过程gydF4y2Ba21gydF4y2Ba]。gydF4y2Ba
独立自动过滤gydF4y2Ba
由于大量的测试执行RNA-seq和其他全基因组分析的实验中,多个测试问题需要解决。一个受欢迎的目标是控制或罗斯福的估计。多个测试调整往往是联系在一起的力量,在某种意义上,罗斯福的一组基因往往高于个人gydF4y2BaPgydF4y2Ba这些基因的值。然而,可以减少损失如果基因有很少或没有被发现的可能性从测试差异表达都省略了,前提是遗漏的标准是独立的检验统计量在虚假设条件下(gydF4y2Ba22gydF4y2Ba)(见材料与方法)。gydF4y2BaDESeq2gydF4y2Ba使用平均每个基因的表达强度,在所有样本,作为它的过滤准则,它省略了所有基因意味着规范化计数低于过滤阈值从多个测试调整。gydF4y2BaDESeq2gydF4y2Ba默认情况下会选择一个阈值最大化的基因数量发现在用户指定的目标罗斯福。在数据gydF4y2Ba2gydF4y2BaA、B、gydF4y2Ba3gydF4y2Ba,基因发现这样重要的估计为罗斯福10%是红中描述。根据分布的均值归一化计算,由此产生的权力可以大幅增加,有时做的差异是否差异表达基因检测。gydF4y2Ba
假设测试与阈值效应的大小gydF4y2Ba
指定最小的影响大小gydF4y2Ba
大多数方法测试微分表达式,包括默认的方法gydF4y2BaDESeq2gydF4y2Ba、测试的零假设gydF4y2Ba零gydF4y2Ba利物浦。然而,如果任何生物过程真正影响实验处理的差异,这种零假设意味着基因在考虑gydF4y2Ba完美的gydF4y2Ba从这些过程解耦。由于细胞的高关联度的监管网络,这个假设是,事实上,难以置信,并为许多(如果不是大多数基因可以说是错误的。因此,有足够的样本容量,甚至基因与一个非常小但是零利物浦最终会发现差异表达。足够大小的改变应该被考虑gydF4y2Ba生物学上重要gydF4y2Ba。小型实验,统计学意义往往比生物学意义更严格的要求,从而减轻研究者需要确定一个阈值的生物意义。gydF4y2Ba
well-powered实验,然而,传统的零假设的统计检验零利物浦可能报告基因效应强度显著变化如此微弱,他们可以被认为是无关紧要的或分散。常见的过程是不估计利物浦的基因gydF4y2BaβgydF4y2Ba红外gydF4y2Ba低于某个阈值,|gydF4y2BaβgydF4y2Ba红外gydF4y2Ba|≤gydF4y2BaθgydF4y2Ba。然而,这种方法失去的利益更容易解释的罗斯福,报道gydF4y2BaPgydF4y2Ba价值和调整gydF4y2BaPgydF4y2Ba价值仍然对应的考验gydF4y2Ba零gydF4y2Ba利物浦。因此需要包括直接统计测试过程中的阈值,即:没有过滤事后报道,叠化gydF4y2Ba估计gydF4y2Ba,而是直接评估统计是否有足够的证据表明,利物浦在选定的阈值。gydF4y2Ba
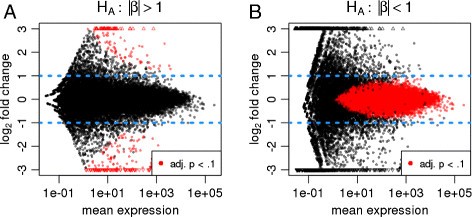
DESeq2gydF4y2Ba为复合材料提供了测试的零假设|gydF4y2BaβgydF4y2Ba红外gydF4y2Ba|≤gydF4y2BaθgydF4y2Ba,在那里gydF4y2BaβgydF4y2Ba红外gydF4y2Ba是缩小的利物浦从上述估计过程。(有关详细信息,请参阅材料和方法)。图gydF4y2Ba4gydF4y2Ba向我们展示了这样一个阈值测试产生弯曲决策边界:达到意义,估计利物浦已经超过指定阈值的数额取决于可用的信息。我们注意到相关方法来生成基因列表,同时满足统计和生物学意义标准之前讨论了微阵列数据gydF4y2Ba23gydF4y2Ba和最近对测序数据gydF4y2Ba19gydF4y2Ba]。gydF4y2Ba
涉及非零阈值的假设检验。gydF4y2Ba显示的情节估计褶皱随平均表达强度变化(“-超过平均水平”,或MA-plots)一百一十年与11比较使用底部gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba]数据集,以强调分表明低调整gydF4y2BaPgydF4y2Ba值。替代假说是对数(基础2)折叠变化gydF4y2Ba(一)gydF4y2Ba在绝对值大于1gydF4y2Ba(B)gydF4y2Ba在绝对值小于1。调整,调整。gydF4y2Ba
指定最大效应大小gydF4y2Ba
研究人员有时是不感兴趣的基因,或者只有非常微弱的,治疗或实验条件的影响。这相当于一个设置类似于刚才讨论,但零的角色和备择假设交换。我们在这里要求的证据作用弱,没有证据的影响为零,因为后者很少是容易处理的问题。的意思gydF4y2Ba弱gydF4y2Ba需要量化生物问题通过选择一个合适的阈值gydF4y2BaθgydF4y2Ba利物浦。这样的分析,gydF4y2BaDESeq2gydF4y2Ba提供一个测试的复合零假设|gydF4y2BaβgydF4y2Ba红外gydF4y2Ba|≥gydF4y2BaθgydF4y2Ba将报告基因显著,有证据表明,他们的利物浦是弱于gydF4y2BaθgydF4y2Ba。图gydF4y2Ba4gydF4y2BaB显示了这样的一个测试的结果。基因与阅读数很低,甚至估计零利物浦并不重要,大不确定性的估计不允许我们排除基因可能实际上比弱受实验条件的影响。注意缺少利物浦收缩:发现基因与疲软的微分表达式,gydF4y2BaDESeq2gydF4y2Ba要求利物浦收缩已被禁用。这是因为zero-centered之前用于利物浦收缩体现了gydF4y2Ba之前gydF4y2Ba相信,利物浦往往比较小,因此是不合适的。gydF4y2Ba
检测统计离群值gydF4y2Ba
参数方法检测微分表达式可以gene-wise估计利物浦过度受到个别异常值的影响,不符合模型的分布假设[gydF4y2Ba24gydF4y2Ba]。离群值的一个例子是一个基因对所有样本单位数项,除了一个样本计数数以千计。微分表达式的目标分析通常是发现gydF4y2Ba始终如一地gydF4y2Ba或抑制基因,它是有用的考虑诊断检测个人过度影响利物浦估计和观察gydF4y2BaPgydF4y2Ba价值的基因。一个标准的异常值诊断是库克的距离(gydF4y2Ba25gydF4y2Ba],它被定义为每个样本在每个基因的比例距离系数向量,gydF4y2Ba的漠视,线性模型或将如果样品被改装和模型。gydF4y2Ba
DESeq2gydF4y2Ba旗帜,对于每个基因,这些样本,库克的距离大于0.99分位数的gydF4y2BaFgydF4y2Ba(gydF4y2BapgydF4y2Ba,gydF4y2Ba米gydF4y2Ba−gydF4y2BapgydF4y2Ba)分布,gydF4y2BapgydF4y2Ba是模型参数的数量包括拦截,然后呢gydF4y2Ba米gydF4y2Ba是样品的数量。的使用gydF4y2BaFgydF4y2Ba分布是出于启发式推理,删除单个样本不应该移动向量gydF4y2Ba外周边地区99%的信心gydF4y2Ba适合使用所有的样品(gydF4y2Ba25gydF4y2Ba]。但是,如果有两个或更少的复制条件,这些不会导致孤立点检测的样品,因为有足够的复制来确定异常状态。gydF4y2Ba
应该如何处理标记异常值吗?与许多复制一个实验,丢弃局外人和继续余下的数据可能充分利用可用的数据。在一个小试验几个样品,然而,一个异类的存在会削弱推理关于受影响的基因,甚至仅仅忽略了离群值可能被视为数据挑选——因此,它更谨慎地排除整个下游基因分析。gydF4y2Ba
因此,gydF4y2BaDESeq2gydF4y2Ba提供了两种可能的反应标记离群值。默认情况下,异常值与六个或更少的条件复制导致整个基因标记,从后来的分析,包括gydF4y2BaPgydF4y2Ba值调整为多个测试。对于包含七个或多个复制的条件,gydF4y2BaDESeq2gydF4y2Ba取代了离群值计算一个估算值,即削减意味着对所有样品,大小比例的因素,然后重新估算色散,利物浦gydF4y2BaPgydF4y2Ba这些基因的值。作为离群值替换值预测的零假设的微分表达式,这是更为保守的选择不仅仅是省略的局外人。当有许多自由度,第二种方法避免丢弃基因可能包含真正的微分表达式。gydF4y2Ba
额外的文件gydF4y2Ba1gydF4y2Ba:图S4显示单个基因的异常值替代过程的七个七比较底gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba数据集。原来的安装方式深受一个样本有一个很大的数,修正后的利物浦提供更好的适合大多数的样本。gydF4y2Ba
正规化的对数转换gydF4y2Ba
对于特定的分析,是有用的转换数据呈现办到的。作为一个例子,考虑评估样本相似的任务在一个无监督的方式使用聚类或分类算法。对于RNA-seq数据,异方差性的问题出现了:如果数据给出这种算法在原始的计算规模,结果将是由高度表达,高度可变的基因;如果logarithm-transformed数据使用,过度的体重将弱表达基因,这表明夸张的利物浦,正如上面所讨论的。因此,我们使用收缩的方法gydF4y2BaDESeq2gydF4y2Ba实现一个gydF4y2Ba正规化的对数gydF4y2Ba转换(rlog)行为类似于log2转换基因高计数,而收缩在一起不同的基因样本的值较低。因此避免了通常的观察到的属性标准对数变换,数据的扩散分离基因数较低,在随机噪声是任何生物意义的信号可能会主导。当我们考虑每个基因的方差,计算样本,这些差异是稳定——即。,一个pproximately the same, or homoskedastic – after the rlog transformation, while they would otherwise strongly depend on the mean counts. It thus facilitates multivariate visualization and ordinations such as clustering or principal component analysis that tend to work best when the variables have similar dynamic range. Note that while the rlog transformation builds upon on our LFC shrinkage approach, it is distinct from and not part of the statistical inference procedure for differential expression analysis described above, which employs the raw counts, not transformed data.
rlog转换计算通过为每个基因的漠视与基线拟合表达式(即。,我ntercept only) and, computing for each sample, shrunken LFCs with respect to the baseline, using the same empirical Bayes procedure as before (Materials and methods). Here, however, the sample covariate information (e.g. treatment or control) is not used, so that all samples are treated equally. The rlog transformation accounts for variation in sequencing depth across samples as it represents the logarithm of问gydF4y2BaijgydF4y2Ba在考虑了大小的因素gydF4y2Ba年代gydF4y2BaijgydF4y2Ba。这与variance-stabilizing转换(VST) overdispersed计数了gydF4y2BaDESeqgydF4y2Ba(gydF4y2Ba4gydF4y2Ba]:威仕特也有效地稳定方差,它不直接考虑的差异大小的因素;在数据集大型测序深度的变化(动态范围的大小的因素gydF4y2Ba4)我们观察到不良工件在威仕特的性能。rlog变换的缺点对威仕特是,然而,样本中基因的排序将会改变,如果邻近基因进行不同强度的收缩。与威仕特rlog的价值(gydF4y2BaKgydF4y2BaijgydF4y2Ba)对于大数量约等于log2 (gydF4y2BaKgydF4y2BaijgydF4y2Ba/gydF4y2Ba年代gydF4y2BajgydF4y2Ba)。rlog转换和威仕特提供的gydF4y2BaDESeq2gydF4y2Ba包中。gydF4y2Ba
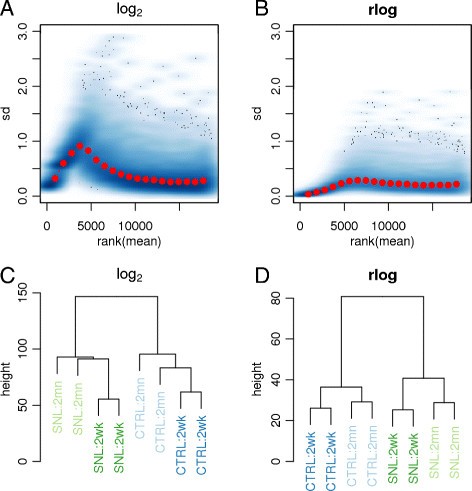
我们将演示使用rlog RNA-seq数据集的转换gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba26gydF4y2Ba),在背根神经节的核糖核酸测序老鼠经历了脊神经结扎和控制,在2周和结扎后2个月。这个数据集的计算矩阵从叙述在线下载资源(gydF4y2Ba27gydF4y2Ba]。这个数据集提供了更微妙的差异比底部的条件gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba数据集。图gydF4y2Ba5gydF4y2Ba提供诊断块普通下的归一化计算对数的pseudocount 1和rlog转换,显示稳定的rlog都通过一系列数的均值和方差有助于数据中找到有意义的模式。gydF4y2Ba
方差稳定化和集群后rlog转换。gydF4y2Ba两个转换应用于锤的计数gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba26gydF4y2Ba数据集:规范化的对数项加上pseudocount,即。gydF4y2BafgydF4y2Ba(gydF4y2BaKgydF4y2BaijgydF4y2Ba)= log2 (gydF4y2BaKgydF4y2BaijgydF4y2Ba/gydF4y2Ba年代gydF4y2BajgydF4y2Ba+ 1),rlog。转换值变量的标准差gene-wise整个范围的意思是使用对数项gydF4y2Ba(一)gydF4y2Ba,而使用rlog相对稳定gydF4y2Ba(B)gydF4y2Ba。层次聚类在使用rlog欧几里得距离和完整的链接gydF4y2Ba(D)gydF4y2Ba转换数据集群的样品到定义的组治疗和时间,而使用logarithm-transformed计数gydF4y2Ba(C)gydF4y2Ba产生一个更模棱两可的结果。sd,标准差。gydF4y2Ba
能够分析gydF4y2Ba
我们这里现在gydF4y2BaDESeq2gydF4y2Ba每个基因的分析,即。,total number of reads that can be uniquely assigned to a gene. In contrast, several algorithms [28gydF4y2Ba]、[gydF4y2Ba29日gydF4y2Ba]使用概率分配读取记录,在多个重叠的成绩单可以源自每个基因。已经指出,阅读总数的方法会导致错误检测的微分表达式实际上只有成绩单同种型长度改变时,甚至在一个错误的利物浦的迹象在极端情况下(gydF4y2Ba28gydF4y2Ba]。然而,在我们的基准,在下一节中所讨论的,我们发现,利物浦签下读总数之间的分歧和probabilistic-assignment-based方法是罕见的基因差异表达方法(附加文件gydF4y2Ba1gydF4y2Ba:图S5)。此外,如果估计平均记录长度可供条件,这些可以被纳入gydF4y2BaDESeq2gydF4y2Ba框架作为基因,sample-specific规范化的因素。此外,使用的方法gydF4y2BaDESeq2gydF4y2Ba可以扩展到isoform-specific分析,通过广义线性模型的外显子层次gene-specific在意味着什么gydF4y2BaDEXSeqgydF4y2Ba包(gydF4y2Ba30.gydF4y2Ba)或通过计算证据替代亚型在拼接图gydF4y2Ba31日gydF4y2Ba]、[gydF4y2Ba32gydF4y2Ba]。事实上,最新的发布版本gydF4y2BaDEXSeqgydF4y2Ba现在使用gydF4y2BaDESeq2gydF4y2Ba作为其推论引擎提供收缩估计的色散和尺度效应的exon-level分析,。gydF4y2Ba
比较基准gydF4y2Ba
评估如何gydF4y2BaDESeq2gydF4y2Ba执行标准分析其他现有方法相比,我们使用模拟和实际数据的组合。negative-binomial-based方法相比gydF4y2BaDESeq(旧)gydF4y2Ba(gydF4y2Ba4gydF4y2Ba),gydF4y2Ba刨边机gydF4y2Ba(gydF4y2Ba33gydF4y2Ba),gydF4y2Ba刨边机gydF4y2Ba健壮的选项(gydF4y2Ba34gydF4y2Ba),gydF4y2BaDSSgydF4y2Ba(gydF4y2Ba6gydF4y2Ba),gydF4y2BaEBSeqgydF4y2Ba(gydF4y2Ba35gydF4y2Ba]。是相比其他方法gydF4y2Ba轰gydF4y2Ba归一化法其次是线性建模使用gydF4y2BalimmagydF4y2Ba包(gydF4y2Ba36gydF4y2Ba)和gydF4y2BaSAMseqgydF4y2Ba的排列方法gydF4y2BasamrgydF4y2Ba包(gydF4y2Ba24gydF4y2Ba]。使用真实的数据为基准,gydF4y2BaCuffdiff 2gydF4y2Ba(gydF4y2Ba28gydF4y2Ba袖扣套件的)方法是包括在内。使用的软件的版本号,请参阅附加的文件gydF4y2Ba1gydF4y2Ba:表S3。对所有算法返回gydF4y2BaPgydF4y2Ba值,gydF4y2BaPgydF4y2Ba值从基因与非零和的阅读数量样本调整使用Benjamini-Hochberg过程[gydF4y2Ba21gydF4y2Ba]。gydF4y2Ba
通过模拟基准gydF4y2Ba
灵敏度和精度gydF4y2Ba我们模拟数据集10000个基因的负二项分布。模拟数据与现实的时刻,意味着和分散的联合分布的手段和gene-wise Pickrell分散估计gydF4y2Baet al。gydF4y2Ba数据,拟合只有截距项。这些数据集是不同的总样本量(gydF4y2Ba米gydF4y2Ba∈gydF4y2Ba{6、8、10、20}),样品被分成两个大小相等的组;80%的模拟基因没有真正的微分表达式,而对于基因的20%,真正的褶皱的变化2、3和4是用于生成计数两组,与褶皱的方向变化随机抽取的。随机模拟差异表达基因选择统一在所有的基因,在整个范围的平均数量。MA-plots真正的褶皱变化用于模拟和观察到的褶皱变化引起仿真的仿真设置额外的文件所示gydF4y2Ba1gydF4y2Ba:图S6。gydF4y2Ba
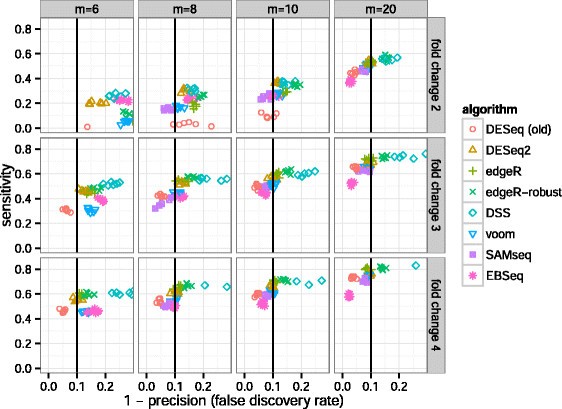
算法的性能仿真指标评估的灵敏度和精度。灵敏度计算的一部分基因与调整gydF4y2BaPgydF4y2Ba值< 0.1与真实之间的差异的基因群。计算精度与真正的差异基因的一部分组织意味着那些调整gydF4y2BaPgydF4y2Ba值< 0.1。策划/(1−精度灵敏度,或罗斯福,在图gydF4y2Ba6gydF4y2Ba。gydF4y2BaDESeq2gydF4y2Ba,也gydF4y2Ba刨边机gydF4y2Ba,通常最高灵敏度的算法控制的i型错误,实际的罗斯福在或低于0.1,调整阈值gydF4y2BaPgydF4y2Ba值用于调用差异表达基因。gydF4y2BaDESeq2gydF4y2Ba有更高的灵敏度比其他算法,特别是对小褶皱变化(2或3),还发现在基准由周gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba34gydF4y2Ba]。对于更大的样本量和较大的褶皱变化各种算法的性能是一致的。gydF4y2Ba
灵敏度和精度的算法在样本大小和影响大小的组合。gydF4y2BaDESeq2gydF4y2Ba和gydF4y2Ba刨边机gydF4y2Ba通常最高灵敏度控制了罗斯福的这些算法,即。这些算法,落在左边的竖线。情节的敏感性对假阳性率,而不是罗斯福,看到额外的文件gydF4y2Ba1gydF4y2Ba:图S8和依赖的敏感性的意思,看到额外的文件gydF4y2Ba1gydF4y2Ba:图S9。请注意,gydF4y2BaEBSeqgydF4y2Ba过滤器低、基因(见正文)。gydF4y2Ba
过于保守的旧的gydF4y2BaDESeqgydF4y2Ba工具可以观察到,降低灵敏度相比其他算法和一个实际的罗斯福小于标称值为0.1。我们注意到,gydF4y2BaEBSeqgydF4y2Ba默认版本1.4.0删除低、基因的75%分位数的规范化计数小于前十——调用微分表达式。算法在模拟数据的敏感性,在一系列重要的均值相比更密切的额外文件gydF4y2Ba1gydF4y2Ba:图S9。gydF4y2Ba
离群值的敏感性gydF4y2Ba我们使用模拟的敏感性和特异性进行比较gydF4y2BaDESeq2gydF4y2Ba的异常处理的方法gydF4y2Ba刨边机gydF4y2Ba最近添加到软件,虽然这手稿出版受到审查。gydF4y2Ba刨边机gydF4y2Ba现在包括一个可选的方法来处理异常值的迭代改装后的漠视,重量降低潜在的离群值计数(gydF4y2Ba34gydF4y2Ba]。模拟,总结在附加的文件gydF4y2Ba1gydF4y2Ba:图S10,表明这两种方法异常值几乎恢复outlier-free数据集上的性能,gydF4y2BaedgeR-robustgydF4y2Ba实际已略高于名义罗斯福,见附加文件gydF4y2Ba1gydF4y2Ba:图S11。gydF4y2Ba
褶皱变化的精确估计gydF4y2Ba我们的基准测试gydF4y2BaDESeq2gydF4y2Ba用实证的方法实现收缩之前利物浦估计对两个竞争方法:gydF4y2BaGFOLDgydF4y2Ba方法,该方法可以分析实验没有复制(gydF4y2Ba20.gydF4y2Ba),也可以处理的复制实验,gydF4y2Ba刨边机gydF4y2Ba包,它提供了一种pseudocount-based收缩称为gydF4y2Ba预测利物浦gydF4y2Ba。结果总结在附加的文件gydF4y2Ba1gydF4y2Ba:数字S12-S16。gydF4y2BaDESeq2gydF4y2Ba一直低的均方根误差和平均绝对误差在一系列样本大小和分布的模型真正的利物浦。gydF4y2BaGFOLDgydF4y2Ba有错误吗gydF4y2BaDESeq2gydF4y2Ba所有的基因;然而,当关注差异表达基因,它表现糟糕,更大的样本量。gydF4y2Ba刨边机gydF4y2Ba用默认设置有错误gydF4y2BaDESeq2gydF4y2Ba当只关注差异表达基因,但有更高的误差对所有基因。gydF4y2Ba
聚类gydF4y2Ba我们比较rlog转换的性能与其他方法的转换或复苏的距离计算模拟集群。调整兰特指数(gydF4y2Ba37gydF4y2Ba)是用来比较层次聚类根据不同的距离,真正的集群成员。我们测试的欧几里得距离标准化,规范化的对数计数+ 1 pseudocount, rlog-transformed计数和威仕特。此外我们比较这些欧几里得距离的泊松距离实现gydF4y2BaPoiClaClugydF4y2Ba包(gydF4y2Ba38gydF4y2Ba),而远处的内部实现gydF4y2BaplotMDSgydF4y2Ba的函数gydF4y2Ba刨边机gydF4y2Ba(虽然不是默认的距离,这是类似于规范化的对数项)。所示的结果,附加的文件gydF4y2Ba1gydF4y2Ba:图肌力,透露,当所有样本的大小的因素都是平等的,泊松距离和欧几里得距离rlog-transformed或威仕特表现优于其他方法。然而,当不等于在样本大小因素,通常rlog方法优于其他方法。最后,我们注意到,rlog转换提供了规范化的数据,可以用于各种各样的应用程序,其中距离计算就是其中之一。gydF4y2Ba
核糖核酸测序数据的基准gydF4y2Ba
虽然仿真是有用的验证算法的行为与理想化的理论数据,因此可以验证该算法执行如预期自己的假设下,模拟无法告知我们如何理论符合现实。RNA-seq数据,是完全不知道的并发症或直接底层的真理;不过,我们可以通过使用更多的间接推断绕过这个限制,下面的解释。gydF4y2Ba
在接下来的基准,我们考虑三个性能指标微分表达式调用:假阳性率(1 -特异性),灵敏度和精度。我们可以从观察获得特异性的有意义的估计数据集,我们相信所有基因属于零假设的微分表达式(gydF4y2Ba39gydF4y2Ba]。灵敏度和精度估计更困难,因为他们需要独立的知识差异表达的基因。为了规避这一问题,我们使用实验重现性对独立样本(尽管从相同的数据集)作为代理。我们使用一个数据集和大量复制两组,我们希望真正存在的差异表达基因。我们重复这个数据集分割成一个评价集和一个更大的验证集,从评价集和比较了调用的调用验证集,被视为真理。重要的是要记住,电话验证组只是一个近似的微分状态,真正的和近似误差有一个系统和一个随机组件。随机误差变小了,小的样本容量验证设置足够大。系统错误,我们的基准假设这些或多或少影响所有算法同样不显著改变的排名算法。gydF4y2Ba
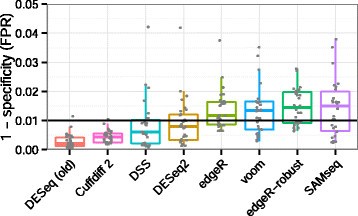
假阳性率gydF4y2Ba评估的假阳性率算法,我们认为模拟比较从一个数据集和许多样品和没有已知条件将样本划分为不同的群体。我们使用了RNA-seq Pickrell的数据gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba17gydF4y2Ba]lymphoblastoid细胞系来自尼日利亚人无关。我们选择一组26 RNA-seq样本长度相同的阅读(46个碱基对)雄性个体。我们不重复随机抽出十集样本比较五对五,这个过程被重复30次。我们估计的假阳性率与一个关键价值0.01的数量除以gydF4y2BaPgydF4y2Ba总数值小于0.01的测试;基因与零和的阅读数量在样品被排除在外。结果30复制,总结在图gydF4y2Ba7gydF4y2Ba指出,所有算法通常控制假阳性的数量。gydF4y2BaDESeq(旧)gydF4y2Ba和gydF4y2BaCuffdiff 2gydF4y2Ba在这个分析中出现过于保守,不使用他们的i型误差预算。gydF4y2Ba
基准的假阳性。gydF4y2Ba显示估计的gydF4y2BaPgydF4y2Ba(gydF4y2BaPgydF4y2Ba零假设下值< 0.01)。玻璃钢的数量gydF4y2BaPgydF4y2Ba值小于0.01除以总数量的测试,从随机选择从Pickrell五对五的比较样本gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba17gydF4y2Ba]数据集,没有已知的条件划分样本。i型错误控制要求工具不大大超过标称值0.01(黑线)。gydF4y2BaEBSeqgydF4y2Ba结果并不包括在这一情节,因为它返回后验概率,不像gydF4y2BaPgydF4y2Ba值不均匀分布在零假设。玻璃钢,假阳性。gydF4y2Ba
灵敏度gydF4y2Ba获得的印象的灵敏度算法,我们考虑下gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba]数据集,其中包含10和11两个不同的复制,基因同质的小鼠品系。这使得分割的三对三对评价集和7和8验证集,是平衡整个三个实验批次。随机分裂复制的30倍。批处理没有提供信息gydF4y2BaDESeq(旧)gydF4y2Ba,gydF4y2BaDESeq2gydF4y2Ba,gydF4y2BaDSSgydF4y2Ba,gydF4y2Ba刨边机gydF4y2Ba或gydF4y2Ba轰gydF4y2Ba算法可以适应复杂的实验设计,可比调用所有算法。gydF4y2Ba
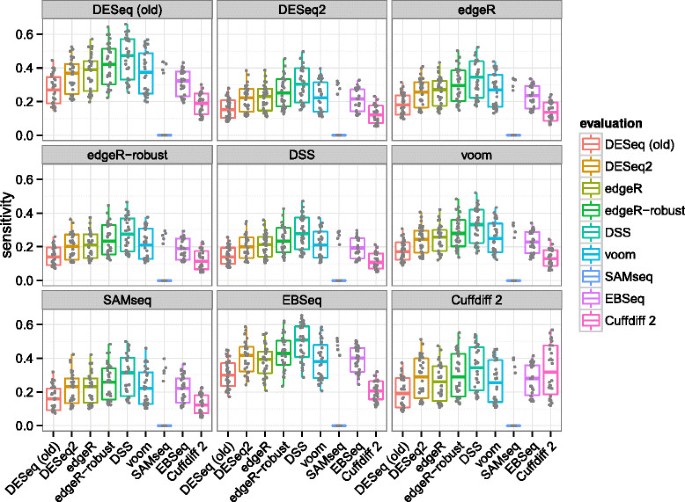
我们虽然每个旋转算法来确定调用验证集的。对于一个给定的算法的验证集电话,我们测试了每个算法的评价集调用。我们使用这种方法,而不是一个一致同意的方法,当我们不想赞成或不赞成任何特定的算法或算法。灵敏度计算的模拟基准,现在真正的微分表达式定义的一个调整gydF4y2BaPgydF4y2Ba大验证设置值< 0.1,额外的说明文件gydF4y2Ba1gydF4y2Ba:S18图美国。图gydF4y2Ba8gydF4y2Ba显示估计每个算法的灵敏度。gydF4y2Ba
的排名算法普遍一致的无论选择哪种算法来确定调用在验证设置。gydF4y2BaDESeq2gydF4y2Ba都比较敏感gydF4y2Ba刨边机gydF4y2Ba和gydF4y2Ba轰gydF4y2Ba虽然不到gydF4y2BaDSSgydF4y2Ba。中值灵敏度估计通常是在0.2和0.4之间为所有算法。所有的算法都相对较低的平均灵敏度可以解释小样本大小的评价集和验证的事实,增加样本量增加力量。这是预期,permutation-basedgydF4y2BaSAMseqgydF4y2Ba方法将很少生产调整gydF4y2BaPgydF4y2Ba在评价集值< 0.1,因为三个和三个比较没有使足够的排列。gydF4y2Ba
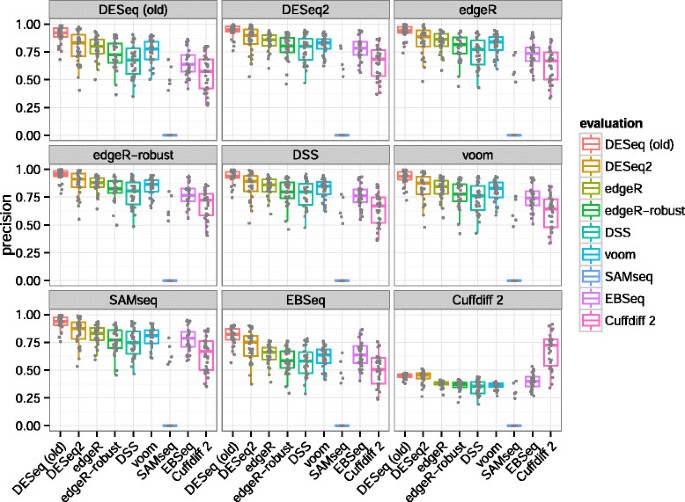
精度gydF4y2Ba另一个重要的考虑从一名调查员的角度精确,或一部分真正的阳性组基因通过调整gydF4y2BaPgydF4y2Ba阈值。这也可以报告为1−罗斯福。再次,“真正的”微分表达式定义的调整gydF4y2BaPgydF4y2Ba值< 0.1大验证集。估计精度是显示在图gydF4y2Ba9gydF4y2Ba,我们可以看到gydF4y2BaDESeq2gydF4y2Ba经常有第二高值的精度,在后面gydF4y2BaDESeq(旧)gydF4y2Ba。我们也可以看到值灵敏度较高的算法,例如:gydF4y2BaDSSgydF4y2Ba,通常是中等精度较低。排名有明显差别gydF4y2BaCuffdiff 2gydF4y2Ba被用来确定验证设置调用。这可能是由于额外的步骤gydF4y2BaCuffdiff 2gydF4y2Ba执行从能够丰富deconvolve isoform-level丰度的变化,这显然是在较低的成本对自己验证调用集精密相比。gydF4y2Ba
进一步比较灵敏度和精度结果,我们计算算法的精度以及网格名义上的调整gydF4y2BaPgydF4y2Ba值(额外的文件gydF4y2Ba1gydF4y2Ba:图S19)。然后我们发现了名义上的调整gydF4y2BaPgydF4y2Ba值为每个算法,导致平均实际精度0.9(罗斯福= 0.1)。因此校准每个算法目标罗斯福,我们评估的灵敏度要求,如额外的文件所示gydF4y2Ba1gydF4y2Ba:图S20。正如预期的那样,这里的算法执行更多的彼此相似。这一分析表明,对于一个给定的目标精度,gydF4y2BaDESeq2gydF4y2Ba通常是由平均灵敏度算法,虽然变化在随机复制比算法之间的差异更大。gydF4y2Ba
绝对数量的要求评估和验证设置中可以看到额外的文件gydF4y2Ba1gydF4y2Ba:数据S21 S22,大多匹配顺序的敏感性阴谋图gydF4y2Ba8gydF4y2Ba。额外的文件gydF4y2Ba1gydF4y2Ba:图S23和S24提供热量地图和集群基于Jaccard呼吁的一个复制指数评价和验证集,表明大量重叠调用不同的算法。gydF4y2Ba
总之,基准测试显示gydF4y2BaDESeq2gydF4y2Ba有效控制i型错误,保持平均误判率略低于临界值的模拟比较选择组样本随机选择从一个更大的游泳池。仿真和实际数据的分析,gydF4y2BaDESeq2gydF4y2Ba经常达到最高灵敏度的算法控制了罗斯福。gydF4y2Ba
结论gydF4y2Ba
DESeq2gydF4y2Ba提供了一个全面的和通用的解决方案能够RNA-seq数据的分析。收缩估计大大提高分析结果的稳定性和重现性相对于maximum-likelihood-based解决方案。经验贝叶斯先验提供自动控制的收缩量的基于信息的数量估计量中可用的数据。这允许gydF4y2BaDESeq2gydF4y2Ba提供一致的性能在一个大范围的数据类型和使它适用于小型研究很少有复制以及大型观察性研究。gydF4y2BaDESeq2gydF4y2Ba为离群值的启发式检测有助于识别基因的建模假设是不合适的,所以避免了i型错误造成的。glm的嵌入这些策略的框架支持的治疗既简单又复杂的设计。gydF4y2Ba
关键进步是褶皱的收缩估计量变化微分表达式的分析,提供一个良好的和统计的相关解决实际问题比较褶皱变化RNA-seq实验的宽动态范围。这是有价值的对于许多下游分析任务,包括后续研究基因的排名和协会的褶皱变化与其他感兴趣的变量。此外,rlog转换,实现收缩折叠的变化在每个样本的基础上,促进可视化差异,例如在热量地图,并支持的应用广泛的技术,需要办到的输入数据,包括机器学习或配合技术,如主成分分析和聚类。gydF4y2Ba
DESeq2gydF4y2Ba因此从业者提供一组广泛的功能和先进的推理能力。其用例并不仅限于RNA-seq数据或其他转录组分析;相反,可以使用多种高通量计算数据。其他地区的gydF4y2BaDESeqgydF4y2Ba或gydF4y2BaDESeq2gydF4y2Ba已经使用包括染色质免疫沉淀反应测序分析(例如,gydF4y2Ba40gydF4y2Ba];看到也gydF4y2BaDiffBindgydF4y2Ba包(gydF4y2Ba41gydF4y2Ba]、[gydF4y2Ba42gydF4y2Ba]),barcode-based化验(例如,gydF4y2Ba43gydF4y2Ba(例如,[]),宏基因组数据gydF4y2Ba44gydF4y2Ba]),核糖体分析(gydF4y2Ba45gydF4y2Ba)和CRISPR / Cas-library化验(gydF4y2Ba46gydF4y2Ba]。最后,gydF4y2BaDESeq2gydF4y2Ba包是集成在Bioconductor基础设施(gydF4y2Ba11gydF4y2Ba),提供了完善的文档,包括装饰图案,展示了一个完整的逐步分析和讨论了先进的用例。gydF4y2Ba
材料和方法gydF4y2Ba
摘要中使用的符号下面一节中提供了额外的文件gydF4y2Ba1gydF4y2BaS1:表。gydF4y2Ba
模型和标准化gydF4y2Ba
读计数gydF4y2BaKgydF4y2BaijgydF4y2Ba基因gydF4y2Ba我gydF4y2Ba在示例gydF4y2BajgydF4y2Ba描述的GLM负二项家庭与对数链接:gydF4y2Ba
符号简单的方程使用自然对数函数的联系,虽然gydF4y2BaDESeq2gydF4y2Ba软件报告估计模型系数及其log2规模估计标准误差。gydF4y2Ba
默认情况下,归一化常数gydF4y2Ba年代gydF4y2BaijgydF4y2Ba在样品被认为是常数,gydF4y2Ba年代gydF4y2BaijgydF4y2Ba=gydF4y2Ba年代gydF4y2BajgydF4y2Ba,估计median-of-ratios先前描述和使用方法gydF4y2BaDESeqgydF4y2Ba(gydF4y2Ba4gydF4y2Ba),gydF4y2BaDEXSeqgydF4y2Ba(gydF4y2Ba30.gydF4y2Ba]:gydF4y2Ba
另外,用户可以供应归一化常数gydF4y2Ba年代gydF4y2BaijgydF4y2Ba使用其他方法(例如,使用计算gydF4y2BacqngydF4y2Ba(gydF4y2Ba13gydF4y2Ba]或gydF4y2BaEDASeqgydF4y2Ba(gydF4y2Ba14gydF4y2Ba),可能不同于基因的基因。gydF4y2Ba
扩展设计矩阵gydF4y2Ba
与我们的软件的文档的一致性,在以下文本我们将使用的术语gydF4y2BaRgydF4y2Ba统计语言。在线性建模,或分类变量gydF4y2Ba因素gydF4y2Ba可以在两个或两个以上的值或gydF4y2Ba水平gydF4y2Ba。在标准的设计矩阵,其中一个值作为参考价值或选择gydF4y2Ba基础水平gydF4y2Ba和吸收拦截。在标准的漠视,基础水平的选择不影响值的对比(利物浦)。然而,这不再是在我们的方法使用ridge-regression-like收缩系数(在下面描述),当有超过两个层次因素出现在设计矩阵,因为基础水平将不会接受收缩,而其他的水平。gydF4y2Ba
恢复所有级别之间的理想的对称,gydF4y2BaDESeq2gydF4y2Ba使用gydF4y2Ba扩展设计矩阵gydF4y2Ba,其中包括一个指标变量gydF4y2Ba每一个gydF4y2Ba每个因素的水平,除了一个拦截列(即。水平,没有一个是拦截吸收)。虽然不再这样的设计矩阵满秩,存在唯一解,因为zero-centered先验分布(见下文)提供正规化。分散估计和估计的宽度利物浦之前,使用标准设计矩阵。gydF4y2Ba
对比gydF4y2Ba
水平和标准误差之间的对比等对比可以计算他们在标准设计矩阵的情况下,即使用:gydF4y2Ba
在哪里gydF4y2Ba代表一个数字的对比,例如,1和−1指定一个简单的分子和分母两级之下,和gydF4y2Ba下面的定义。gydF4y2Ba
分散估计gydF4y2Ba
我们假设弥散参数gydF4y2BaαgydF4y2Ba我gydF4y2Ba遵循对数正态分布先验分布,围绕这一趋势取决于基因的意思是规范化阅读数:gydF4y2Ba
在这里,gydF4y2BaαgydF4y2BatrgydF4y2Ba基因是一个函数的均值归一化计算,gydF4y2Ba
它描述了mean-dependent之前的预期。gydF4y2BaσgydF4y2BadgydF4y2Ba之前的宽度,hyperparameter描述单个基因的多少真正的分散体系分散的趋势。对于趋势函数,我们使用相同的参数化gydF4y2BaDEXSeqgydF4y2Ba(gydF4y2Ba30.gydF4y2Ba),即gydF4y2Ba
我们得到最终分散估计该模型在三个步骤中,实现快速计算近似一个完整经验贝叶斯治疗。我们第一次使用统计数据为每个基因分别得到初步gene-wise分散估计gydF4y2Ba最大似然估计。然后,我们符合分散的趋势gydF4y2BaαgydF4y2BatrgydF4y2Ba。最后,我们结合之前趋势的可能性最大gydF4y2Ba后验gydF4y2Ba(MAP)值作为最终的分散估计。三个步骤的细节。gydF4y2Ba
Gene-wise分散估计gydF4y2Ba得到一个gene-wise分散估计基因gydF4y2Ba我gydF4y2Ba的漠视,我们首先安装一个负二项没有利物浦之前设计矩阵gydF4y2BaXgydF4y2Ba基因的统计数据。全球语言监测机构使用一个粗略的矩量法估计的分散,基于类内方差和手段。最初的全球语言监测机构必须获得一组初始的拟合值,gydF4y2Ba。然后我们最大化Cox-Reid调整分散的可能性,在安装条件值gydF4y2Ba从最初的健康,获得gene-wise估计gydF4y2Ba,也就是说,gydF4y2Ba
与gydF4y2Ba
在哪里gydF4y2BafgydF4y2Ba注gydF4y2Ba(gydF4y2BakgydF4y2Ba;gydF4y2BaμgydF4y2Ba,gydF4y2BaαgydF4y2Ba)是负二项分布的概率质量函数的意思gydF4y2BaμgydF4y2Ba和色散gydF4y2BaαgydF4y2Ba,第二项提供Cox-Reid偏差调整(gydF4y2Ba47gydF4y2Ba]。这种调整,第一次使用的分散估计(SAGE数据的gydF4y2Ba48gydF4y2Ba然后对高温超导数据(gydF4y2Ba3gydF4y2Ba在gydF4y2Ba刨边机gydF4y2Ba,纠正负偏压的分散使用的ml估计拟合值gydF4y2Ba(类似于通常的样本方差贝塞尔的修正公式;,(gydF4y2Ba49gydF4y2Ba),10.6节)。这是费舍尔信息形成的拟合值,就是在这里计算的依据(gydF4y2BaXgydF4y2BatgydF4y2BaWgydF4y2BaXgydF4y2Ba),gydF4y2BaWgydF4y2Ba从标准迭代矩阵对角重量是再加权最小二乘算法。GLM的链接功能gydF4y2BaggydF4y2Ba(gydF4y2BaμgydF4y2Ba)=日志(gydF4y2BaμgydF4y2Ba)及其方差函数gydF4y2BaVgydF4y2Ba(gydF4y2BaμgydF4y2Ba;gydF4y2BaαgydF4y2Ba)=gydF4y2BaμgydF4y2Ba+gydF4y2BaαgydF4y2BaμgydF4y2Ba2gydF4y2Ba对角矩阵的元素gydF4y2BaWgydF4y2Ba我gydF4y2Ba是由:gydF4y2Ba
方程的优化(gydF4y2Ba7gydF4y2Ba)执行的日志gydF4y2BaαgydF4y2Ba使用回溯线搜索和接受建议,满足Armijo条件(gydF4y2Ba50gydF4y2Ba]。gydF4y2Ba
分散的趋势gydF4y2Ba参数曲线的形式(gydF4y2Ba6gydF4y2Ba)是适合通过回归gene-wise分散估计gydF4y2Ba在规范化的重要手段,gydF4y2Ba。的抽样分布gene-wise分散估计的真正价值gydF4y2BaαgydF4y2Ba我gydF4y2Ba可以高度倾斜,因此我们不要使用普通最小二乘回归的漠视,而是gamma-family回归。此外,分散异常值可以倾斜,因此计划排除使用这样的异常值。gydF4y2Ba
的hyperparametersgydF4y2Ba一个gydF4y2Ba1gydF4y2Ba和gydF4y2BaαgydF4y2Ba0gydF4y2Ba(gydF4y2Ba6gydF4y2Bagamma-family GLM)是通过迭代拟合。在每个迭代中,基因的比例分散范围外的拟合值(10gydF4y2Ba−4gydF4y2Ba,15]直到利物浦新系数的平方和的旧系数小于10gydF4y2Ba−6gydF4y2Ba(同样的方法gydF4y2BaDEXSeqgydF4y2Ba(gydF4y2Ba30.gydF4y2Ba])。gydF4y2Ba
参数化(gydF4y2Ba6gydF4y2Ba)是基于报告由美国和其他减少色散的依赖意味着在许多数据集(gydF4y2Ba3gydF4y2Ba]- [gydF4y2Ba6gydF4y2Ba]、[gydF4y2Ba51gydF4y2Ba]。有理由保持一份谨慎解开真正潜在的依赖影响的估计偏差,可以创建一个感知依赖分散的意思。考虑一个负二项分布的随机变量的期望gydF4y2BaμgydF4y2Ba和色散gydF4y2BaαgydF4y2Ba。它的方差gydF4y2BavgydF4y2Ba=gydF4y2BaμgydF4y2Ba+gydF4y2BaαgydF4y2BaμgydF4y2Ba2gydF4y2Ba有两个组件,gydF4y2BavgydF4y2Ba=gydF4y2BavgydF4y2BaPgydF4y2Ba+gydF4y2BavgydF4y2BaDgydF4y2Ba泊松组件gydF4y2BavgydF4y2BaPgydF4y2Ba=gydF4y2BaμgydF4y2Ba独立的gydF4y2BaαgydF4y2Ba,overdispersion组件gydF4y2BavgydF4y2BaDgydF4y2Ba=gydF4y2BaαgydF4y2BaμgydF4y2Ba2gydF4y2Ba。当gydF4y2BaμgydF4y2Ba很小,gydF4y2BaμgydF4y2Ba≲gydF4y2Ba1 /gydF4y2BaαgydF4y2Ba(竖线的额外文件gydF4y2Ba1gydF4y2Ba:图S1),泊松分量占主导地位,在这个意义上gydF4y2Ba和观察到的数据没有提供信息的价值gydF4y2BaαgydF4y2Ba。因此抽样方差的估计量gydF4y2BaαgydF4y2Ba时将大gydF4y2BaμgydF4y2Ba≲gydF4y2Ba1 /gydF4y2BaαgydF4y2Ba,从而导致偏差的出现。为简单起见,我们声明以上论点不考虑大小的影响因素,gydF4y2Ba年代gydF4y2BajgydF4y2Ba的价值gydF4y2BaμgydF4y2Ba。从结构来看,这是允许的,因为几何平均数的大小因素是接近1,因此,跨样本均值的非规范读计数,gydF4y2Ba规范化阅读数的均值,gydF4y2Ba将大致相同。gydF4y2Ba
这种现象可能会产生一个明显的依赖关系gydF4y2BaαgydF4y2Ba在gydF4y2BaμgydF4y2Ba。可能的形状dispersion-mean适合底层数据(图gydF4y2Ba1gydF4y2Ba)以这种方式可以解释:渐近扩散gydF4y2BaαgydF4y2Ba0gydF4y2Ba≈0.01,平均分散的零斜率情节的范围仅限于意味着计数到100年左右,的倒数gydF4y2BaαgydF4y2Ba0gydF4y2Ba。然而,过高的gydF4y2BaαgydF4y2Ba低、几乎没有对推理的影响范围,在这个范围内方差gydF4y2BavgydF4y2Ba是无论如何主导gydF4y2BaαgydF4y2Ba独立的泊松组件gydF4y2BavgydF4y2BaPgydF4y2Ba。Pickrell数据的情况是不同的:在这里,分散的依赖意味着观察计数明显高于渐近扩散的倒数gydF4y2BaαgydF4y2Ba0gydF4y2Ba(图gydF4y2Ba1gydF4y2BaB),因此并不是因为仅仅估计偏差。模拟(见附加文件gydF4y2Ba1gydF4y2Ba:图S25)证实,观察到的联合分布的估计分散体系和手段单一,不兼容常数分散。因此,参数化(gydF4y2Ba6gydF4y2Ba)是一个灵活的和温和的保守建模的选择:它能够接dispersion-mean依赖如果它存在,虽然它的权力可能会导致轻微的损失低、范围由于倾向于高估分散。gydF4y2Ba
分散之前gydF4y2Ba也观察到gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba6gydF4y2Ba),前一个对数正态分布符合典型的观察弥散分布RNA-seq数据集。我们解决处理的计算困难non-conjugate之前使用以下论点:对数残差的趋势,gydF4y2Ba,来自两个贡献,即真正的对数分散体系的分散的趋势,由之前的方差gydF4y2Ba抽样分布、对数的分散估计量,与方差gydF4y2Ba。抽样分布的分散估计大约是一个按比例缩小的gydF4y2BaχgydF4y2Ba2gydF4y2Ba分布与gydF4y2Ba米gydF4y2Ba−gydF4y2BapgydF4y2Ba自由度,gydF4y2Ba米gydF4y2Ba样品和数量gydF4y2BapgydF4y2Ba系数的数量。的对数的方差gydF4y2Ba给出了分布式随机变量(gydF4y2Ba52gydF4y2Batrigamma)的函数gydF4y2BaψgydF4y2Ba1gydF4y2Ba,gydF4y2Ba
因此,gydF4y2Ba,也就是说,the年代一个米pling variance of the logarithm of a variance or dispersion estimator is approximately constant across genes and depends only on the degrees of freedom of the model.
额外的文件gydF4y2Ba1gydF4y2Ba:表S2比较这个近似对数离差的方差估计的方差对数Cox-Reid调整分散估计模拟负二项数据,在不同的样本大小、数量的参数和色散值用于创建模拟数据。样本方差的近似接近不同的典型值gydF4y2Ba米gydF4y2Ba,gydF4y2BapgydF4y2Ba和gydF4y2BaαgydF4y2Ba。gydF4y2Ba
因此,先验方差gydF4y2Ba通过减去预计抽样方差估计的对数残差的方差,gydF4y2Ba:gydF4y2Ba
之前的方差gydF4y2Ba是阈值最小值为0.25,这样分散估计不完全萎缩gydF4y2Ba如果对数残差的方差小于预期的抽样方差。gydF4y2Ba
为了避免通货膨胀的gydF4y2Ba由于色散离群值(即。,genes not well captured by this prior; see below), we use a robust estimator for the standard deviation年代gydF4y2BalrgydF4y2Ba的对数残差,gydF4y2Ba
疯狂代表平均绝对偏差,像往常一样除以比例因子gydF4y2BaΦgydF4y2Ba−1gydF4y2Ba(3/4)。gydF4y2Ba
三个或更少的剩余自由度gydF4y2Ba当有三个或更少的剩余自由度(数量的负样本数量的参数估计),之前的估计方差gydF4y2Ba使用对数残差的方差gydF4y2Ba倾向于低估gydF4y2Ba。在这种情况下,我们通过仿真之前估计方差。我们比赛对数残差的分布密度模拟对数残差。这些是的对数gydF4y2Ba分布式随机变量添加到gydF4y2Ba随机变量考虑到传播由于之前。模拟的分布是由−转移日志(gydF4y2Ba米gydF4y2Ba−gydF4y2BapgydF4y2Ba)占的比例gydF4y2BaχgydF4y2Ba2gydF4y2Ba分布。我们重复仿真网格的值gydF4y2Ba,并选择的值最小化Kullback-Leibler对数残差的散度从观察到的密度模拟密度。gydF4y2Ba
最后分散估计gydF4y2Ba我们形成一个对数后的色散Cox-Reid调整对数似然(gydF4y2Ba7gydF4y2Ba)和对数之前(gydF4y2Ba5gydF4y2Ba),利用其最大(即。,地图v一个lue) as the final estimate of the dispersion,
在哪里gydF4y2Ba
一个积分常数,之前的密度的对数(gydF4y2Ba5gydF4y2Ba)。再次,回溯搜索用于执行优化。gydF4y2Ba
分散离群值gydF4y2Ba对于某些基因,gene-wise估计gydF4y2Ba可以这么远高于之前的期望gydF4y2Ba之前,是不合理的假设适用于基因。如果这样的分散估计基因down-moderated向安装的趋势,这可能导致假阳性。因此,我们将考虑基因的启发式作为色散离群值,如果剩余的趋势符合对数残差大于两个标准差,gydF4y2Ba年代gydF4y2BalrgydF4y2Ba(见方程(gydF4y2Ba8gydF4y2Ba)),符合以上,也就是说。,如果gydF4y2Ba
对于这类基因,gene-wise估计gydF4y2Ba不是之前向趋于萎缩的意思。而不是映射值gydF4y2Ba,我们使用gene-wise估计gydF4y2Ba作为最后的色散值在随后的步骤。此外,迭代拟合上述参数分散趋势过程避免了这样的色散异常值影响之前的意思。gydF4y2Ba
收缩估计对数褶皱的变化gydF4y2Ba
将经验贝叶斯收缩的利物浦,我们假设一个zero-centered之前正常的系数gydF4y2BaβgydF4y2Ba红外gydF4y2Ba的模型(gydF4y2Ba2gydF4y2Ba),代表利物浦(即。,typically, all coefficients except for the interceptβgydF4y2Ba我gydF4y2Ba0gydF4y2Ba):gydF4y2Ba
与微分表达式使用微阵列分析,观察基因与低强度值会从一个小信噪比。替代估计可以发现更稳定的标准计算折叠变化的比率平均观测值为每个条件(gydF4y2Ba53gydF4y2Ba]- [gydF4y2Ba55gydF4y2Ba]。gydF4y2BaDESeq2gydF4y2Ba的方法可以被视为一个扩展这些方法的稳定的基因表达改变折叠计数数据估算。gydF4y2Ba
之前经验估计gydF4y2Ba宽度之前获得的经验值gydF4y2BaσgydF4y2BargydF4y2Ba为模型系数,我们再次近似一个完整的经验贝叶斯方法,与分散估计之前,虽然我们不减去预期的抽样方差观测方差的极大似然估计。利物浦的估计之前宽度计算如下。我们使用标准的迭代再加权最小二乘算法(gydF4y2Ba12gydF4y2Ba)对于每一个基因的模型,方程(gydF4y2Ba1gydF4y2Ba)和(gydF4y2Ba2gydF4y2Ba),毫升的系数gydF4y2Ba。然后我们健康,每一列gydF4y2BargydF4y2Ba设计的矩阵(除了拦截),一个zero-centered大中型企业的褶皱变化的经验分布正态分布估计gydF4y2Ba。gydF4y2Ba
对异常值进行健康健壮与绝对利物浦值很高,我们使用分位数匹配:宽度gydF4y2BaσgydF4y2BargydF4y2Ba选择这样(1−gydF4y2BapgydF4y2Ba)的经验分位数的绝对值观察到利物浦,gydF4y2Ba(1−匹配gydF4y2BapgydF4y2Ba/ 2)之前的理论分位数,gydF4y2Ba,在那里gydF4y2BapgydF4y2Ba默认设置为0.05。如果我们写的理论正态分布的分位数gydF4y2Ba问gydF4y2BaNgydF4y2Ba(1−gydF4y2BapgydF4y2Ba大中型企业的利物浦)和实证上分位数gydF4y2Ba之前,那么宽度计算为:gydF4y2Ba
确保之前的宽度gydF4y2BaσgydF4y2BargydF4y2Ba将独立的基础水平的选择,分位数的估计匹配过程平均每个因素在所有可能的因素水平的对比。在确定实证上分位数,极端利物浦值(gydF4y2Ba规模,在基地或10 2)被排除在外。gydF4y2Ba
最后估计对数褶皱的变化gydF4y2Ba对数后的向量,gydF4y2Ba模型系数的,gydF4y2BaβgydF4y2Ba红外gydF4y2Ba基因gydF4y2Ba我gydF4y2Ba是漠视的对数可能性的总和(gydF4y2Ba2gydF4y2Ba)和前密度的对数(gydF4y2Ba10gydF4y2Ba),它的最高收益率最终映射系数估计:gydF4y2Ba
在哪里gydF4y2Ba
和gydF4y2BaαgydF4y2Ba我gydF4y2Ba是最后的分散估计基因吗gydF4y2Ba我gydF4y2Ba,也就是说,gydF4y2Ba,除了分散异常值,gydF4y2Ba。gydF4y2Ba
这个词gydF4y2BaΛgydF4y2Ba(gydF4y2BaβgydF4y2Ba),即。,日志一个r我th米of the density of the normal prior (up to an additive constant), can be read as a ridge penalty term, and therefore, we perform the optimization using the迭代再加权岭回归算法gydF4y2Ba(gydF4y2Ba56gydF4y2Ba),也被称为gydF4y2Ba加权更新gydF4y2Ba(gydF4y2Ba57gydF4y2Ba]。具体地说,对于一个给定的基因的更新的表单gydF4y2Ba
与gydF4y2Ba和gydF4y2Ba
在当前安装的价值观gydF4y2Ba从目前的估计计算吗gydF4y2Ba在每个迭代中。gydF4y2Ba
费舍尔的信息。gydF4y2Bazero-centered正常之前的效果可以被理解为缩小地图利物浦估计基于实验提供的信息量系数,在这里我们简要阐述。具体地说,对于一个给定的基因gydF4y2Ba我gydF4y2Ba,利物浦的收缩gydF4y2BaβgydF4y2Ba红外gydF4y2Ba取决于gydF4y2Ba观察费舍尔信息gydF4y2Ba,由gydF4y2Ba
在哪里gydF4y2Ba的对数可能性,和偏导数对利物浦gydF4y2BaβgydF4y2Ba红外gydF4y2Ba。负二项的漠视,观察费舍尔信息,或尖峰的对数可能性,受很多因素的影响,包括自由度,估计意味着计数gydF4y2BaμgydF4y2BaijgydF4y2Ba,基因的分散估计gydF4y2BaαgydF4y2Ba我gydF4y2Ba。之前影响地图估计可能性的密度和前乘以计算后。较低的基因估计的平均值gydF4y2BaμgydF4y2BaijgydF4y2Ba或高分散估计gydF4y2BaαgydF4y2Ba我gydF4y2Ba可能奉承的档案,数据集一样很少有剩余的自由度,因此在这些情况下zero-centered之前把地图估计从大中型企业的逼近零高度的不确定性。gydF4y2Ba
瓦尔德测试gydF4y2Ba
瓦尔德测试比较了β估计gydF4y2BaβgydF4y2Ba红外gydF4y2Ba除以它的估计标准误差SE (gydF4y2BaβgydF4y2Ba红外gydF4y2Ba标准正态分布)。估计标准误差的平方根是估计协方差矩阵的对角元素,gydF4y2BaΣgydF4y2Ba我gydF4y2Ba系数,也就是说,gydF4y2Ba。对比系数测试同样的形成一个瓦尔德统计使用(gydF4y2Ba3gydF4y2Ba)和(gydF4y2Ba4gydF4y2Ba)。我们使用以下公式系数的协方差矩阵的漠视与正常之前系数(gydF4y2Ba56gydF4y2Ba]、[gydF4y2Ba58gydF4y2Ba]:gydF4y2Ba
标准正态分布的尾部积分乘以2实现双尾检验。瓦尔德测试gydF4y2BaPgydF4y2Ba值的基因子集,通过独立过滤步骤调整为多个测试使用Benjamini和业务的过程gydF4y2Ba21gydF4y2Ba]。gydF4y2Ba
独立的过滤gydF4y2Ba
独立过滤不妥协i型错误控制只要略微检验统计量的分布是统计独立的过滤器gydF4y2Ba在虚假设条件下gydF4y2Ba(gydF4y2Ba22gydF4y2Ba),我们认为在下面,这是在我们的应用程序。过滤数据的gydF4y2BaDESeq2gydF4y2Ba的意思是规范化的重要基因,而检验统计量是什么gydF4y2BapgydF4y2Ba,gydF4y2BaPgydF4y2Ba从瓦尔德测试值。我们首先考虑的情况gene-wise色散的大小因素是相等的,估计是用于每个基因,即没有色散收缩。家庭的负二项分布参数化gydF4y2BaθgydF4y2Ba= (gydF4y2BaμgydF4y2Ba,gydF4y2BaαgydF4y2Ba)。除了不连续性的gydF4y2BapgydF4y2Ba由于低计数,对于一个给定的gydF4y2BaμgydF4y2Ba的分布gydF4y2BapgydF4y2Ba是统一的(0,1)在零假设下,gydF4y2BapgydF4y2Ba是一个辅助数据。样本均值的基因gydF4y2Ba我gydF4y2Ba,gydF4y2Ba有限,完全足够了gydF4y2BaμgydF4y2Ba。然后从苏定理,gydF4y2Ba和gydF4y2BapgydF4y2Ba是独立的。gydF4y2Ba
而对于非常低,一个可以观察到的不连续性和不均匀性gydF4y2BapgydF4y2Ba在零假设下,gydF4y2BaDESeq2gydF4y2Ba不使用的分布gydF4y2BapgydF4y2Ba在估计过程——例如,gydF4y2BaDESeq2gydF4y2Ba不估计零基因的比例使用的分布gydF4y2BapgydF4y2Ba——所以这类的依赖关系gydF4y2BapgydF4y2Ba在gydF4y2BaμgydF4y2Ba不会导致i型错误。gydF4y2Ba
如果在样本大小的因素是不平等的,但不是与条件,调节的意思gydF4y2Ba归一化gydF4y2Ba还应该提供均匀分布gydF4y2BapgydF4y2Ba与调节的意思,gydF4y2Ba。我们可以考虑病理情况下,大小因素完全驳倒与条件,在这种情况下,即使在零假设下,意味着较低的基因数的不均匀分布gydF4y2BapgydF4y2Ba,因为一个条件可能有积极的方面和其他条件通常为零。这可能导致不均匀的gydF4y2BapgydF4y2Ba在虚假设条件下;然而,这样一个病态的情况下会造成问题对于许多差异的统计测试的意思。gydF4y2Ba
我们使用仿真证明零的独立性检验统计量的分布从过滤器统计仍然适用于分散收缩。额外的文件gydF4y2Ba1gydF4y2Ba:图S26显示边际的零分布gydF4y2BapgydF4y2Ba整个范围的意思是规范化。尽管分布峰值最低的基因意味着重要的由于数据的离散性,这些密度几乎是统一整个范围的平均强度表达式。gydF4y2Ba
复合零假设gydF4y2Ba
DESeq2gydF4y2Ba提供测试组合形式的零假设gydF4y2Ba寻找基因的利物浦大大超过一个阈值gydF4y2BaθgydF4y2Ba> 0。复合零假设被两个简单的零假设:gydF4y2Ba和gydF4y2Ba。双尾gydF4y2BaPgydF4y2Ba值是由集成为中心的正态分布gydF4y2BaθgydF4y2Ba用标准差SE (gydF4y2BaβgydF4y2Ba红外gydF4y2Ba)|gydF4y2BaβgydF4y2Ba红外gydF4y2Ba|向gydF4y2Ba∞gydF4y2Ba。积分的值然后乘以2和阈值1。即使这个过程控制i型错误gydF4y2BaβgydF4y2Ba红外gydF4y2Ba=±gydF4y2BaθgydF4y2Ba,相当于标准gydF4y2BaDESeq2gydF4y2BaPgydF4y2Ba值的时候gydF4y2BaθgydF4y2Ba= 0。gydF4y2Ba
相反,当寻找基因的绝对利物浦显著低于一个阈值,即:零假设,当测试gydF4y2Ba,gydF4y2BaPgydF4y2Ba值构造成的最大的两个片面的测试简单的零假设:gydF4y2Ba和gydF4y2Ba。片面gydF4y2BaPgydF4y2Ba值是由集成为中心的正态分布gydF4y2BaθgydF4y2Ba用标准差SE (gydF4y2BaβgydF4y2Ba红外gydF4y2Ba)gydF4y2BaβgydF4y2Ba红外gydF4y2Ba向−gydF4y2Ba∞gydF4y2Ba−和集成为中心的正态分布gydF4y2BaθgydF4y2Ba用标准差SE (gydF4y2BaβgydF4y2Ba红外gydF4y2Ba)gydF4y2BaβgydF4y2Ba红外gydF4y2Ba向gydF4y2Ba∞gydF4y2Ba。gydF4y2Ba
注意,虽然在利物浦之前zero-centered符合测试小利物浦的零假设,它不应该用于测试大型利物浦的零假设,因为之前将支持备择假设。gydF4y2BaDESeq2gydF4y2Ba要求没有之前的零假设在测试使用大的利物浦,这样数据就必须提供证据对零假设。gydF4y2Ba
的相互作用gydF4y2Ba
两个例外的违约gydF4y2BaDESeq2gydF4y2Ba利物浦估计步骤用于实验设计与交互。首先,当任何交互条款中包括设计,前利物浦宽度主要影响方面不是估计的数据,但设置为大值(gydF4y2Ba,或1000 2规模)固定在底座上。这确保了收缩的主要影响方面不会造成假阳性电话互动的意义。第二,当交互,所有因素都包含两个层次,然后使用标准设计矩阵而不是扩展模型矩阵,这样只有一个术语是用来测试的零假设,结合两种效果仅仅是添加剂在对数刻度。gydF4y2Ba
正规化的对数gydF4y2Ba
rlog转换计算如下。实验设计矩阵gydF4y2BaXgydF4y2Ba被替换的设计矩阵与一个指示符变量每个样本除了一个拦截列。一个模型中描述方程(gydF4y2Ba1gydF4y2Ba)和(gydF4y2Ba2gydF4y2Ba)符合zero-centered正常使用安装前按难以截获色散值gydF4y2Ba捕获的数据集的总体variance-mean依赖。真正的实验设计矩阵gydF4y2BaXgydF4y2Ba然后只用于估计variance-mean趋势对所有基因。无人监督的分析,例如样本质量评估,是理想的实验设计没有影响变换,因此gydF4y2BaDESeq2gydF4y2Ba默认情况下忽略了设计矩阵和估算分散治疗所有的样品复制,即。,它使用gydF4y2Ba盲目的gydF4y2Ba分散估计。rlog-transformed值拟合值,gydF4y2Ba
在哪里gydF4y2BaβgydF4y2BaijgydF4y2Ba是缩小的利物浦2固定在底座上规模gydF4y2BajgydF4y2Ba样本。之前的方差将使用类似的方法用微分表达式,通过匹配zero-centered正态分布来解释观察到的利物浦。利物浦的第一个矩阵计算通过规范化的对数(基础2)计数+ pseudocountgydF4y2Ba为每个样本除以规范化计数+ pseudocountgydF4y2Ba。的pseudocountgydF4y2Ba允许计算对数比率对所有基因,和几乎没有影响的方差的估计之前或最后rlog转换。这个矩阵的利物浦就代表了相同等级对数比率每个样本的拟合值只使用一个拦截。前发现方差匹配97.5% zero-centered正态分布的分位数的95%分位数的绝对值利物浦矩阵。gydF4y2Ba
库克的距离为异常值检测gydF4y2Ba
的大中型企业gydF4y2Ba用于计算库克的距离。考虑一个基因gydF4y2Ba我gydF4y2Ba和样本gydF4y2BajgydF4y2Ba的漠视,库克的距离是由(gydF4y2Ba59gydF4y2Ba]:gydF4y2Ba
在哪里gydF4y2BaRgydF4y2BaijgydF4y2Ba皮尔森剩余的样品吗gydF4y2BajgydF4y2Ba,gydF4y2BaτgydF4y2Ba是一个overdispersion参数(负二项的漠视,gydF4y2BaτgydF4y2Ba设置为1),gydF4y2BapgydF4y2Ba参数的数量包括拦截,gydF4y2BahgydF4y2BajjgydF4y2Ba是gydF4y2BajgydF4y2Ba这顶帽子矩阵的对角元素gydF4y2BaHgydF4y2Ba:gydF4y2Ba
皮尔森残差gydF4y2BaRgydF4y2BaijgydF4y2Ba计算为gydF4y2Ba
在哪里gydF4y2BaμgydF4y2BaijgydF4y2Ba负二项估计的GLM没有利物浦之前,使用方差函数gydF4y2BaVgydF4y2Ba(gydF4y2BaμgydF4y2Ba)=gydF4y2BaμgydF4y2Ba+gydF4y2BaαgydF4y2BaμgydF4y2Ba2gydF4y2Ba。矩量法估计gydF4y2Ba,使用一个健壮的估计量的方差gydF4y2Ba提供鲁棒性对离群值,使用:gydF4y2Ba
R / Bioconductor包gydF4y2Ba
DESeq2gydF4y2Ba被实现为一个包R统计环境和可用gydF4y2Ba10gydF4y2Ba]作为Bioconductor项目的一部分gydF4y2Ba11gydF4y2Ba]。数矩阵和元数据,包括基因模型和样本信息,存储在一个S4类派生的gydF4y2BaSummarizedExperimentgydF4y2Ba类的gydF4y2BaGenomicRangesgydF4y2Ba包(gydF4y2Ba60gydF4y2Ba]。gydF4y2BaSummarizedExperimentgydF4y2Ba对象包含数矩阵可以很容易地生成的使用gydF4y2BasummarizeOverlapsgydF4y2Ba的函数gydF4y2BaGenomicAlignmentsgydF4y2Ba包(gydF4y2Ba61年gydF4y2Ba]。这个工作流自动存储另外基因模型元数据和其他信息如基因组和基因注释的版本。其他方法获得数矩阵包括gydF4y2Bahtseq-countgydF4y2Ba脚本(gydF4y2Ba62年gydF4y2Ba)和Bioconductor包gydF4y2BaeasyRNASeqgydF4y2Ba(gydF4y2Ba63年gydF4y2Ba),gydF4y2BafeatureCountgydF4y2Ba(gydF4y2Ba64年gydF4y2Ba]。gydF4y2Ba
的gydF4y2BaDESeq2gydF4y2Ba包有一个详细的装饰图案,它是通过一些例子微分表达式分析真实数据集,并使用rlog转换为质量评价和可视化。一个函数,调用gydF4y2BaDESeqgydF4y2Ba用于运行默认的分析,而低级函数也可用于高级用户。gydF4y2Ba
读底部对齐gydF4y2Baet al。gydF4y2Ba和PickrellgydF4y2Baet al。gydF4y2Ba数据集gydF4y2Ba
读是一致使用TopHat2对准器[gydF4y2Ba65年gydF4y2Ba),并分配给使用的基因gydF4y2BasummarizeOverlapsgydF4y2Ba的函数gydF4y2BaGenomicRangesgydF4y2Ba包(gydF4y2Ba60gydF4y2Ba]。Pickrell的序列读取存档fastq文件gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba17gydF4y2Ba加入]数据集(号码(SRA: SRP001540])是一致的gydF4y2Ba智人gydF4y2Ba参考序列从Illumina公司iGenomes GRCh37下载2013年3月。读取在定义的基因数运用GTF文件,发布70年,包含在Illumina公司iGenome。顺序读取存档fastq文件的底部gydF4y2Baet al。gydF4y2Ba(gydF4y2Ba16gydF4y2Ba加入]数据集(号码(SRA: SRP004777])是一致的gydF4y2Ba亩骶gydF4y2Ba参考序列从Illumina公司iGenomes NCBIM37下载2013年3月。读取在定义的基因数运用GTF文件,发布66年,包含在Illumina公司iGenome。gydF4y2Ba
复制代码gydF4y2Ba
Sweave片段复制所有数据和表,包括实验数据对象所提到的,和代码将读取和基准测试,可以在一个包中找到gydF4y2BaDESeq2papergydF4y2Ba(gydF4y2Ba66年gydF4y2Ba]。gydF4y2Ba
额外的文件gydF4y2Ba
缩写gydF4y2Ba
- 罗斯福:gydF4y2Ba
-
错误发现率gydF4y2Ba
- 全球语言监测机构:gydF4y2Ba
-
广义线性模型gydF4y2Ba
- 高温超导:gydF4y2Ba
-
高通量测序gydF4y2Ba
- 利物浦:gydF4y2Ba
-
对数褶皱变化gydF4y2Ba
- 地图:gydF4y2Ba
-
最大gydF4y2Ba后验gydF4y2Ba
- 大中型企业:gydF4y2Ba
-
最大似然估计gydF4y2Ba
- RNA-seq:gydF4y2Ba
-
RNA序列gydF4y2Ba
- 威仕特:gydF4y2Ba
-
Variance-stabilizing转换gydF4y2Ba
引用gydF4y2Ba
- 1。gydF4y2Ba
Lonnstedt我,速度T:复制微阵列数据。统计学报。2002年,12:脉络。gydF4y2Ba
- 2。gydF4y2Ba
罗宾逊博士Smyth GK:主持统计测试来评估不同的标签。生物信息学。2007年,23日:2881 - 2887。10.1093 /生物信息学/ btm453。gydF4y2Ba
- 3所示。gydF4y2Ba
麦卡锡DJ,陈Y,史密斯GK:微分表达式的分析多因素RNA-seq实验对生物变异。核酸研究》2012年,40:4288 - 4297。10.1093 / nar / gks042。gydF4y2Ba
- 4所示。gydF4y2Ba
W:安德斯年代,Huber微分表达式分析序列计数数据。基因组医学杂志2010年11:106 - 10.1186 / gb - 2010 - 11 - 10 - r106。gydF4y2Ba
- 5。gydF4y2Ba
周Y-H,夏K,赖特FA:一个功能强大且灵活的RNA序列统计数据的分析方法。生物信息学。2011年,27日:2672 - 2678。10.1093 /生物信息学/ btr449。gydF4y2Ba
- 6。gydF4y2Ba
王吴H, C,吴Z:一种新的色散收缩估计量提高了检测RNA-seq数据微分表达式。生物统计学。2013年,14:232 - 243。10.1093 /生物统计学/ kxs033。gydF4y2Ba
- 7所示。gydF4y2Ba
Hardcastle T,凯利K: baySeq:经验贝叶斯方法确定微分表达式序列计数数据。BMC生物信息学。2010年,11:422 - 10.1186/1471 - 2105 - 11 - 422。gydF4y2Ba
- 8。gydF4y2Ba
Van De由马、Leday应用GGR Pardo L H街,范德法特啊,范Wieringen WN:核糖核酸测序数据的贝叶斯分析由多个收缩先验估计。生物统计学。2013年,14:113 - 128。10.1093 /生物统计学/ kxs031。gydF4y2Ba
- 9。gydF4y2Ba
布尔JM, Huber周Sultmann H,威尔默F·冯·Heydebreck,哈斯年代,Korn B, B谷纳温,出售,Fuzesi L, Vingron M, Poustka:识别和分类在肾细胞癌的差异表达基因的表达分析在全球人类31500 -元素cDNA数组。基因组研究》2001年11:1861 - 1870。gydF4y2Ba
- 10。gydF4y2Ba
DESeq2。(gydF4y2Bahttp://www.bioconductor.org/packages/release/bioc/html/DESeq2.htmlgydF4y2Ba]gydF4y2Ba
- 11。gydF4y2Ba
绅士RC,凯里VJ,贝茨DM Bolstad B, Dettling M, Dudoit年代,埃利斯B, Gautier L,通用电气Y,贵族J, Hornik K, Hothorn T, Huber W, Iacus年代,伊R, Leisch F,李C, Maechler M,罗西尼AJ, Sawitzki G、C史密斯,史密斯G, Tierney L,杨司法院,张J: Bioconductor:打开软件开发计算生物学和生物信息学。基因组医学杂志。2004年,5:r80 - 10.1186 / gb - 2004 - 5 - 10 - r80。gydF4y2Ba
- 12。gydF4y2Ba
McCullagh P, Nelder JA:广义线性模型。专著统计&应用概率。1989年,查普曼&大厅/ CRC,伦敦,英国,gydF4y2Ba
- 13。gydF4y2Ba
汉森KD,伊RA,吴Z:去除技术变化使用条件分位数RNA-seq数据规范化。生物统计学。2012年,13:204 - 216。10.1093 /生物统计学/ kxr054。gydF4y2Ba
- 14。gydF4y2Ba
Risso D,施瓦茨K,夏洛克G, Dudoit S: GC-content RNA-seq数据规范化。BMC生物信息学。2011年,12:480 - 10.1186/1471 - 2105 - 12 - 480。gydF4y2Ba
- 15。gydF4y2Ba
史密斯GK:线性模型和经验贝叶斯方法评估微分表达式在微阵列实验。统计:麝猫杂志。2004年,3:1 - 25。gydF4y2Ba
- 16。gydF4y2Ba
底D,沃尔特·NAR猎人我,Darakjian P, Kawane年代,巴克KJ,瑟RP,穆尼M, McWeeney SK, Hitzemann R:评估基因表达在C57BL / 6 j和DBA / 2 j鼠纹状体使用RNA-seq和微阵列。PLoS ONE。2011年,6:17820 - 10.1371 / journal.pone.0017820。gydF4y2Ba
- 17所示。gydF4y2Ba
Pickrell JK Marioni JC, Pai AA, Degner摩根富林明,恩格尔哈特,Nkadori E, Veyrieras jb,史蒂芬斯M,吉拉德·Y, Pritchard JK:理解人类基因表达变异机制与RNA序列。大自然。2010年,464:768 - 772。10.1038 / nature08872。gydF4y2Ba
- 18岁。gydF4y2Ba
Hastie T, Tibshirani R,弗里德曼J:统计学习的元素:数据挖掘、推理和预测。2009年,施普林格,美国纽约gydF4y2Ba
- 19所示。gydF4y2Ba
Bi Y, Davuluri R: NPEBseq:非参数经验Bayesian-based RNA-seq数据的分析过程的微分表达式。BMC生物信息学。2013年,14:262 - 10.1186/1471 - 2105 - 14 - 262。gydF4y2Ba
- 20.gydF4y2Ba
刘冯J, Meyer CA,王Q, JS,刘x, Y:张GFOLD:广义褶皱变化从RNA-seq数据排名差异表达基因。生物信息学。2012年,28日:2782 - 2788。10.1093 /生物信息学/ bts515。gydF4y2Ba
- 21。gydF4y2Ba
业务,Benjamini Y Y:控制错误发现率:一种实用和强大的多个测试方法。J R统计Soc Ser B Methodol。1995年,57:289 - 300。gydF4y2Ba
- 22。gydF4y2Ba
Bourgon R, R绅士,Huber W:独立过滤增加了高通量实验的检测能力。《美国国家科学院刊。2010年,107:9546 - 9551。10.1073 / pnas.0914005107。gydF4y2Ba
- 23。gydF4y2Ba
麦卡锡DJ,史密斯GK:测试意义相对于叠化阈值是一个治疗。生物信息学。2009年,25岁:765 - 771。10.1093 /生物信息学/ btp053。gydF4y2Ba
- 24。gydF4y2Ba
李江,Tibshirani R:找到一致的模式:非参数方法确定微分表达式RNA-seq数据。地中海统计方法研究》2013年,22日:519 - 536。10.1177 / 0962280211428386。gydF4y2Ba
- 25。gydF4y2Ba
库克理查德·道金斯:检测线性回归的影响观察。技术计量学。1977年,19:15 - 18。10.2307 / 1268249。gydF4y2Ba
- 26岁。gydF4y2Ba
王锤P, Banck女士,Amberg R, C, Petznick G,罗年代,Khrebtukova我Schroth GP, Beyerlein P,布鲁斯:mRNA-seq与不可知论者拼接网站发现慢性疼痛的神经系统转录组进行测试。基因组研究》2010年,20:847 - 860。10.1101 / gr.101204.109。gydF4y2Ba
- 27。gydF4y2Ba
Frazee A、B Langmead韭菜J:重新计票:multi-experiment资源analysis-ready RNA-seq基因数据集。BMC生物信息学。2011年,12:449 - 10.1186/1471 - 2105 - 12 - 449。gydF4y2Ba
- 28。gydF4y2Ba
杰尔C, Hendrickson DG,工作M,高夫L, Rinn杰,Pachter L:微分分析基因调控的转录与RNA-seq决议。生物科技Nat》。2012年,31:46-53。10.1038 / nbt.2450。gydF4y2Ba
- 29。gydF4y2Ba
Glaus P, Honkela Rattray M:识别差异表达从RNA-seq数据记录与生物变异。生物信息学。2012年,28日:1721 - 1728。10.1093 /生物信息学/ bts260。gydF4y2Ba
- 30.gydF4y2Ba
W:安德斯年代,雷耶斯,Huber检测从RNA-seq数据微分使用外显子。基因组研究》2012年,22日:2008 - 2017。10.1101 / gr.133744.111。gydF4y2Ba
- 31日。gydF4y2Ba
Sammeth M:完整的可变剪接事件是泡沫拼接图。J第一版。2009年,16:1117 - 1140。10.1089 / cmb.2009.0108。gydF4y2Ba
- 32。gydF4y2Ba
页面H, Bindreither D,卡尔森M,摩根M: SplicingGraphs:创建、操作、可视化拼接图,并分配RNA-seq them2013读取。Bioconductor包(gydF4y2Bahttp://www.bioconductor.orggydF4y2Ba]gydF4y2Ba
- 33。gydF4y2Ba
罗宾逊博士麦卡锡DJ,史密斯GK:磨边机:Bioconductor包微分表达式数字基因表达数据的分析。生物信息学。2009年,26日:139 - 140。10.1093 /生物信息学/ btp616。gydF4y2Ba
- 34。gydF4y2Ba
周X,林赛H,罗宾逊MD:强劲检测微分表达式使用观察RNA序列数据的权重。核酸研究》2014年,42:e91 - 10.1093 / nar / gku310。gydF4y2Ba
- 35。gydF4y2Ba
愣N,道森JA,汤姆森是的,Ruotti V, Rissman AI,史密特BMG, Haag JD,古尔德MN,斯图尔特RM, Kendziorski C: EBSeq:实证贝叶斯推理的层次模型RNA-seq实验。生物信息学。2013年,29日:1035 - 1043。10.1093 /生物信息学/ btt087。gydF4y2Ba
- 36。gydF4y2Ba
陈法连续波,Y, W,史密斯GK:轰:精密权重解锁线性模型分析工具对RNA-seq读计数。基因组医学杂志。2014年,15:29 - 10.1186 / gb - 2014 - 15 - 2 r29。gydF4y2Ba
- 37岁。gydF4y2Ba
休伯特L, Arabie P:比较分区。J Classif。1985年,2:193 - 218。10.1007 / BF01908075。gydF4y2Ba
- 38。gydF4y2Ba
威滕DM:分类和聚类的测序数据使用泊松模型。安:Stat。2011年,5:2493 - 2518。10.1214 / 11-aoas493。gydF4y2Ba
- 39岁。gydF4y2Ba
哈伊RA,吴Z,贾菲:比较affymetrix GeneChip表达式的措施。生物信息学,2006,22日:789 - 794。10.1093 /生物信息学/ btk046。gydF4y2Ba
- 40。gydF4y2Ba
王Asangani IA Dommeti六世,X,马利克R, Cieslik M,杨R, Escara-Wilke J, Wilder-Romans K, Dhanireddy年代,Engelke C,艾耶可,经X, y M,曹X, z代表秦王年代,冯,财政年度Chinnaiyan问:治疗针对castration-resistant打赌bromodomain蛋白质的前列腺癌。大自然。2014年,510:278 - 282。10.1038 / nature13229。gydF4y2Ba
- 41岁。gydF4y2Ba
布朗鲜明的R, G: DiffBind:微分约束分析ChIP-seq data2013峰值。Bioconductor包(gydF4y2Bahttp://www.bioconductor.orggydF4y2Ba]gydF4y2Ba
- 42。gydF4y2Ba
Ross-Innes CS,鲜明的R, Teschendorff AE,福尔摩斯KA,阿里•人力资源邓宁MJ,布朗GD,枸杞子O,埃利斯IO,绿色的基于“增大化现实”技术,阿里年代,下巴S-F, Palmieri C,卡尔达斯C,卡罗尔JS:微分雌激素受体结合与乳腺癌的临床结果。大自然。2012年,481:389 - 393。gydF4y2Ba
- 43。gydF4y2Ba
罗宾逊DG,陈W、层JD Gresham D: bar-seq实验设计与分析。G3(贝塞斯达)。2013年,4:11到18门。10.1534 / g3.113.008565。gydF4y2Ba
- 44岁。gydF4y2Ba
McMurdie PJ,福尔摩斯S:不浪费,要不为什么纯化微生物数据是不可接受的。公共科学图书馆第一版杂志。2014,10:1003531 - 10.1371 / journal.pcbi.1003531。gydF4y2Ba
- 45岁。gydF4y2Ba
Vasquez J,鸿C, Vanselow JT, Schlosser, Siegel TN:比较核糖体分析揭示了大量转化的复杂性不同的锥虫属brucei生命周期阶段。核酸研究》2014年,42:3623 - 3637。10.1093 / nar / gkt1386。gydF4y2Ba
- 46岁。gydF4y2Ba
周Y,朱年代,Cai C,元P,李C,黄Y,魏W:大规模筛选CRISPR / Cas9库的功能基因组学在人类细胞。大自然。2014年,509:487 - 491。10.1038 / nature13166。gydF4y2Ba
- 47岁。gydF4y2Ba
考克斯博士,里德N:参数正交性和近似条件推理。J R统计Soc Ser B Methodol。1987年,49:1-39。gydF4y2Ba
- 48。gydF4y2Ba
罗宾逊博士Smyth GK:负二项分布的小样本估计,SAGE数据的应用程序。生物统计学。2007年,9:321 - 332。10.1093 /生物统计学/ kxm030。gydF4y2Ba
- 49。gydF4y2Ba
Pawitan Y:在所有可能性:统计建模和推理使用可能性。2001年,牛津大学出版社,美国纽约gydF4y2Ba
- 50。gydF4y2Ba
Armijo L:函数最小化李普希茨第一偏导数连续。Pac J数学。1966年,16:1 - 3。10.2140 / pjm.1966.16.1。gydF4y2Ba
- 51。gydF4y2Ba
Di Y,谢弗DW, Cumbie JS Chang JH:平衡负二项模型评估从RNA-seq基因差异表达。统计:麝猫杂志。2011年,10:28。gydF4y2Ba
- 52岁。gydF4y2Ba
阿布拉莫维茨M, Stegun我:数学函数的手册。1965年,多佛出版物,纽约,美国gydF4y2Ba
- 53岁。gydF4y2Ba
牛顿米,Kendziorski C, C里士满,布拉特纳F,徐凯西:微分表达变化比率:改善从微阵列数据统计推断关于基因表达的变化。J第一版。2001年,8:37-52。10.1089 / 106652701300099074。gydF4y2Ba
- 54。gydF4y2Ba
Huber W·冯·Heydebreck, Sultmann H, Poustka, Vingron M:方差稳定化应用微阵列数据校准和量化的微分表达式。生物信息学。2002年,18:96 - 104。10.1093 /生物信息学/ 18. suppl_1.s96。gydF4y2Ba
- 55。gydF4y2Ba
德宾BP,哈丁JS、霍金斯DM Rocke DM: variance-stabilizing基因表达微阵列数据的转换。生物信息学。2002年,18:105 - 110。10.1093 /生物信息学/ 18. suppl_1.s105。gydF4y2Ba
- 56。gydF4y2Ba
公园我:广义线性模型和正规化。gydF4y2Ba博士论文。gydF4y2Ba斯坦福大学统计系;2006年。gydF4y2Ba
- 57。gydF4y2Ba
弗里德曼J, Hastie T, Tibshirani R:正则化路径对广义线性模型通过坐标下降。J Stat Softw。2010年,33:22页。gydF4y2Ba
- 58岁。gydF4y2Ba
Cule E, Vineis P, De人工M:意义的岭回归测试基因数据。BMC生物信息学。2011年,12:372 - 10.1186/1471 - 2105 - 12 - 372。gydF4y2Ba
- 59。gydF4y2Ba
库克RD,韦斯伯格年代:在回归残差和影响。1982年,查普曼和大厅/ CRC,纽约,美国gydF4y2Ba
- 60。gydF4y2Ba
Lawrence M, Huber W,页面H, Aboyoun P,卡尔森M,绅士R,摩根太,凯里VJ:软件计算和注释基因组范围。公共科学图书馆第一版杂志。2013年,9:1003118 - 10.1371 / journal.pcbi.1003118。gydF4y2Ba
- 61年。gydF4y2Ba
页面H、V Obenchain摩根M:和操纵基因alignments2013 GenomicAlignments:表示。Bioconductor包(gydF4y2Bahttp://www.bioconductor.orggydF4y2Ba]gydF4y2Ba
- 62年。gydF4y2Ba
W:安德斯年代,所有供试PT, Huber HTSeq——一个Python框架来处理高通量测序数据。生物信息学,2015,31日:166 - 10.1093 /生物信息学/ btu638。gydF4y2Ba
- 63年。gydF4y2Ba
Delhomme N, Padioleau我,弗隆EE,斯坦梅茨LM: easyRNASeq: Bioconductor包处理RNA-seq数据。生物信息学。2012年,28日:2532 - 2533。10.1093 /生物信息学/ bts477。gydF4y2Ba
- 64年。gydF4y2Ba
廖Y,史密斯GK,施W: featureCounts:一个有效的通用程序分配顺序读取基因特性。生物信息学。2014年,30:923 - 930。10.1093 /生物信息学/ btt656。gydF4y2Ba
- 65年。gydF4y2Ba
金正日D, Pertea G,杰尔C, Pimentel H,凯利R,扎尔茨贝格S: TopHat2:准确对齐的转录组的插入,删除和基因融合。基因组医学杂志。2013年,14:36 - 10.1186 / gb - 2013 - 14 - 4 - r36。gydF4y2Ba
- 66年。gydF4y2Ba
DESeq2paper。(gydF4y2Bahttp://www-huber.embl.de/DESeq2papergydF4y2Ba]gydF4y2Ba
确认gydF4y2Ba
作者感谢所有用户DESeq和DESeq2提供了有价值的反馈。我们感谢Judith Zaugg有用的评论的手稿。MIL承认通过助学金资助国际马克斯·普朗克研究学校的计算生物学和科学计算和国立卫生研究院的资助(5 t32ca009337-33)。WH和SA承认资助欧盟第七框架计划(健康)通过项目gydF4y2Ba辐射gydF4y2Ba。我们感谢匿名审稿人提出的问题估计dispersion-mean趋势拟合偏差。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称没有利益冲突。gydF4y2Ba
作者的贡献gydF4y2Ba
所有作者开发的方法和写的手稿。MIL实现方法并进行分析。所有作者阅读和批准最终的手稿。gydF4y2Ba
电子辅料gydF4y2Ba
作者提交的原始图像文件gydF4y2Ba
权利和权限gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
爱,M.I.,Huber, W. & Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2.基因组医学杂志gydF4y2Ba15日,gydF4y2Ba550 (2014)。https://doi.org/10.1186/s13059 - 014 - 0550 - 8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s13059 - 014 - 0550 - 8gydF4y2Ba
关键字gydF4y2Ba
- 阅读数gydF4y2Ba
- 差异表达分析gydF4y2Ba
- DESeq2包gydF4y2Ba
- 观察费舍尔信息gydF4y2Ba
- 负二项推广线性模型gydF4y2Ba