摘要
严重急性呼吸系统综合症(SARS)和寨卡病毒病等新发传染病对公共卫生构成重大威胁1,2,3..尽管进行了大量的研究工作,但新疾病如何、何时和何地出现仍然是一个相当不确定的来源。中国湖北省武汉市最近报告了一种严重的呼吸道疾病。截至2020年1月25日,自2019年12月12日第一位患者住院以来,报告了至少1975例病例。流行病学调查显示,此次疫情与武汉的一个海鲜市场有关。在这里,我们研究了一名曾在市场工作的患者,她于2019年12月26日被武汉市中心医院收治,期间出现了包括发烧、头晕和咳嗽在内的严重呼吸综合征。宏基因组RNA序列4从病人的支气管肺泡灌洗液样本中鉴定出一种新的RNA病毒株Coronaviridae在这里被称为“WH-Human 1”冠状病毒(也被称为“2019-nCoV”)。病毒全基因组(29903个核苷酸)的系统发育分析显示,该病毒与一组sars样冠状病毒(β冠状病毒属,沙贝病毒亚属)的亲缘关系最密切(核苷酸相似性为89.1%),此前在中国蝙蝠中发现过5.这次疫情突出表明,病毒从动物身上蔓延到人类身上造成严重疾病的持续能力。
主要
该患者为41岁男性,无肝炎、肺结核和糖尿病病史。发病6天后,于2019年12月26日在武汉市中心医院住院治疗。患者在就诊时报告发热、胸闷、干咳、疼痛和虚弱1周(见表)1).对心血管、腹部和神经系统的体格检查均为正常。轻度淋巴减少症(定义为小于9 × 105在全血细胞计数试验中,白细胞和血小板计数正常。C反应蛋白水平升高(41.4 mg l−1的血液;参考范围,0-6毫克l−1)在血液化学测试中,观察到天冬氨酸转氨酶、乳酸脱氢酶和肌酸激酶的水平略有升高。该患者有轻度低氧血症,动脉血气测试确定氧水平为67毫米汞柱。入院第一天(发病后第6天),胸片异常,伴有空气空间阴影,如磨玻璃影、局灶性实变和双肺斑片状实变(扩展数据图。1).胸部计算机断层扫描显示双侧局灶性实变、大叶性实变和斑片状实变,尤其是下肺(扩展数据图)。1模拟).入院后第5天(发病后第11天)胸片显示双侧弥漫性斑片状模糊阴影(扩展数据图)。1e).初步病原学调查排除了流感病毒的存在,衣原体肺炎和肺炎支原体使用商业病原体抗原检测试剂盒,并经PCR证实。其他常见呼吸道病原体,包括人类腺病毒,也通过定量PCR (qPCR)检测为阴性(扩展数据图)。2).尽管给予了抗生素、抗病毒药物和糖皮质激素的联合治疗,但患者出现了呼吸衰竭,并给予了高流量无创通气。经过3天的治疗,患者病情没有好转,进入重症监护病房。患者入院6天后转武汉市另一医院治疗。
武汉市疾病预防控制中心的流行病学调查显示,该患者在当地一家室内海鲜市场工作。值得注意的是,除了鱼类和贝类,在疫情开始前,市场上还出售各种野生动物,包括刺猬、獾、蛇和鸟类(斑鸠),以及动物尸体和动物肉。没有蝙蝠出售。虽然病人可能在市场上接触过野生动物,但他回忆说没有接触活家禽。
为了研究与本病相关的可能病因,我们收集了支气管肺泡灌洗液(BALF)并进行了深度转录组测序。临床标本在上海市公共卫生临床中心生物安全三级实验室处理。从200 μl BALF中提取总RNA,利用Illumina MiniSeq构建元转录组文库进行150-bp reads的对端测序,如前所述4,6,7,8.最终,我们获得了56,565,928条重新组装的序列,并筛选了潜在的致病因子。由Megahit组装的384096个零件9其中最长的(30474个核苷酸(nt))丰度高,与此前在中国采集的蝙蝠sars样冠状病毒(CoV)分离物蝙蝠SL-CoVZC45 (GenBank登录号MG772933)密切相关,核苷酸同源性为89.1%(补充表)1,2).采用逆转录聚合酶链反应(RT-PCR)对该病毒的基因组序列及其末端进行了测定和确认。10和5 ' /3 ' cDNA末端快速扩增(RACE)。该病毒株被命名为wh -人1冠状病毒(WHCV)(也被称为“2019-nCoV”),其全基因组序列(29903 nt)被指定为GenBank登录号MN908947。将rna测序数据重新定位到WHCV的全基因组,得到123,613个reads,提供了99.99%的基因组覆盖率,平均深度为6.04×(范围,0.01-78.84×)(扩展数据图)。3.).经qPCR检测,BALF标本的病毒载量为3.95 × 108(扩展数据图。4).
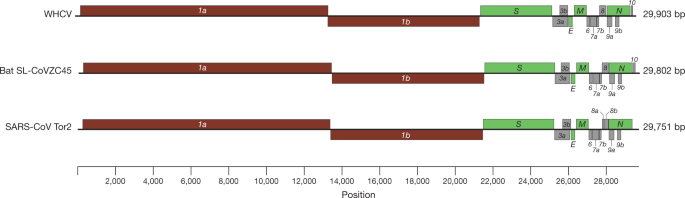
WHCV的病毒基因组结构是通过对两个Betacoronavirus属的代表性成员的序列比对确定的:一个与人类相关的冠状病毒(SARS-CoV Tor2, GenBank登录号AY274119)和一个与蝙蝠相关的冠状病毒(bat SL-CoVZC45, GenBank登录号MG772933)。在此基础上绘制了WHCV的非翻译区和开放阅读框(open-reading frame, ORF)。WHCV病毒基因组与这两种冠状病毒相似(图。1和补充表3.).5′~ 3′基因的排列顺序为:复制酶ORF1ab,尖峰(年代),信封(E),膜(米),核衣壳(N).WHCV有5 '和3 '端序列,是乙型冠状病毒的典型特征,5 '端265 nt, 3 '端229 nt。预测的复制酶ORF1ab基因全长21,291 nt,包含16个预测的非结构蛋白(Supplementary Table)4),其次是(至少)13个下游orf。此外,WHCV共享一个高度保守的结构域(LLRKNGNKG:氨基酸122-130)nsp1冠。预测的年代,ORF3a,E,米和NWHCV的基因长度分别为3822、828、228、669和1260 nt。除了这些由所有Sarbecovirus亚属成员共享的ORF区域外,WHCV与SARS-CoV相似,因为它携带预测的ORF8基因(长度366 nt)位于米和NORF的基因。WHCV orf的功能在已知冠状病毒的基础上进行了预测,并在补充表中进行了描述5.与SARS-CoV Tor2类似,WHCV的ORF 5 '端上游很容易识别出一个leader transcription regulatory sequence (TRS)和9个假定体TRS,假定保守的TRS核心序列以两种形式出现:acgaac或CUAAAC (Supplementary Table)6).
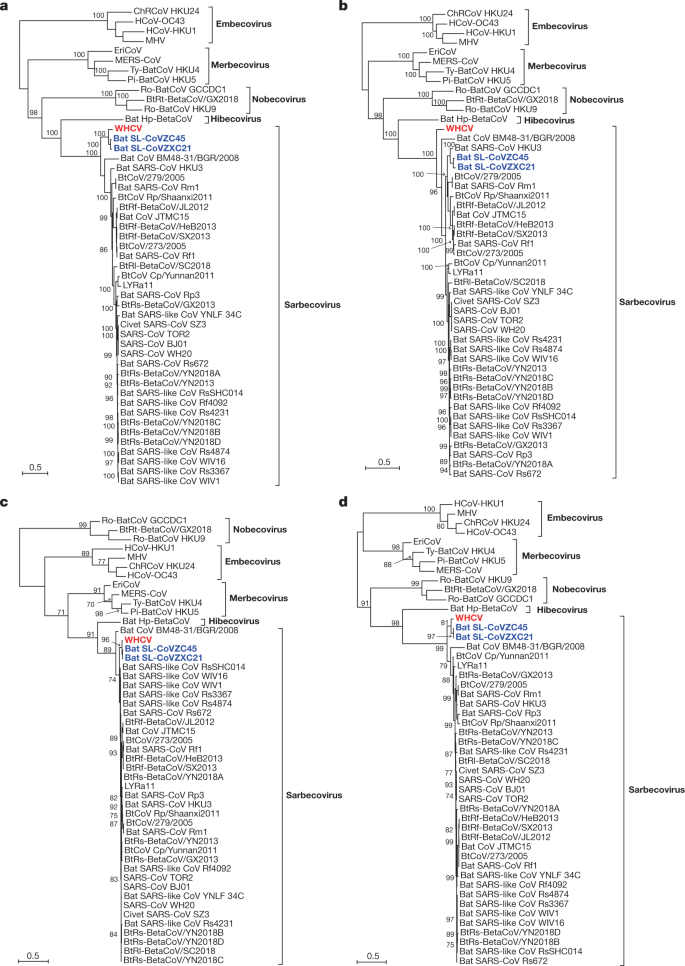
为了确定WHCV与之前发现的冠状病毒之间的进化关系,我们基于全基因组序列的核苷酸序列(非结构蛋白基因)估计了系统发生树ORF1a和ORF1b的主要结构蛋白年代,E,米和N基因(图。2和扩展数据图。5).在所有的系统发育中,WHCV与沙伯病毒亚属的成员聚集在一起,包括导致全球SARS大流行的SARS- cov1,2以及一些从蝙蝠身上获得的类似sars的冠状病毒5,11,12,13.然而,WHCV在Sarbecovirus亚属中的拓扑位置取决于使用的基因,这表明这组病毒过去曾发生过重组(图。2和扩展数据图。5).具体地说,在年代(扩展数据图5)WHCV与蝙蝠冠状病毒SL-CoVZC45最为密切相关,氨基酸同源性为82.3%(与SARS-CoV的氨基酸同源性约为77.2%;补充表3.),而在ORF1b系统发育中,WHCV处于Sarbecovirus亚属的基础位置(图。2).在复制酶多蛋白pp1ab的保守结构域估计的系统发育树中也观察到这种拓扑划分,这可能反映了蝙蝠肉瘤病毒之间的重组(扩展数据图)。6).
为了更好地了解WHCV感染人类的潜力,我们将其刺突蛋白的受体结合域(RBD)与sars - cov和蝙蝠sars样cov进行了比较。WHCV的RBD序列与sars - cov(73.8-74.9%氨基酸同源性)和sars样cov(75.9-76.9%氨基酸同源性)更接近,包括Rs4874、Rs7327和Rs4231(75.9-76.9%氨基酸同源性),它们能够利用人ACE2受体进入细胞11(补充表7).此外,WHCV刺突蛋白的RBD仅比sars冠状病毒刺突蛋白的RBD长1个氨基酸(扩展数据图)。7一个).相比之下,其他蝙蝠sars类cov,包括不能与人类ACE2结合的Rp3菌株14与sars - cov序列相比,-在433-437和460-472位点存在氨基酸缺失(扩展数据图)。7一个).之前确定的15SARS-CoV棘蛋白RBD的晶体结构(protein Data Bank (PDB) 2AJF)揭示了433-437和460-472区域与人类ACE2直接相互作用,因此可能对确定物种特异性很重要(扩展数据图)。7 b).我们利用SWISS-MODEL服务器对WHCV、Rs4874和Rp3刺突蛋白RBD结构域的三维蛋白质结构进行了预测,并将其与SARS-CoV刺突蛋白(PDB 2GHV) RBD结构域的晶体结构进行了比较(扩展数据图)。7c-f)根据序列比对,WHCV和Rs4874的RBD结构域的预测蛋白质结构与SARS-CoV的RBD结构域密切相关,并且与Rp3的RBD结构域的预测结构不同。此外,与其他人类冠状病毒(HKU1和OC43)相比,WHCV刺突蛋白的N端与SARS-CoV更相似(扩展数据图。8)可以与唾液酸结合16.综上所述,WHCV与SARS-CoV的RBD结构域的氨基酸序列和预测蛋白结构高度相似,提示WHCV可能有效地利用人ACE2作为细胞进入受体,可能促进人传人11,17,18.
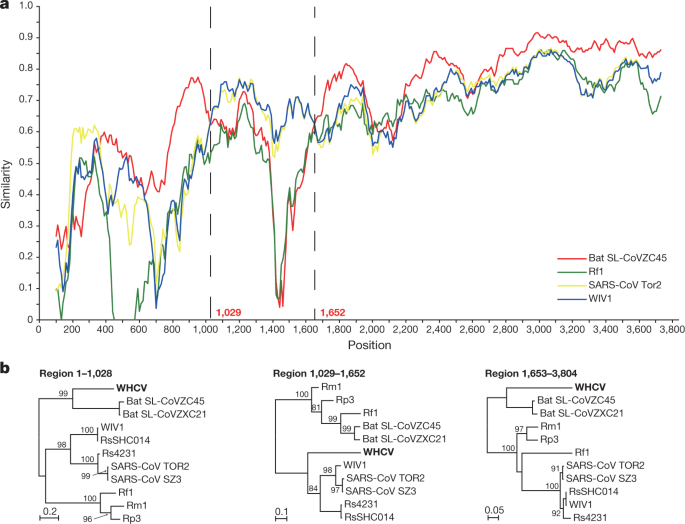
为了进一步描述sarbecovirus进化史上可能发生的重组事件,我们利用重组检测程序v.4对WHCV和四种代表性冠状病毒——蝙蝠sars样CoV Rp3、CoVZC45、CoVZXC21和SARS-CoV tor2的全基因组序列进行了分析(RDP4)19.虽然相似图表明WHCV与sars - cov或sars样cov之间可能发生了重组事件(扩展数据图)。9),没有明显的证据表明整个基因组都发生了重组。然而,一些证据过去的重组被发现在年代WHCV、SARS-CoV和蝙蝠sars样cov (WIV1和RsSHC014)基因(P< 3.147 × 10−3到P< 9.198 × 10−9),其相似性图显示在核苷酸1029和1652处存在重组断点,这两个核苷酸将年代将WHCV基因分为三个区域(图。3.).在序列的1 ~ 1029和1652 ~末端核苷酸片段的系统发育中,WHCV与蝙蝠SL-CoVZC45和蝙蝠SL-CoVZXC21的亲缘关系最密切,而在核苷酸1030至1651的区域(RBD区域),WHCV与SARS-CoV和蝙蝠sars样cov (WIV1和RsSHC014)分组,能够直接在人类传播17,20. 尽管这些重组事件在Sarbecovirus中似乎比较常见,但没有证据表明重组促进了WHCV的出现。
冠状病毒与许多人类传染病暴发有关,包括2002-2003年的SARS和2012年的中东呼吸综合征(MERS)1,21.其他四种冠状病毒——人类冠状病毒HKU1、OC43、NL63和229e也与呼吸道疾病有关22,23,24,25.尽管自2005年以来,中国已经在包括蝙蝠在内的哺乳动物中广泛发现了类似sars的冠状病毒10,26,27,28,人类感染冠状病毒的确切来源仍不清楚。在这里,我们描述了一种新的冠状病毒- whcv -在BALF中从中国武汉经历了严重呼吸道疾病的患者。系统发育分析表明,WHCV属于β冠状病毒属(Sarbecovirus亚属),与SARS-CoV有一定的基因组和系统发育相似性1,尤其是刺突蛋白的RBD。这些与SARS的基因组和临床相似性以及在临床样本中的高丰度,为WHCV与武汉和世界各地正在爆发的呼吸系统疾病之间的关联提供了证据。虽然仅从一名患者身上分离出的病毒不足以得出它导致了这些呼吸道症状的结论,但我们的发现已经在另一项研究中在进一步的患者中得到了独立的证实29.
在蝙蝠体内发现了多种类似sars的冠状病毒,这使得人们认为这些动物是这些病毒的自然宿主22,23.虽然在中国的蝙蝠中广泛发现了与sars类似的病毒,但与sars冠状病毒相同的病毒尚未被记录下来。值得注意的是,WHCV与蝙蝠冠状病毒的亲缘关系最为密切,在nsp7和E蛋白中与蝙蝠SL-CoVZC45具有100%的氨基酸相似性(Supplementary Table)3.).因此,这些数据表明蝙蝠可能是WHCV病毒库的宿主。然而,由于该病首次报道时市场上有多种动物品种出售,需要进一步研究确定WHCV的自然宿主和任何中间宿主。
证明中增加的说明:这篇论文被接受后,ICTV将病毒命名为SARS-CoV-230;此外,世界卫生组织公布了由这种病毒引起的疾病的官方名称,即COVID-1931.
方法
数据报告
没有使用统计学方法来预先确定样本量。实验不是随机的,研究人员在实验和结果评估过程中对分配也不是盲目的。
患者信息和临床资料及样本的收集
中国武汉武汉中心医院收治的一名患者出现急性发热(温度超过37.5°C)、咳嗽和胸闷,被认为是疑似病例。在入院期间,收集BALF并在−80°C保存,直到进一步处理。从患者的临床记录中检索人口学、临床和实验室数据。本研究经中国疾病预防控制中心传染病预防控制所伦理委员会审核批准。获得患者签署的书面知情同意书。
RNA文库构建与测序
使用RNeasy Plus通用迷你试剂盒(Qiagen)按照制造商的说明从BALF样品中提取总RNA。在构建文库和测序前,使用Qbit机和安捷伦2100生物分析仪(Agilent Technologies)对RNA溶液的数量和质量进行评估。然后使用SMARTer strand Total RNA- seq kit v.2构建RNA库(豆类)。核糖体RNA耗尽是在文库建设过程中按照制造商的说明进行的。在MiniSeq平台(Illumina)上对RNA文库进行配对(150-bp reads)测序。中国上海复旦大学上海公共卫生临床中心进行了图书馆准备和测序。
病毒制剂的数据处理和鉴定
测序读取是第一个适配器和质量修剪使用Trimmomatic程序32.剩余的565928次读取使用两个Megahit(v.1.1.3)从头组装9和三一(v.2.5.1)33使用默认参数设置。Megahit共生成384,096个已组装的contigs(大小范围为200-30,474 nt),而Trinity生成1,329,960个已组装的contigs(大小范围为201-11,760 nt)e值设置为1 × 10−101 × 10−5,分别。为了识别测序数据中可能存在的病因,首先使用RSEM程序评估组装的contigs的丰度作为预期计数34在三一实现。非人类reads (23,712,657 reads),通过使用人类基因组(human release 32, GRCh38)过滤宿主reads生成。p13,从Gencode下载)由Bowtie235,用于RSEM丰度评价。
作为Megahit (30,474 nt)和Trinity (11,760 nt)生成的最长的contigs,都与蝙蝠sars样冠状病毒分离物bat SL-CoVZC45具有高度的相似性,并被发现具有高丰度(Supplementary Tables)1,2),较长的序列(30,474 nt)几乎覆盖了整个病毒基因组,用于引物设计,PCR确认和确定基因组末端。用于PCR、qPCR和RACE实验的引物见补充表8.PCR检测方法如前所述10根据制造商的说明,使用Takara SMARTer RACE 5 ' /3 '试剂盒(Takara)确定全基因组末端。随后,利用Bowtie2将所有adaptor-和quality-trim reads重新映射到WHCV的全基因组,从而确定基因组覆盖率和测序深度35和Samtools36.
采用Takara One Step PrimeScript RT-PCR试剂盒(Takara RR064A),按照制造商说明,采用实时定量RT-PCR检测BALF中WHCV的病毒载量。实时rt - PCR进行使用2.5μl核糖核酸与每个引物和4 8 pmol pmol探针在下列条件:逆转录42°C 10分钟,95°C 1分钟,其次是40 95°C的周期为1分钟15秒和60°C。反应进行,检测到ABI 7500实时PCR系统。将覆盖Taqman引物和探针区域的PCR产物克隆到pLB载体中,采用天根致死快速克隆试剂盒(Lethal Based Simple Fast Cloning Kit, TianGen)作为定量病毒载量检测标准。
病毒基因组特征及系统发育分析
对于新鉴定的病毒基因组,利用冠状病毒蛋白酶识别的切割位点的保守签名预测和注释潜在的orf,并在Lasergene软件包(v.7.1, DNAstar)中进行处理。使用MAFFT中实现的L-INS-i算法对病毒基因进行比对(v.7.407)。37.
然后使用不同冠状病毒基因数据集的核苷酸序列进行系统发育分析:(1)全基因组,(2)ORF1a, (3) ORF1b, (4) nsp5 (3CLpro), (5) RdRp (nsp12), (6) nsp13 (Hel), (7) nsp14 (ExoN), (8) nsp15 (NendoU), (9) nsp16 (O-MT), (10) spike (S)和(11)nuclear衣壳(N)。38,采用广义时间可逆替换模型和子树修剪和重接枝交换。引导支持值是从1,000个伪复制树中计算出来的。采用MEGA (v.5)确定了核苷酸替代的最佳拟合模型。39.使用Lasergene软件包(v.7.1,DNAstar)中实施的MegAlign程序计算序列之间的氨基酸同源性。
基因组重组分析
利用RDP4评估了sarbecovirus历史上潜在的重组事件19和Simplot (v.3.5.1)40.基于全基因组(核苷酸)序列,采用RDP、GENECONV、BootScan、maximum卡方、Chimera、SISCAN和3SEQ方法进行RDP4分析。假定的重组事件被鉴定为Bonferroni校正P-值截止0.01。相似性图使用Simplot来进一步描述潜在的重组事件,包括可能的断点的位置。
WHCV穗突蛋白RBD结构域分析
利用MUSCLE对WHCV、sars - cov和蝙蝠sars样cov的RBD序列进行氨基酸序列比对41.使用ProMod3在SWISS-MODEL服务器(https://swissmodel.expasy.org/).根据SWISS-MODEL模板库中包含的一阶氨基酸序列,BLAST对WHCV、Rs4874和Rp3的RBD结构域spike序列进行了搜索(最近更新,2020年1月9日;最后包括PDB版本,2020年1月3日)。使用ProMod3建立基于目标-模板对齐的模型。使用QMEAN评分函数评估全局和每残差模型的质量42显示预测蛋白质结构的PDB文件,并与SARS冠状病毒(PDB 2GHV)的峰RBD的晶体结构进行比较43SARS-CoV与人ACE2复合物(PDB 2AJF)穗状RBD的晶体结构15.
报告摘要
有关研究设计的进一步资料,请参阅《自然》研究报告摘要链接到本文。
数据可用性
本研究生成的序列读取可从NCBI序列读取档案(SRA)数据库中获得,数据库编号为BioProject登录号PRJNA603194.WHCV的全基因组序列已以登录号保存在GenBank中MN908947.
改变历史
2020年4月2日
参考文献
- 1.
等。新型冠状病毒在严重急性呼吸综合征患者中的鉴别。英语。医学博士.348, 1967 - 1976(2003)。
- 2.
主要人类传染性疾病的起源。自然447, 279 - 283(2007)。
- 3.
文图拉,C. V.,迈亚,M., Bravo-Filho, V., Góis, a . L. & Belfort, R. Jr.,巴西寨卡病毒和小头症儿童黄斑萎缩。《柳叶刀》387228(2016)。
- 4.
Shi, M.等。重新定义无脊椎RNA病毒圈。自然540, 539 - 543(2016)。
- 5.
胡,D.等。一种新型sars样冠状病毒在中国蝙蝠中的基因组特征及其传染性紧急情况。微生物感染.7, 1 - 10(2018)。
- 6.
Shi, M.等。脊椎动物RNA病毒的进化史。自然556, 197 - 202(2018)。
- 7.
Yadav,P.D.等人,《尼帕疫情期间人类和蝙蝠的尼帕病毒序列》,印度喀拉拉邦,2018年。紧急传染病.25, 1003 - 1006(2019)。
- 8.
McMullan,L.K.等人。刚果民主共和国正在进行的埃博拉病毒暴发的传染性特征,以指导应对活动:系统发育和体外分析。柳叶刀感染。说.19, 1023 - 1032(2019)。
- 9.
李丹,刘春梅,罗瑞敏,李丹。基于简化de Bruijn图的宏基因组组装的快速单节点解。生物信息学31, 1674 - 1676(2015)。
- 10.
王伟等。中国啮齿动物新型冠状病毒的发现、多样性和进化病毒学47419-27(2015)。
- 11.
胡,B.等。蝙蝠SARS相关冠状病毒的丰富基因库的发现,为SARS冠状病毒的起源提供了新的见解。公共科学图书馆Pathog.13e1006698(2017)。
- 12
林,X.-D。et al。中国蝙蝠体内冠状病毒的多样性病毒学507, 1 - 10(2017)。
- 13
Xu,L.等。中国蝙蝠中多种α-和β-冠状病毒的检测和鉴定。性研究。罪.31, 69 - 77(2016)。
- 14
Ren, W.等。SARS冠状病毒与蝙蝠源SARS样冠状病毒受体使用的差异j .性研究.82, 1899 - 1907(2008)。
- 15
李飞,李伟,李伟,李文华。SARS冠状病毒尖突受体结合域与受体的结构。科学309, 1864 - 1868(2005)。
- 16
Hulswit,R.J.G.等。人类冠状病毒OC43和HKU1与9结合-O-乙酰化唾液酸通过一个保守的受体结合位点在刺突蛋白结构域a。美国国家科学院学报。美国116, 2681 - 2690(2019)。
- 17
葛晓燕等。使用ACE2受体的蝙蝠sars样冠状病毒的分离和特征分析。自然503, 535–538 (2013).
- 18
杨晓林等。一种与严重急性呼吸综合征冠状病毒直接祖细胞密切相关的新型蝙蝠冠状病毒的分离与鉴定j .性研究.90, 3253 - 3256(2016)。
- 19.
Martin,D.P.等。RDP3:一种灵活快速的分析重组的计算机程序。生物信息学26, 2462 - 2463(2010)。
- 20.
梅纳赫里,V.D.等。一个类似SARS的蝙蝠冠状病毒群显示了人类出现的可能性。地中海Nat。.21, 1508 - 1513(2015)。
- 21.
2012年9月,从中东转移到英国的一位患者,一种新型冠状病毒引起的严重呼吸道疾病。欧元监管.17, 20290 (2012).
- 22.
一种从人类呼吸道分离出来的新病毒。Proc, Soc。实验医学杂志。地中海.121, 190 - 193(1966)。
- 23.
来自上呼吸道疾病患者的“ibv样”病毒在哺乳小鼠大脑中的生长。美国国家科学院学报。美国58, 2268 - 2273(1967)。
- 24.
范德虎,等。一种新型人类冠状病毒的鉴定。地中海Nat。.10, 368 - 373(2004)。
- 25.
哇,P. C.等人。一种新型冠状病毒HKU1的特征和全基因组序列。j .性研究.79, 884 - 895(2005)。
- 26.
李,W.等。蝙蝠是类sars冠状病毒的天然宿主。科学310, 676 - 679(2005)。
- 27.
刘淑娟等。中国马蹄蝠体内的严重急性呼吸综合征冠状病毒样病毒。美国国家科学院学报。美国102, 14040 - 14045(2005)。
- 28.
王伟等。在来自中国的亚洲家鼠中发现一种高度分化的冠状病毒,阐明了阿尔法冠状病毒的起源。j .性研究.91e00764-17(2017)。
- 29.
周鹏等。与一种可能源自蝙蝠的新型冠状病毒相关的肺炎暴发。自然https://doi.org/10.1038/s41586-020-2012-7(2020).
- 30.
Gorbalenya a E。严重急性呼吸综合征相关的冠状病毒——物种及其病毒,冠状病毒研究小组的声明。在预印本bioRxivhttps://doi.org/10.1101/2020.02.07.93786(2020).
- 31.
谁。世卫组织总干事2020年2月11日在2019-nCoV疫情媒体通报会上的讲话https://www.who.int/dg/speeches/detail/who-director-general-s-remarks-at-the-media-briefing-on-2019-ncov-on-11-february-2020(WHO, 2020年2月11日)。
- 32.
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学30, 2114 - 2120(2014)。
- 33.
等。没有参考基因组的RNA-seq数据的全长度转录组组装。Nat。.29, 644–652 (2011).
- 34.
李斌,李志强,李志强,李志强。基于不确定读图的RNA-seq基因表达估计。生物信息学26, 493 - 500(2010)。
- 35.
Langmead, B. & Salzberg, s.l., Fast gap -read alignment with Bowtie 2。Nat方法。9, 357 - 359(2012)。
- 36
李,H.等。序列对齐/映射格式和SAMtools。生物信息学25, 2078 - 2079(2009)。
- 37
kaoh, K. & Standley, D. M. MAFFT多序列比对软件版本7:性能和可用性的改进。摩尔。杂志。另一个星球.30, 772 - 780(2013)。
- 38
金登等。估计最大似然系统发育的新算法和方法:评估PhyML 3.0的性能。系统。医学杂志.59, 307 - 321(2010)。
- 39
田村等。利用最大似然、进化距离和最大简约法进行分子进化遗传学分析。摩尔。杂志。另一个星球.28, 2731–2739 (2011).
- 40.
Lole, K. S.等。来自印度c亚型感染血清转化体的全长人类免疫缺陷病毒1型基因组,有亚型间重组的证据。j .性研究.73, 152 - 160(1999)。
- 41.
Edgar,R.C.MUSCLE:具有高精度和高通量的多序列比对。核酸Res.32, 1792 - 1797(2004)。
- 42.
Waterhouse,A.等人,《瑞士模式:蛋白质结构和复合物的同源性建模》。核酸Res.46W296-W303(2018)。
- 43.
等。人抗严重急性呼吸综合征刺突蛋白抗体80R中和的结构基础。生物。化学.281, 34610 - 34616(2006)。
确认
本研究由国家基础资源调查专项(no . 2019FY101500)和国家自然科学基金(no . 81861138003和no . 31930001)资助。E.C.H.由ARC澳大利亚获奖者奖学金(FL170100022)资助。
作者信息
从属关系
贡献
y - z - z构思并设计了这项研究。沈志、杨华、张文涛和李玉玲分别进行了临床工作和标本采集。B.Y.和J.-H.T.进行了流行病学调查和样本收集。Z.-G.S F.W。,L.X Y.-Y.P, Y.-L.Z, F.-H.D, Y.L J.-J.Z.和Q.-M.W.进行了实验。Y.-M.C砂石,F.W。E.C.H. Y.-Z.Z.分析数据。y - z.z, E.C.H.和F.W.在所有作者的参与下撰写了论文。y - z - z领导了这项研究。
相应的作者
道德宣言
相互竞争的利益
两位作者宣称没有相互竞争的利益。
额外的信息
同行评议信息自然感谢Nicholas Loman和其他匿名审稿人对这项工作的同行评议做出的贡献。
出版商的注意施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
扩展数据图表
图1患者胸片。
一个- - - - - -d入院当天(发病后第6天)进行胸部ct扫描。双侧局灶性实变、大叶性实变、斑片状实变清晰,下肺尤为明显。e入院后第5天(发病后第11天)行胸片。双侧弥漫斑片状模糊阴影。
图2 BALF标本中real-time RT-PCR未检测到其他呼吸道病原体。
一个- - - - - -e,化验该BALF样本是否含有甲型流感病毒(一个)、维多利亚系B型流感病毒(b)、乙型流感病毒山形系(c)、人类腺病毒(d),衣原体肺炎(e).样本1为患者BALF样本,以水作为阴性(NEG)对照和阳性(POS)对照样本,包括覆盖a型流感病毒、B型流感病毒Victoria和Yamagata系、人腺病毒和a型流感病毒Taqman引物和探针区域的质粒衣原体肺炎.
图3 WHCV基因组读计数图谱。
直方图显示了WHCV基因组每个碱基的覆盖深度。WHCV基因组的平均测序深度为604.21 nt。
图4实时RT-PCR对临床样本WHCV的定量分析。
一个、WHCV引物特异性评价。检测样本包括至少对下列一种病毒呈阳性的临床样本:甲型流感病毒(09H1N1和H3N2)、乙型流感病毒、人腺病毒、呼吸道合胞病毒、鼻病毒、副流感病毒1-4型、人肉毒杆菌病毒、人偏肺病毒、冠状病毒OC43、冠状病毒NL63、冠状病毒229E和冠状病毒HKU1。只有WHCV标准质粒(WHCV在pLB载体中为15,704-16,846 bp)扩增呈阳性(棕色曲线)。b, WHCV DNA标准扩增曲线。从左到右,DNA浓度为1.8 × 108, 1.8 × 107, 1.8 × 106, 1.8 × 105, 1.8 × 1041.8 × 103..c的线性拟合曲线Ct值到WHCV DNA标准浓度。d,实时RT-PCR定量BALF标本中WHCV。阳性对照为WHCV DNA标准品(POS),阴性对照为水(NEG)和空白。BALF样品的放大曲线为绿色。
图5全基因组核苷酸序列的最大似然系统发育树年代和NWHCV和相关冠状病毒的基因。
分支上面或下面的数字(>70)表示百分比引导值。这些树的根在中间,只是为了清晰。比例尺表示每个站点的替换数量。
图6拟南芥核苷酸序列的最大似然系统发育树3氯,RdRp,冥界,外显子,嫩豆和O-MTWHCV和相关冠状病毒的基因。
分支上面或下面的数字(>70)表示百分比引导值。这些树的根在中间,只是为了清晰。比例尺表示每个站点的替换数量。
图7 WHCV冠状病毒刺突蛋白RBD分析。
一个, sars样冠状病毒RBD序列的氨基酸序列比对。三种蝙蝠sars样冠状病毒可以有效地利用人类ACE2作为受体,它们的RBD序列大小与SARS-CoV相似。WHCV包含一个Val470插入。与人类ACE2相互作用的关键氨基酸残基用橙色方块表示。相比之下,5种蝙蝠sars样冠状病毒,包括Rp3,此前已发现它不会与ACE2结合14-与SARS-CoV.11相比,在两个基序(氨基酸433-437和460-472,用红色方框突出显示)中有氨基酸缺失b这两个基元(433-437和460-472氨基酸)以红色显示,是SARS-CoV刺突蛋白RBD与人类ACE2受体(PDB 2AJF)复合物的晶体结构。人类ACE2为蓝色,sars冠状病毒刺突蛋白RBD为绿色。标记人类ACE2与sars冠状病毒刺突蛋白RBD相互作用的重要残基。c,在SWISS-MODEL服务器上使用Promot3基于目标-模板比对预测WHCV棘突蛋白RBD的蛋白质结构。dsars样冠状病毒Rs4874刺突蛋白RBD结构预测esars样冠状病毒Rp3刺突蛋白RBD结构预测f, SARS-CoV刺突蛋白RBD的晶体结构(绿色)(PDB 2GHV)。与sars冠状病毒刺突蛋白433-437和460-472氨基酸相似的基序用红色表示。
图8穗蛋白n端氨基酸序列比较。
比较WHCV、牛冠状病毒(BCoV)、小鼠肝炎病毒(MHV)、人冠状病毒(HCoV OC43和HKU1)与不能结合唾液酸的sars冠状病毒(SZ3、WH20、BJ0和Tor2)刺突蛋白n端结构域的氨基酸序列。的关键残基16唾液酸与BCoV、MHV、HCoV OC43和HKU1的结合用橙色方格标出。
扩展数据图9
WHCV中的重组事件。WHCV、sars样冠状病毒和蝙蝠sars样冠状病毒的序列相似性图揭示了假定的重组事件。
权利和权限
188滚球软件本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于这篇文章
引用这篇文章
吴凡,赵淑芳,余波。et al。一种与中国人类呼吸道疾病相关的新型冠状病毒。自然579年,265 - 269(2020)。https://doi.org/10.1038/s41586-020-2008-3
收到了:
接受:
发表:
发行日期:
进一步的阅读
COVID - 19大流行封锁措施对外科急诊的影响:从希腊一家三级医院吸取的经验教训
世界急诊外科杂志(2021)
2019冠状病毒病扩散的社会空间决定因素:全球化的影响、定居特征和人口
全球化和健康(2021)
有或无急性后遗症的COVID-19幸存者的纵向SARS-CoV-2生物库
BMC传染病(2021)
新型SARS-CoV-2多表位疫苗的疫苗组学研究
BMC免疫学(2021)
SARS-CoV-2:高分辨率显微镜对感染人鼻咽组织的初步研究
病毒学杂志(2021)