文摘gydF4y2Ba
RNA的丰度是一个强大的指标单个细胞的状态。单细胞测序可以揭示RNA RNA丰度定量准确性高、灵敏度和吞吐量gydF4y2Ba1gydF4y2Ba。然而,这种方法只捕捉一个静态快照时间点,构成挑战的分析时间分辨现象如胚胎发生或组织再生。在这里,我们表明,RNA基因表达的流通时间导数国家可以直接估计区分unspliced和拼接mrna共同点单细胞RNA序列协议。RNA速度是一个高维向量预测未来状态的时间尺度上的单个细胞。我们验证其准确性神经嵴血统,证明它的使用在多个数据集出版和技术平台,揭示发展的分支家族树鼠海马,并检查转录的动力学人类胚胎的大脑。我们预计RNA速度极大的援助发展血统和细胞动力学的分析,特别是在人类。gydF4y2Ba
这是一个预览的订阅内容gydF4y2Ba
访问选项gydF4y2Ba
订阅自然+gydF4y2Ba
得到直接在线访问整个自然家庭50 +的期刊gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅杂志gydF4y2Ba
得到完整的期刊访问1年gydF4y2Ba
199.00美元gydF4y2Ba
只有3.90美元的问题gydF4y2Ba
所有价格是净价格。gydF4y2Ba
增值税将被添加后在付款。gydF4y2Ba
税计算期间将完成付款。gydF4y2Ba
买条gydF4y2Ba
时间有限或全文访问ReadCube。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格是净价格。gydF4y2Ba

引用gydF4y2Ba
Linnarsson, s &摄影师,s .答:(单细胞基因组学时代的到来。gydF4y2Ba基因组医学杂志gydF4y2Ba。gydF4y2Ba17gydF4y2Ba97 (2016)。gydF4y2Ba
蔡塞尔,a . et al .耦合pre-mRNA和mRNA动态发布操作策略潜在的转录对刺激的反应。gydF4y2Ba摩尔。系统。医学杂志gydF4y2Ba。gydF4y2Ba7gydF4y2Ba529 (2011)。gydF4y2Ba
灰色,j . m . et al . SnapShot-Seq:方法提取全基因组,体内总RNA信使RNA动力从一个样本。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Bae89673 (2014)。gydF4y2Ba
Gaidatzis D。汉堡,L。,Florescu, M. & Stadler, M. B. Analysis of intronic and exonic reads in RNA-seq data characterizes transcriptional and post-transcriptional regulation.Nat。gydF4y2Ba。gydF4y2Ba33gydF4y2Ba,722 - 729 (2015)。gydF4y2Ba
Picelli, s . et al . Smart-seq2敏感全身的单个细胞的转录组分析。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba,1096 - 1098 (2013)。gydF4y2Ba
伊斯兰教,美国et al .定量单细胞RNA-seq具有独特的分子标识符。gydF4y2BaNat方法。gydF4y2Ba11gydF4y2Ba,163 - 166 (2013)。gydF4y2Ba
克莱恩,a . m . m . et al .液滴条码为单细胞转录组应用于胚胎干细胞。gydF4y2Ba细胞gydF4y2Ba161年gydF4y2Ba,1187 - 1201 (2015)。gydF4y2Ba
郑,g . x y . et al .大规模并行数字单个细胞的转录分析。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba8gydF4y2Ba14049 (2017)。gydF4y2Ba
Schwalb, b . et al . TT-seq地图人类短暂的转录组。gydF4y2Ba科学gydF4y2Ba352年gydF4y2Ba,1225 - 1228 (2017)。gydF4y2Ba
伊斯兰教,美国et al .表征的单细胞转录景观高度多元化RNA-seq。gydF4y2Ba基因组ResgydF4y2Ba。gydF4y2Ba21gydF4y2Ba,1160 - 1167 (2011)。gydF4y2Ba
横膈的缪里斯财团、地震s R。Wyss-Coray, T。,&Darmanis, S. Single-cell transcriptomic characterization of 20 organs and tissues from individual mice creates a Tabula Muris. Preprint athttps://biorxiv.org/content/early/2018/03/29/237446gydF4y2Ba(2018)。gydF4y2Ba
Vollmers, c . et al .生理振荡的蛋白质编码和监管rna在一个高度动态的哺乳动物肝脏表观基因组。gydF4y2Ba细胞金属底座gydF4y2Ba。gydF4y2Ba16gydF4y2Ba,833 - 845 (2012)。gydF4y2Ba
可•福尔兰正,a . et al .多功能外围神经胶质细胞产生的神经内分泌细胞肾上腺髓质。gydF4y2Ba科学gydF4y2Ba357年gydF4y2Baeaal3753 (2017)。gydF4y2Ba
克里斯坦,a . & Alvarez-Buylla a .胶质胚胎和成年神经干细胞的性质。gydF4y2Ba为基础。启>gydF4y2Ba。gydF4y2Ba32gydF4y2Ba,149 - 184 (2009)。gydF4y2Ba
Malatesta, p . et al .神经元或神经胶质的后代:区域差异在径向神经胶细胞的命运。gydF4y2Ba神经元gydF4y2Ba37gydF4y2Ba,751 - 764 (2003)。gydF4y2Ba
约翰斯顿(r . j . & Desplan c .随机细胞命运的机制规范,产生随机或健壮的结果。gydF4y2Ba为基础。启细胞Dev。杂志gydF4y2Ba。gydF4y2Ba26gydF4y2Ba,689 - 719 (2010)。gydF4y2Ba
Iwano, T。,米一个年代ud一个,A., Kiyonari, H., Enomoto, H. & Matsuzaki, F. Prox1 postmitotically defines dentate gyrus cells by specifying granule cell identity over CA3 pyramidal cell fate in the hippocampus.发展gydF4y2Ba139年gydF4y2Ba,3051 - 3062 (2012)。gydF4y2Ba
布特就,m . et al . Prox1 postmitotically定义齿状回细胞通过指定颗粒细胞的身份在海马CA3锥体细胞的命运。gydF4y2Ba科学gydF4y2Ba360年gydF4y2Baeaaq1723 (2018)。gydF4y2Ba
Petukhov诉等人下降:管道分子数的准确估计droplet-based单细胞RNA-seq实验。gydF4y2Ba基因组医学杂志gydF4y2Ba。gydF4y2Ba19gydF4y2Ba78 (2018)。gydF4y2Ba
蔡塞尔,et al。分子老鼠神经系统的体系结构。预印在gydF4y2Bahttps://biorxiv.org/content/early/2018/04/06/294918gydF4y2Ba(2018)。gydF4y2Ba
Hrvatin, s . et al .单细胞分析经验依赖转录组的国家鼠标视觉皮层。gydF4y2BaNat。>gydF4y2Ba。gydF4y2Ba21gydF4y2Ba,120 - 129 (2018)。gydF4y2Ba
哈伯,A . l . et al .单细胞小肠上皮细胞的调查。gydF4y2Ba自然gydF4y2Ba551年gydF4y2Ba,333 - 339 (2017)。gydF4y2Ba
Hochgerner, H。蔡塞尔,。,Lönnerberg, P. & Linnarsson, S. Conserved properties of dentate gyrus neurogenesis across postnatal development revealed by single-cell RNA sequencing.Nat。>。gydF4y2Ba21gydF4y2Ba,290 - 299 (2018)。gydF4y2Ba
Lein e . s . et al .全基因组基因表达图谱的成年老鼠的大脑。gydF4y2Ba自然gydF4y2Ba445年gydF4y2Ba,168 - 176 (2007)。gydF4y2Ba
确认gydF4y2Ba
支持的工作报告是瑞典战略研究基金会(rif14 - 0057和sb16 - 0065),克努特和爱丽丝•瓦伦堡基金会(2015.0041),粉嫩一步裙佩尔森基金会(HumDevCellAtlas)和威康信托(108726 / Z / 15 / Z) S.L.;创新医学中心(cim) K.L.和电脑;瑞典研究理事会,居里夫人集成格兰特EPIOPC, 333713年,欧洲研究理事会反射幻灯机,681893年,瑞典的大脑基础,明围刘修复医学中心Cancerfonden和G.C.-B卡罗林斯卡医学院。P.V.K.支持的NIH R01HL131768 NHLBI和职业(NSF - 14 - 532)从NSF奖。gydF4y2Ba
审核人信息gydF4y2Ba
自然gydF4y2Ba由于a . Klein r . Satija m .施,另一个匿名的评论家(s)为他们的贡献的同行评审工作。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
RNA速度和P.V.K. S.L.构思的概念表明,RNA速度可以通过分析发现unspliced记录在单个细胞。P.V.K. S.L.设计和监督学习。P.V.K.,S.L.,G.L.M. and R.S. developed the analytical framework, analysed data, made figures and drafted the manuscript, with contributions from all co-authors. P.V.K., G.L.M., R.S. and P.L. implemented the software, with assistance from V.P. and J.F. Z.L. examined RNA degradation signals. A.Z. and H.H. performed the mouse hippocampus experiment. P.C. supervised and K.L. and H.H. performed metabolic labelling. M.E.K. and I.A. performed validations of chromaffin differentiation rate. E.B. and L.E.B. performed and analysed the fluorescent in situ hybridization experiment on tissue dissected by X.H. E.S. and R.B. provided human embryonic brain tissue. D.v.B. performed the human forebrain single-cell RNA-seq experiment under supervision of G.C.-B. J.G. assisted with measurement and interpretation of mouse bone marrow. The paper was read and approved by all co-authors.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
扩展数据数据和表gydF4y2Ba
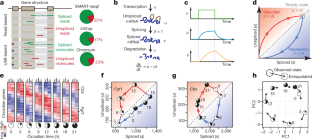
扩展数据图1的大部分intronic读取出现由于内部启动从稳定的位置。gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba,读的例子密度intronic玻利亚和律polyT序列。浏览器截图显示读取的密度从10 x铬小鼠海马数据集(上跟踪每个面板),小鼠骨髓inDrop数据集(第二个跟踪从顶部)和肾上腺髓质分化评估使用SMART-seq2(第三轨)。底部两个跟踪显示基因注释和立场的聚(或polyT序列的长度至少15 bp与一个允许不匹配。聚/ polyT盒子颜色是蓝色的,如果在整合方向延伸到底层的基因的转录(也就是说,将导致新兴的RNA分子聚序列转录),或红色,如果他们在不整合面向位置(也就是说,将导致polyT RNA序列)。3′-end-based 10 x铬和inDrop协议显示离散山峰下游的聚(启动网站,10倍的数据也显示峰值上游的polyT网站。SMART-seq2协议显示更加扩散高峰,预计从全身的净化过程所使用的协议。gydF4y2BaegydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba密度、平均阅读资料整合和不和谐的内部启动网站。情节展现观察/预期intronic读周围密度(A)gydF4y2Ba15gydF4y2Ba或(T)gydF4y2Ba15gydF4y2Ba与1允许错配序列()内intronic地区。的gydF4y2BaxgydF4y2Ba轴显示位置相对于图案位置(完全),在基因组参考方向。粗线显示全基因组(平均削减染色体之间的两个极端值为每个职位)。单个染色体显示为半透明的平均线。gydF4y2BaegydF4y2Ba,概要文件的鼠标海马10倍的数据集(铬gydF4y2BangydF4y2Ba= 18213)。gydF4y2BafgydF4y2Ba,对人类前脑10 x档案数据(gydF4y2BangydF4y2Ba= 1720)。gydF4y2BaggydF4y2Ba肾上腺髓质分化的概要文件数据测量使用SMART-seq2 (gydF4y2BangydF4y2Ba= 385)。gydF4y2BahgydF4y2Ba,老鼠骨髓概要数据测量使用inDrop (gydF4y2BangydF4y2Ba= 3018)。每个图的左上角显示所有intronic读取的数量(即基因内的下降,但不接触一个外显子),属于内部启动站点周围的250个基点(1500个基点是用于SMART-seq2数据集)。在10 x数据,整合内部启动站点产生更强的信号,它们在基因组中的频率低于不和谐的网站,这样整体不和谐的网站占intronic信号稍高的分数。相比之下,inDrop数据集似乎不和谐的启动非常有限。gydF4y2Ba
扩展数据图2的估计时间在人类细胞中RNA代谢特征。gydF4y2Ba
一个gydF4y2Ba在人类细胞中,代谢标签的设计实验。HEK293细胞暴露于4-thiouridine su(4) 5、15或30分钟,贴上分数是分离和分析。也没有下拉控制分析,代表了平衡态(∞)。gydF4y2BabgydF4y2Ba,预计的概要的丰富和分数标记拼接和unspliced RNA分子。gydF4y2BacgydF4y2Ba观察到的基因集群的动态资料,产生两组:绝大多数(83.4%)和增加的期望一致标签;和一个较小的比例(16.6%)的不和谐的基因。酒吧表明s.e.m。gydF4y2BangydF4y2Ba基因gydF4y2Ba= 998,gydF4y2BangydF4y2Ba技术gydF4y2Ba= 2,gydF4y2BangydF4y2Ba生物gydF4y2Ba= 2。gydF4y2BadgydF4y2Ba、曲线显示最大似然适合数据,基于解析解的步骤增加转录率。健康的收益率的值gydF4y2BaβgydF4y2Ba和gydF4y2BaγgydF4y2Ba,时间常数的特征gydF4y2BaτgydF4y2Ba,定义为所需的时间达到1 - 1 / e≈渐近值的63.2%。gydF4y2BaegydF4y2Ba的分布gydF4y2BaτgydF4y2Ba值。gydF4y2BafgydF4y2Ba的联合分布gydF4y2BaβgydF4y2Ba和gydF4y2BaγgydF4y2Ba参数(gydF4y2BangydF4y2Ba= 832)。gydF4y2Ba
扩展数据图3退化率守恒的广泛的终末分化细胞类型。gydF4y2Ba
保护的RNA降解率在各种不同的细胞在成年小鼠(横膈缪里斯数据集)。gydF4y2Ba一个gydF4y2Ba分布的相关性的基因拼接和unspliced分子数量在所有细胞类型(gydF4y2BangydF4y2Ba基因gydF4y2Ba= 8385)。gydF4y2BabgydF4y2Ba,传说列举的组织和细胞类注释牌缪里斯联盟(gydF4y2BangydF4y2Ba= 48)。功能、发育或表型相关类的相似的颜色来辅助解释下面的情节。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba代表选择与高度相关的基因(gydF4y2BaρgydF4y2Ba> 0.9)(gydF4y2BacgydF4y2Ba)和典型相关(0.9 >gydF4y2BaρgydF4y2Ba> 0.4)(gydF4y2BadgydF4y2Ba)。gydF4y2BaγgydF4y2Ba被强大的线性回归估计(RANSAC)。gydF4y2BaegydF4y2Ba情节显示选择的基因显示两个明显不同的降解率(双基因gydF4y2BaγgydF4y2Ba占总数的10.8%)。两个不同的值gydF4y2BaγgydF4y2Ba被混合回归模型估计。gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba,两个基因的多个值的例子gydF4y2BaγgydF4y2Ba在不同的细胞类型是解释为可变剪接。gydF4y2Ba
扩展数据图4基于结构的速度估计。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,只观察到的基因以外的稳定状态,如基因调节肾上腺髓质分化晚期(gydF4y2Ba一个gydF4y2Ba)或在许旺细胞前体的早期表达下调(gydF4y2BabgydF4y2Ba),gene-relativegydF4y2BaγgydF4y2Ba可能会偏离其稳态值。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba正确的影响,基于结构的gydF4y2BaγgydF4y2Ba适合将首先预测gydF4y2BaγgydF4y2Ba对于每一个基因根据其结构参数,然后再使用gydF4y2BakgydF4y2Ba最相关的基因数据集进行调整gydF4y2Ba米gydF4y2Ba值(gydF4y2Ba\ (M = {\ mathrm{日志}}_{2}({你}_ {{\ rm {o}}} /{你}_ {{\ rm s} {} {\ rm{年代}}})\)gydF4y2Ba,在那里gydF4y2BaugydF4y2Ba党卫军gydF4y2Ba是拼接的unspliced数量预测计算稳态下,然后呢gydF4y2BaugydF4y2BaogydF4y2Ba使用健壮的意思是,观察unspliced计数)和重新评估吗gydF4y2BaγgydF4y2Ba。gydF4y2BaegydF4y2Ba散点图比较gene-relative和基于结构gydF4y2BaγgydF4y2Ba估计,用彩色的圆圈强调gydF4y2BaγgydF4y2Ba调整在scp早期基因表达下调(蓝色)和调节在嗜铬细胞(绿色)。自然对数尺度所示的值。gydF4y2BafgydF4y2Ba- - - - - -gydF4y2Ba我gydF4y2Ba嗜铬细胞表达速度E12.5数据集,基于小gydF4y2BaγgydF4y2Ba估计,显示在第一个五个人电脑。gydF4y2Ba
扩展数据图5 RNA inDrop数据集的速度分析:视觉皮质锥体神经元的刺激响应和嗜中性粒细胞分化。gydF4y2Ba
一个gydF4y2Ba,模型的简化插图的视觉皮层锥体神经元的激活后暴露在光刺激。gydF4y2BabgydF4y2Ba、速度估计投影到二维主成分分析数据集的情节(gydF4y2BangydF4y2Ba= 952)。gydF4y2BacgydF4y2Ba如果细胞的转移概率,平均(顶部),细胞刺激1 h(中)和细胞刺激4 h(底部)。如果细胞主要是静止的,只有少数细胞显示激活早期反应基因的倾向(可能是由于分离过程)。细胞刺激1 h被立即早期基因表达特征和高速度响应基因,年末他们,因此,过渡到一个国家更类似于一个观察四健会激活时间点。4 h后刺激细胞似乎恢复状态与如果样本(底部)。gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba前面板,早期阶段的画像(gydF4y2BadgydF4y2Ba)和后期(gydF4y2BaegydF4y2Ba)反应的基因。底部面板、小提琴情节展示在细胞群表达分布在每一个时间点(小提琴的左半部分)和外推未来使用速度(对小提琴的一半)。情节,单个细胞的转型是由线条表示连接两部分的小提琴和彩色的每个基因的速度的迹象。gydF4y2BafgydF4y2Ba电网可视化显示,细胞表达速度估计inDrop老鼠骨髓数据集gydF4y2BatgydF4y2Ba新力情节(gydF4y2BangydF4y2Ba= 3018)。gydF4y2BaggydF4y2Ba,主要的细胞数量是基于人工注释标记。速度流gydF4y2Ba一个gydF4y2Ba捕捉嗜中性粒细胞的成熟,从右边的细胞分裂开始,一直到gydF4y2BaIl1bgydF4y2Ba激活在左边。对五个标记基因表达谱如下所示。gydF4y2BahgydF4y2Ba,故事情节说明gene-relative基因模型适合几个例子。对于每一个基因,第一列显示拼接分子在不同的细胞计数。第二列显示unspliced分子数量。第三列显示了基因的相图(unspliced与拼接的依赖),由此产生的gydF4y2BaγgydF4y2Bafit(虚线红线),使用极端分位数方法决定的。每个点对应于一个细胞中,根据集群彩色标签所示gydF4y2BaggydF4y2Ba。最后一列显示unspliced计数信号基于估计的剩余gydF4y2BaγgydF4y2Ba健康,积极的残差表示预计upregulation,和消极的剩余工资的差别表明预期对这些基因。gydF4y2Ba
动态扩展数据图6成熟的肠上皮细胞在肠道内稳态。gydF4y2Ba
一个gydF4y2Ba,速度场投影到二维的gydF4y2BatgydF4y2Ba新力阴谋。集群是标签和颜色在最初的出版物gydF4y2Ba11gydF4y2Ba为了便于比较(gydF4y2BangydF4y2Ba= 2683)。速度分析揭示了一个过渡与远端和近端肠上皮细胞的成熟。没有一致的速度在歧管的一部分被干细胞和交通放大(TA)细胞,这表明干细胞动力学更难以捕捉的速度较慢,或更多的随机性质。运输的小速度放大细胞可能受细胞周期。gydF4y2BabgydF4y2Ba细胞周期基因的选择,从分析绘制在被移除gydF4y2BatgydF4y2Ba新力。尽管取消注释的基因是细胞周期基因,我们还观察到细胞周期重要的隔离,说明很难理清细胞周期阶段的细胞状态。gydF4y2BacgydF4y2Ba的选择阶段肖像显示基因潜在的观察到的速度场。标记的内分泌,酒杯和簇细胞显示没有检测到速度。速度对和从干细胞状态检测为一组有限的基因(如干细胞标记gydF4y2BaLgr5就gydF4y2Ba),然而,在全基因组水平上,这个过程可能是困惑的确切动态高与细胞周期相关。gydF4y2Ba
扩展数据图7 RNA速度揭示分化和少突胶质细胞髓鞘形成的动力。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BatgydF4y2Ba新力投影显示少突细胞谱系分化和髓鞘形成过程的景观在后脑(桥)的青少年(P20)小鼠(gydF4y2BangydF4y2Ba= 6307)。速度场的动力学表达式反映了初始分化波和下面的表达变化与髓鞘形成的过程。细胞集群由拟时间如彩色gydF4y2BacgydF4y2Ba为了方便解释。gydF4y2BabgydF4y2Ba,表达的具有里程碑意义的基因分化过程的模式。gydF4y2BaPdgfragydF4y2Ba是规范化的标志少突细胞前体(信息公开化,gydF4y2BaNeu4gydF4y2Ba标志着承诺少突细胞前体(警察),gydF4y2BaTmem2gydF4y2Ba浓缩在新成立的少突胶质细胞(NFOLs)的表达gydF4y2BaMoggydF4y2Ba髓鞘形成过程的初调节在myelin-forming寡树突胶质细胞(MFOLs)。gydF4y2BacgydF4y2Ba的选择阶段肖像底层所示的速度场gydF4y2Ba一个gydF4y2Ba。gydF4y2BadgydF4y2Ba,gydF4y2BatgydF4y2Ba新力预测和速度矢量场相同的数据集,但分析使用一个更天真的特征选择,保留其他轴的变化在少突细胞成熟(解剖性和的一天)。注意,尽管人口的分离gydF4y2BaXistgydF4y2Ba+gydF4y2Ba和gydF4y2BaXistgydF4y2Ba−gydF4y2Ba正确,速度场从祖细胞捕捉发展新成立的少突胶质细胞在两条平行的铁轨。gydF4y2BaegydF4y2Ba的表达水平gydF4y2BaXistgydF4y2Ba显示,大部分的额外的变化是由动物的性别。gydF4y2BafgydF4y2Ba实验,细胞彩色的那一天。gydF4y2Ba
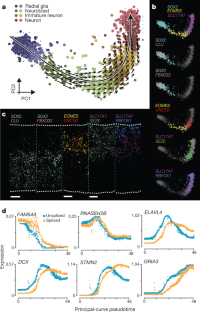
扩展数据的速度预测图8协议表达衍生品。gydF4y2Ba
一个gydF4y2Ba、成熟的颗粒在小鼠海马神经元近似数据集拟时间(估计主要曲线)。gydF4y2BabgydF4y2Ba,一对基因(行),情节显示unspliced和拼接基因表达谱拟时间(左面板),观察到的基因表达的经验估计平滑拟时间导数和速度估计RNA(中间板),以及拼接和unspliced表达之间的关系(右面板)。速度估计的两个选择的基因是高度相关的实证观察导数,指示准确的速度估计。gydF4y2BacgydF4y2Ba,绝大多数(75%)的不同的基因调控在拟时间轨迹显示正相关的经验表达的导数。这类基因的分布划分根据三类trajectory-associated基因所示gydF4y2BadgydF4y2Ba。相比之下,速度估计没有差异表达基因在拟时间轨迹并没有显示出这种相关性(灰色)。将信息重新使用基因基因导入速度估计gydF4y2BakgydF4y2Ba神经网络聚类(见补充说明gydF4y2Ba1gydF4y2Ba)可以显著提高速度预测的准确性(下图)。gydF4y2BadgydF4y2BaTrajectory-associated基因分为早期,瞬态和后期,根据峰值时间表达式。gydF4y2BaxgydF4y2Ba由拟时间轴,细胞有序;gydF4y2BaygydF4y2Ba轴,基因下令峰值时间表达式。gydF4y2BaegydF4y2Ba,好的关联的基因拼接的表达模式gydF4y2BaPtprggydF4y2Ba,还显示高相关性的速度估计gydF4y2BaPtprggydF4y2Ba。评估的一致性程度的速度重新基因,我们引入了测量速度的协调对于一个给定的基因,作为区别的平均速度的相关性估计所有基因的基因和速度估计粘住。显示的两个量相比gydF4y2BaPtprggydF4y2Ba虚线竖线:灰色,代表速度相关的基因;红色,与前重新平均速度相关的基因。速度的协调提供了一个客观的衡量质量的速度估计。gydF4y2BafgydF4y2Ba的速度重新基因相关。基因的分布速度协调值显示了基因重新基因(即基因密切相关的基因邻国的拼接表达模式,绿色),以及没有足够的基因重新基因(没有邻居,灰色)。gydF4y2BaggydF4y2Ba,重新基因有很高的速度协调往往和经验衍生品有高度的相关性。斯皮尔曼相关系数。gydF4y2BahgydF4y2Ba- - - - - -gydF4y2BakgydF4y2Ba在锥体神经元的成熟速度性能(gydF4y2BahgydF4y2Ba)。差异表达基因在成熟与经验导数(高速度的相关性gydF4y2Ba我gydF4y2Ba),重新基因倾向于有相关速度估计(gydF4y2BajgydF4y2Ba)和速度协调的程度与与经验的相关衍生品(gydF4y2BakgydF4y2Ba)。gydF4y2BalgydF4y2Ba,gydF4y2Ba米gydF4y2Ba在肾上腺髓质分化速度性能。gydF4y2BapgydF4y2Ba- - - - - -gydF4y2Ba年代gydF4y2Ba在成熟的少突胶质细胞,速度性能。许多顶尖的粘住速度相关的基因分析:200 (gydF4y2BaggydF4y2Ba),150 (gydF4y2BakgydF4y2Ba,gydF4y2BaogydF4y2Ba,gydF4y2Ba年代gydF4y2Ba)。gydF4y2Ba
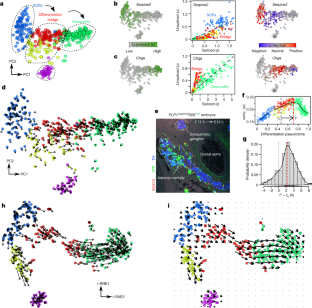
扩展数据图9个分支发展海马体的发展轨迹。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BatgydF4y2Ba新力的情节发展的齿状回的数据集。细胞由集群身份,彩色标签显示的主要细胞类型。gydF4y2BabgydF4y2Ba,表达的径向神经胶质和星形胶质细胞标记gydF4y2BaHes1gydF4y2Ba和细胞周期基因gydF4y2BaTop2agydF4y2Ba和gydF4y2BaCdk1gydF4y2Ba上显示gydF4y2BatgydF4y2Ba新力阴谋。gydF4y2BacgydF4y2Ba标记基因的不同地区的海马体(原位杂交图像从艾伦老鼠大脑图集gydF4y2Ba24gydF4y2Ba显示突出表达信号在不同的四肢的分支情节。规模酒吧、0.5毫米。gydF4y2Ba
扩展数据图10单细胞速度估计单个细胞的胚胎海马数据集。gydF4y2Ba
一个gydF4y2Ba投射到,箭头表示推测的状态gydF4y2BatgydF4y2Ba新力歧管的阴谋。gydF4y2BabgydF4y2Ba,选择平衡的肖像画和符合阶段斜率(gydF4y2BaγgydF4y2Ba)发展中细胞胚胎海马数据集。对于每一个基因,第一列显示spliced-unspliced相图。虚线显示gydF4y2BaγgydF4y2Ba健康。第二列显示了残差的大小(也就是说,观察和预期unspliced丰度之间的区别,密切跟踪速度)对多个基因参与不同的神经血统的发展。第三列显示了拼接的观察表达谱分子。gydF4y2Ba
补充信息gydF4y2Ba
补充注1gydF4y2Ba
理论和计算方法:本补充报告中描述的理论公式采用转录模型、基本假设、数学和计算估计过程的细节,并提供额外的相关细节处理个人数据集。gydF4y2Ba
补充注2gydF4y2Ba
考虑对准确测定和可视化的RNA速度:这个补充报告说明了模拟底层模型在不同的场景下,细节各种方式速度可以估计和可视化,并讨论了已知的限制的过程。它分为11个部分,包括补充数据1 - 20。gydF4y2Ba
权利和权限gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
没有,G。,Soldatov, R., Zeisel, A.et al。gydF4y2Ba单个细胞的RNA速度。gydF4y2Ba自然gydF4y2Ba560年,gydF4y2Ba494 - 498 (2018)。https://doi.org/10.1038/s41586 - 018 - 0414 - 6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586 - 018 - 0414 - 6gydF4y2Ba
进一步的阅读gydF4y2Ba
单细胞测序数据的生物信息学分析指南在阿尔茨海默氏症:评论、建议、实施和应用gydF4y2Ba
分子神经退化gydF4y2Ba(2022)gydF4y2Ba
DevKidCC允许强大的分类和肾脏瀑样数据集的直接比较gydF4y2Ba
基因组医学gydF4y2Ba(2022)gydF4y2Ba
分子生物学组织的空间组件gydF4y2Ba
自然生物技术gydF4y2Ba(2022)gydF4y2Ba
单细胞转录组的糖尿病足溃疡gydF4y2Ba
自然通讯gydF4y2Ba(2022)gydF4y2Ba
通过RSPO2监管途径抑制脂肪形成的识别gydF4y2Ba
自然的新陈代谢gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba
