要点
疾病的表型很少是单个效应基因产物异常的结果,而是反映了在一个复杂的网络中相互作用的各种病理生物学过程。
在这里,我们概述了管理细胞网络的组织原则,以及这些原则对理解疾病的影响。基于网络的方法有潜在的生物学和临床应用,从疾病基因的识别到更好的药物靶标。
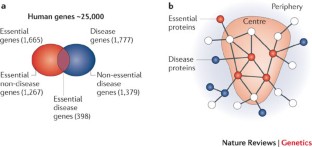
虽然基本基因往往与中枢或高度连接的蛋白质相关,但疾病基因往往在网络的功能外围分离,避开中枢。
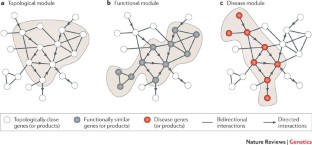
疾病基因有很高的相互作用的倾向,形成疾病模块。这些疾病模块的识别可以帮助我们识别疾病途径和预测其他疾病基因。
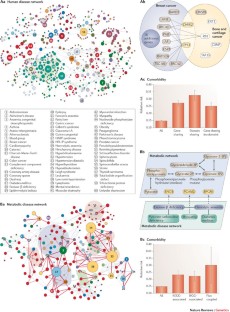
的相互作用组装置的高度互连的性质是,在分子水平上,则难以考虑疾病为彼此独立的。pathophenotypes之间的基于网络的依赖性的映射已最终于diseasome的概念,它代表疾病地图的节点是疾病和其链接代表疾病相关的细胞成分之间的各种分子的关系。
在分子水平相关的疾病往往表现出检测合并症。
网络医学在药物设计方面有着重要的应用,导致了网络药理学的出现,也导致了疾病分类的出现。
摘要
考虑到人类细胞中分子成分之间功能上的相互依赖关系,一种疾病很少是单个基因异常的结果,而是反映了连接组织和器官系统的细胞内和细胞间复杂网络的紊乱。新兴的网络医学工具提供了一个平台,不仅可以系统地探索特定疾病的分子复杂性,从而识别疾病模块和途径,而且还可以系统地探索明显不同(病理)表型之间的分子关系。这一方向的进展对于识别新的疾病基因、揭示全基因组关联研究和全基因组测序发现的疾病相关突变的生物学意义以及识别复杂疾病的药物靶标和生物标志物至关重要。
访问选项
订阅期刊
获得为期1年的完整日记账访问权限
59.00美元
每期仅4.92美元
所有价格均为净价。
VAT将在结账后稍后添加。
税收计算将在结账时完成。
租赁或购买物品
在ReadCube上获取时间限制或全文访问。
从8.99美元
所有价格均为净价。


参考文献
- 1
修饰特异性蛋白质组学:利用富集技术鉴定翻译后修饰的策略。蛋白质组学9, 4632 - 4641(2009)。
- 2
Venkatesan,K.等人。二元相互作用组映射的经验框架。自然方法6, 83 - 90(2008)。
- 3.
普通遗传变异与人类特征。心血管病。j .地中海。360, 1696 - 1698(2009)。
- 4
Schadt,E.E.将分子网络作为人类常见疾病的传感器和驱动因素。本性461, 218 - 223(2009)。
- 5
巴斯A.-L。网络医学——从肥胖到“疾病”。心血管病。j .地中海。357, 404–407 (2007).
- 6
帕森,T.&林鼎,R.网的远程医疗。2月。582, 1266 - 1270(2008)。
- 7
张志强,张志强,张志强,等。网络医学在人类疾病研究中的应用。2月。583, 1759–1765 (2009).
- 8
疾病中的蛋白质网络。基因组Res。18, 644 - 652(2008)。
- 9
农村,肯尼迪。et al。人类蛋白质-蛋白质相互作用网络的蛋白质组尺度图。本性437, 1173 - 1178(2005)。
- 10
等。人类蛋白质-蛋白质相互作用网络:注释蛋白质组的资源。细胞122, 957 - 968(2005)。
- 11
郑,H.等。代谢网络的大规模组织。本性407, 651 - 654(2000)。
- 12
费尔,d。a。瓦格纳,a。新陈代谢的小世界。自然生物技术。18, 1121 - 1122(2000)。
- 13
杜阿尔特等。基于基因组和文献组学数据的人体代谢网络的全球重建。美国国家科学院学报。美国104, 1777 - 1782(2007)。
- 14
卡宁西等。哺乳动物基因组的转录景观。科学309, 1559 - 1563(2005)。
- 15
林丁等。NetworKIN:探索细胞磷酸化网络的资源。核酸RES。36D695-D699(2008)。
- 16
保守的种子配对,通常在腺甘酸的两侧,表明成千上万的人类基因是小核糖核酸的靶点。细胞12015 - 20(2005)。
- 17
刘志强等。用于RNA干扰的siRNA设计。自然生物技术。22, 326–330 (2004).
- 18
Stuart, J. M. et al。全球发现保守遗传模块的基因共表达网络。科学302,249-255(2003)。
- 19
布恩,C.,布西,H. &安德鲁斯,B. J.探索与酵母的遗传相互作用和网络。自然遗传学牧师。8, 437 - 449(2007)。
- 20.
Beltrao,P.,卡格尼,G。&Krogan,N.定量遗传相互作用揭示生物模块化。细胞141, 739 - 745(2010)。
- 21
挖掘组织特异性、基因连通性和疾病相关性,揭示一组修饰致病基因行为的基因。个人经历分钟。1,8(2008)。
- 22
Lage, K.等。对人类疾病基因和复合物的组织特异性病理和基因表达的大规模分析。美国国家科学院学报。美国105, 20870 - 20875(2008)。
- 23
Lage, K.等。解剖时空蛋白质网络推动人类心脏发育和相关疾病。摩尔。系统。医学杂志。6381(2010)。
- 24
Schwartz,A.S.,Yu,J.,Gardenour,K.R.,Finley,R.L.&Ideker,T.完成interactome的成本效益策略。自然方法6,55-61(2009)。
- 25
Yu,H.等。酵母相互作用组网络的高质量二元蛋白质相互作用图。科学322, 104 - 110(2008)。
- 26
基鲁亚克等。层次组织组织中的动态互动网络。摩尔。系统。医学杂志。6417(2010)。
- 27
巴斯A.-L。网络生物学:理解细胞的功能组织。自然遗传学牧师。5, 101–113 (2004).
- 28
Albert,R.&Barabási,A.-L.《复杂网络的统计力学》。启Mod。物理。7447 - 97(2002)。
- 29
朱,X.,格斯坦,M&斯奈德,M.快速连接:分析和生物网络的原则。基因开发。21, 1010 - 1024(2007)。
- 30.
Caldarelli G。无标度网络(牛津大学出版社,英国,2007)。
- 31
细胞生物学中的无标度网络。j .细胞科学。118, 4947 - 4957(2005)。
- 32
纽曼,M., Barabási, a.l。&瓦茨,d.j.网络的结构和动态(美国普林斯顿大学出版社,2006)。
- 33
Amberger,J.,Bocchini,C.A.,Scott,A.F.和Hamosh,A.McKusick的在线孟德尔遗传(OMIM)®).核酸RES。37,D793-D796(2009)。
- 34
郑,H.等。蛋白质网络中的致命性和中心性。本性411, 41–42 (2001).
- 35
陈志强等。蛋白质相互作用网络中的进化速率。科学296, 750 - 752(2002)。
- 36
Eisenberg,E.&Levanon,E.Y.蛋白质网络进化中的优先连接。物理。rev. lett。91, 138701 (2003).
- 37
蛋白质-蛋白质相互作用,进化速率,丰度和年龄。BMC生物信息学7, 128 (2006).
- 38
蛋白质进化速率和蛋白质相互作用的数量之间不存在简单的依赖关系:只有最多产的相互作用者倾向于进化缓慢。BMC Evol。医学杂志。3.5(2003)。
- 39
和知,S.,米田,K。和Wu,R.相互作用组转录组分析揭示了在肺癌组织中差异表达的基因的高中心地位。生物信息学21, 4205 - 4208(2005)。
- 40
在人类相互作用组中癌症蛋白的整体拓扑特征。生物信息学22,2291年至2297年(2006年)。
- 41
基于蛋白质相互作用网络拓扑结构的疾病基因发现。生物信息学22,2800至05年(2006年)。本文表明,利用蛋白质相互作用网络的拓扑特征可以发现疾病基因。
- 42
吴作栋,K.-I。et al。人类疾病网络。美国国家科学院学报。美国104, 8685 - 8690(2007)。本文通过联疾病份额疾病基因构建第一疾病网络,并且它表明,大多数疾病的基因是非必需,并且不受毂蛋白编码。
- 43
关键词:遗传疾病,基因突变,网络特性美国国家科学院学报。美国105, 4323 - 4328(2008)。
- 44
从分子到模块化细胞生物学。本性402C47-C52(1999)。
- 45
Oti, M.等。利用蛋白质相互作用预测疾病基因。j .地中海,麝猫。43, 691 - 698(2006)。本文探讨了与已知疾病基因相关的蛋白质在多大程度上也与同一表型相关。
- 46
甘地,T。等。人类蛋白质相互作用组的分析以及与酵母、蠕虫和苍蝇相互作用数据集的比较。自然遗传学。38, 285 - 293(2006)。
- 47
社会网络与生物网络的社群结构。美国国家科学院学报。美国99, 7821–7826 (2002).
- 48
研究自然与社会中复杂网络的重叠群落结构。本性435, 814 - 818(2005)。
- 49
安,y y。,Bagrow, J. P. & Lehmann, S. Link communities reveal multiscale complexity in networks.本性466, 761 - 764(2010)。
- 50
一种大规模检测蛋白质家族的高效算法。核酸RES。30., 1575 - 1584(2002)。
- 51
拉瓦兹,E., Somera, A. L., Mongru, D. A., Oltvai, Z. N. & Barabási, A. L.。代谢网络模块化的层次组织。科学297, 1551 - 1555(2002)。
- 52
Wood, l.d.等。人类乳腺癌和直肠癌的基因组景观。科学318, 1108 - 1113(2007)。
- 53
Hirschhorn,J.N.全基因组关联研究——阐明生物学途径。心血管病。j .地中海。360,1699至1701年(2009年)。
- 54
Krauthammer,M.等人。分子三角测量:识别阿尔茨海默病候选基因的桥接连锁和分子网络信息。美国国家科学院学报。美国101, 15148 - 15153(2004)。
- 55
法兰克,L.等。功能人类基因网络的重建及其在定位候选基因排序中的应用。点。j .的嗡嗡声。麝猫。78, 1011 - 1025(2006)。
- 56
Iossifov,I.,郑,T.,男爵,M.,吉利厄姆T.C。&Rzhetsky,A.复杂遗传疾病的全基因组分子相互作用网络的遗传联的映射。基因组Res。18, 1150 - 1162(2008)。
- 57
Navlakha, S. & Kingsford, C.将基因与疾病联系起来的蛋白质相互作用网络的力量。生物信息学26,1057年至1063年(2010年)。本文可用的致病基因预测方法进行比较,显示出基于随机行走的工具跑赢clustering-和基础连接的方法。
- 58
Lage,K.等人。一种与遗传疾病有关的蛋白质复合物的人类表型相互作用组网络。自然生物技术。25, 309 - 316(2007)。
- 59
李,E.等。AML基因在失调分子网络中的分析。BMC生物信息学10,S2(2009年)。
- 60
Bonifaci,N。等人。生物过程,性质和候选低外显率的乳腺癌易感基因分子的接线图。BMC医学基因组学。1, 62 (2008).
- 61
等。乳腺癌细胞系的综合分析揭示了独特的信号通路。基因组生物学。10R31(2009)。
- 62
壮族,H.-Y。et al。基于网络的乳腺癌转移分类。摩尔。系统。医学杂志。3.140(2007)。
- 63
尼伯,R. K.等。识别晚期结肠癌蛋白相互作用亚网络的发现和评分。摩尔。细胞。蛋白质组学8,827-845(2009)。
- 64
张,W.等。胃癌肝转移相关新hub基因的鉴定国际癌症杂志125, 2844 - 2853(2009)。
- 65
的Ergun,A.,劳伦斯,C. A.,Kohanski,M.A.,布伦南,T.A。&柯林斯,J. J.网络生物学方法对前列腺癌。摩尔。系统。医学杂志。3.82(2007)。
- 66
蛋白质相互作用网络中的动态模块性预测乳腺癌的预后。自然生物技术。27, 199–204 (2009).本文探讨了枢纽的模块化性质是否可用于预测患者结局,并将其应用于乳腺癌。
- 67
Moran, L. B. & Graeber, M. B.研究帕金森病的路径定义:一种与癌症、糖尿病和炎症相关的复杂疾病。神经遗传学91-13(2008)。
- 68
阿尔茨海默病转录组的变异揭示了心血管疾病的分子网络。基因组生物学。9R148(2008)。
- 69
黄,D.等。朊病毒疾病的系统方法。摩尔。系统。医学杂志。5252(2009)。
- 70
惠洛克,C. E.等。研究心血管疾病的系统生物学方法和途径工具。摩尔。Biosyst。5, 588–602 (2009).
- 71
卡尔瓦诺,S. E.等。全身性炎症的网络分析。本性437, 1032 - 1037(2005)。
- 72
Iliopoulos,D。等人。一体化微RNA和蛋白质组方法鉴定新的骨关节炎基因和它们的协同代谢和炎症网络。公共科学图书馆一号3.e3740(2008)。
- 73
陈勇等。DNA的变异说明了引起疾病的分子网络。本性452, 429 - 435(2008)。
- 74
等。基因表达的遗传学及其对疾病的影响。本性452, 423 - 428(2008)。
- 75
多布林等。多组织共表达网络揭示了与疾病相关的意外亚网络。基因组生物学。10R55(2009)。
- 76
等。与哮喘相关的蛋白质相互作用网络。理论。比尔。252, 722–731 (2008).
- 77
Liu,M.等人。2型糖尿病模型中受影响生物过程的网络分析。公共科学图书馆麝猫。3.e96(2007)。
- 78
等。综合加权基因共表达网络分析及其在慢性疲劳综合征中的应用。BMC系统。医学杂志。295(2008)。
- 79
Uetz等。疱疹病毒蛋白质网络及其与人类蛋白质组的相互作用。科学311, 239 - 242(2006)。
- 80
卡尔德伍德,m.a.等。eb病毒和病毒人蛋白相互作用图。美国国家科学院学报。美国104, 7606 - 7611(2007)。
- 81
谢伦伯格,J.,帕尔松,B. Ø。& Jamshidi, N.洞见人肺泡巨噬细胞和结核分枝杆菌通过代谢重建的互动。摩尔。系统。医学杂志。6422(2010)。
- 82
宏基因组学与代谢组学的联姻。细胞134, 708–713 (2008).
- 83
Goehler等。一种蛋白质相互作用网络将GIT1连接到亨廷顿舞蹈症,GIT1是一种亨廷顿舞蹈症的增强剂。摩尔。细胞15, 853–865 (2004).
- 84
等。人类遗传性共济失调和浦肯野细胞变性紊乱的蛋白质-蛋白质相互作用网络。细胞125, 801 - 814(2006)。本文采用酵母双杂交法,绘制脊髓小脑共济失调蛋白与其他人类蛋白的相互作用图谱,构建共济失调模型。
- 85
Pujana, m.a.等。网络建模联系乳腺癌易感性和中心体功能障碍。自然遗传学39, 1338 - 1349(2007)。
- 86
L. M. Camargo等。精神分裂症相互作用组中断:精神分裂症风险基因紧密连接和潜在突触基础的证据。精神病学摩尔。12, 74–86 (2007).
- 87
氨基,T.等。重新定义16q22.1连锁常染色体显性小脑共济失调的疾病位点。j .的嗡嗡声。麝猫。52, 643 - 649(2007)。
- 88
科勒等。行走相互作用组以确定候选疾病基因的优先级。点。j .的嗡嗡声。麝猫。82,949-958(2008)。
- 89
Vanunu, O.等。通过网络传播将基因和蛋白质复合物与疾病联系起来。公共科学图书馆第一版。医学杂志。6,e1000641(2010)。
- 90
王志强等。细胞网络对疾病共病的影响。摩尔。系统。医学杂志。5262(2009)。这篇论文表明,共享基因或涉及相互作用的蛋白质的疾病显示出较高的共病率,表明了细胞网络结构和人群疾病模式之间的相关性。
- 91
达力,A. M.,Janse,D. M.,Tanay,A.,沙米尔,R。&教堂,G. M.全局多效性的视图,并在酵母表型衍生的基因功能。摩尔。系统。医学杂志。11(2005)。
- 92
钟强等。人类遗传疾病的边缘扰动模型。摩尔。系统。医学杂志。5, 321 (2009).
- 93
李,D.-S.et al。人体代谢网络拓扑的疾病共病的影响。美国国家科学院学报。美国105, 9880 - 9885(2008)。本文通过将疾病与相邻代谢反应联系起来,并发现相关疾病的高共病率,构建了代谢疾病的关联。
- 94
吕晓东,等。人类microRNA与疾病的相关性分析。公共科学图书馆一号3.e3420(2008)。
- 95
等。探索复杂的人类表型之间的遗传重叠。美国国家科学院学报。美国104, 11694–11699 (2007).的患者记录此分析表明,疾病表型形成强成对的相关性的高度连接的网络,帮助研究人员对推定的遗传重叠的大小。
- 96
伊达尔戈等。人类表型研究的动态网络方法。公共科学图书馆第一版。医学杂志。5,e1000353(2009年)。本文通过利用3000万名医保患者的病史获得的数据显著合并症链接疾病推出了PDN。由此看来,研究人员建立了一个开放式访问合并症数据库。
- 97
面包车Driel,M.A.等人。人类phenome的文本挖掘分析。欧·J·哼·吉奈。14, 535 - 542(2006)。
- 98
Suthram, S.等。基于网络的人类疾病的阐明,丰富了多能药物靶点的人类疾病的阐明。公共科学图书馆第一版。医学杂志。6e1000662(2010)。
- 99
刘玉仪,怀斯,P.H.和巴特,A.J.“病因”:人类疾病病因的识别和聚类。BMC生物信息学10S14系列(2009)。
- One hundred.
Campillos, M, Kuhn, M, Gavin, a - c。,Jensen, L. J. & Bork, P. Drug target identification using side-effect similarity.科学321, 263–266 (2008).
- 101
Kuhn, M., Campillos, M., Letunic, I, Jensen, L. J. & Bork, P. A副作用资源来捕获药物的表型效应。摩尔。系统。医学杂志。6343(2010)。
- 102
Audouze,K.等人,《破译疾病和生物信息》目标。使用毒性基因组学网络用于环境化学品。公共科学图书馆第一版。医学杂志。6e1000788(2010)。
- 103
疾病与复合筛查的网络观点。自然药物光盘。8, 286 - 295(2009)。
- 104
药物发现:预测滥交。本性462, 167 - 168(2009)。
- 105
楚,L.&陈,对细胞凋亡的药物靶点发现与癌症干扰蛋白质 - 蛋白质相互作用网络的B. S.建设。BMC系统。医学杂志。256(2008)。
- 106
Azmi,A.,Wang,Z.,Philip,P.A.,Mohammad,R.M.和Sarkar,F.H.概念证明:网络和系统生物学方法如何帮助发现有效抗癌药物组合的综述。摩尔。癌症。”(doi: 10.1158/1535-7163.MCT-10-0642)。
- 107
Zhao,S.&Li,S.基于网络的药物靶点识别相关药理学和基因组空间。公共科学图书馆一号5e11764(2010)。
- 108
芳,S. S.&保尔松,B.Ø。的代谢基因缺失菌株大肠杆菌进化到计算预测的生长表型。自然遗传学。36, 1056 - 1058(2004)。
- 109
Segrè,D.,Vitkup,D.&Church,G.M.自然和扰动代谢网络中的最优性分析。美国国家科学院学报。美国99,15112-15117(2002)。
- 110
沈勇等。针对代谢网络的抗菌素打击发现蓝图。美国国家科学院学报。美国107,一零八二年至1087年(2010年)。
- 111
Motter, a.e., Gulbahce, N. Almaas, E. & Barabási, a.l.。预测代谢网络中的合成救援。摩尔。系统。医学杂志。4168(2008)。
- 112
诺兰,G.P.今天药物筛查有什么问题吗。化学性质。医学杂志。3.,187-191(2007)。
- 113
多靶点药物的有效性:网络方法可能有助于药物设计。趋势杂志。科学。26,178-182(2005)。
- 114
Motter,A.E.通过对抗改善网络性能:从合成救援到多种药物组合。生物信息32, 236 - 245(2010)。
- 115
杨,K等人。在寻找疾病相关的分子网络中的多目标优化干预。摩尔。系统。医学杂志。4,228(2008)。
- 116
最佳药物组合和最小打击集。BMC系统。医学杂志。3.,81(2009)。
- 117
Yildirim, m.a.等。药物靶网络。自然生物技术。25, 1119 - 1126(2007)。
- 118
凯泽,M. J.等。预测已知药物的新分子靶点。本性462, 175 - 181(2009)。
- 119
何志强,陈志强,陈志强。一种肥厚性心肌病的治疗方法。循环113e858-e862(2006)。
- 120
森田,Y。等。在儿童和成人cadiac肥大的共享遗传原因。心血管病。j .地中海。358, 1899 - 1908(2008)。
- 121
Loscalzo, J., Kohane, I., Barabási, a.l。后基因组时代的人类疾病分类:人类病理生物学的复杂系统方法。摩尔。系统。医学杂志。3.124(2007)。
- 122
Dreze, M.等。高质量的二进制交互映射。冰毒。Enzymol。470, 281 - 315(2010)。
- 123
尤因等。用质谱法绘制人体蛋白质相互作用的大规模图谱。摩尔。系统。医学杂志。3.89(2007)。
- 124
M. E. Cusick等人。文献策划的蛋白质相互作用数据集。自然方法639-46(2009)。
- 125
马,H。等。爱丁堡人体代谢网络重建及其功能分析。摩尔。系统。医学杂志。3., 135 (2007).
- 126
巴斯A.-L。随机网络中缩放的出现。科学286, 509–512 (1999).
- 127
韩,J. D.等。酵母蛋白-蛋白相互作用网络中动态组织模块化的证据。本性430, 88–93 (2004).
- 128
“小世界”网络的集体动态。本性393, 440 - 442(1998)。
- 129
米洛,R.,申奥尔,S.,伊兹科维茨,S.,北卡什坦,契克洛夫斯基和阿隆,U.网络主题:复杂网络的简单构建块。科学298, 824 - 827(2002)。
- 130
于,H.,金,P. M.,Sprecher的,E.,特里福诺夫,V.&格斯坦,M.瓶颈中蛋白质网络的重要性:与基因必要性和表达动力学的相关性。公共科学图书馆第一版。医学杂志。3.,E59(2007年)。
- 131
吴旭,蒋锐,张明强,李胜。基于网络的人类疾病基因的全局推断。摩尔。系统。医学杂志。4, 189 (2008).
致谢
我们感谢Z. Oltvai, A. Sharma, d . s。Lee和J. Park进行了有益的讨论和建议。A.L.B.和N.G.获得了美国国立卫生研究院(NIH)通过基因组科学卓越中心(CEGS)的资助,J.L.获得了NIH HL061795 (Merit Award)、HL81587、HL70819和HL48743的资助。
作者信息
隶属关系
相应的作者
道德声明
利益争夺
作者声明没有相互竞争的经济利益。
相关链接
词汇表
- 节点(或顶点)
-
一种系统组件,通过与其他组件相互作用形成一个网络。在生物网络中,节点可以表示蛋白质、基因、代谢物、RNA分子,甚至疾病和表型。
- 链接(或边)
-
链路表示网络中各节点之间的相互作用。在生物系统中,相互作用可以对应于蛋白质-蛋白质结合相互作用或代谢耦合,或者它们可能代表基于共同遗传起源或共同表型特征的疾病之间的联系。
- 学位
-
一个节点的度是连接到它的链路的数量。一种蛋白质的程度可以表示与它相互作用的蛋白质的数量,而一种疾病的程度可以表示与同一基因相关或具有共同表型的其他疾病的数量。
- 模块(或社区)
-
网络上的密集子图,通常表示具有联合角色的一组节点。在生物学中,一个模块可以对应于一组相互作用以实现某些共同功能的分子。
- 伴随疾病
-
共病是指除患者原有疾病或疾病外,还存在一种或多种疾病(或疾病)。当一种疾病加剧了其他疾病的出现时,共病可能掩盖了因果关系,例如被广泛研究的糖尿病和肥胖之间的共病。
- Edgetic
-
边缘扰动是指突变不会导致基因产物完全丢失,而是影响蛋白质的一种或几种相互作用(从而影响蛋白质的功能)。从网络的角度来看,边缘扰动移除了一个或几个链接,但不影响其他链接和节点。
权利和权限
关于这篇文章
引用这篇文章
Barabási, AL., Gulbahce, N. & Loscalzo, J.网络医学:人类疾病的基于网络的方法。Nat牧师麝猫12,56 - 68(2011)。https://doi.org/10.1038/nrg2918
发表:
发行日期:
进一步的阅读
基于人工智能的计算框架,用于阿尔茨海默病药物靶标优先排序和新型可重新定位药物推理
阿尔茨海默病研究与治疗(2021)
患者从动态模块化网络的慢性肝病的肝细胞肝癌的风险预测
转化医学杂志(2021)
转化医学精度:行业观点
转化医学杂志(2021)
新冠病毒-19的深度学习应用
大数据杂志(2021)
基于致癌信号通路的系统药理学方法,通过转录组数据确定天然产物在乳腺癌中的作用机制
补充医学和治疗(2021)
