文摘
人类基因组编码生命的蓝图,但绝大多数的功能其近三十亿基地是未知的。百科全书的DNA元素(编码)项目系统映射区域的转录,转录因子协会,染色质结构和组蛋白修饰。这些数据使我们能够分配生化功能基因组的80%,特别是在研究蛋白质编码区域。许多发现的候选监管元素彼此身体相关表达基因,基因调控机制提供了新的见解。新发现的元素也显示一个统计对应的序列变异与人类疾病有关,并且可以从而指导解释这种变化。总的来说,该项目提供了新的见解的组织和监管我们的基因和基因组功能注释和是一个广阔的资源用于生物医学研究。
主要

人类基因组序列为人类生物学提供了底层代码。尽管深入研究,特别是在确定蛋白质编码基因,我们理解的基因组还远未完成,特别是关于非编码rna,或者拼接成绩单和监管序列。系统分析记录和监管信息至关重要基因的鉴定和监管区域,和人类生物学研究是一个重要的资源和疾病。这种分析也可以提供全面的视图的组织和跨细胞基因变异和监管信息情况下,物种和个体。
DNA序列元素的百科全书(编码)项目旨在描绘所有的功能元素在人类基因组中编码1,2,3。操作上,我们定义了一个功能性元素作为一个离散的基因组片段编码定义产品(例如,蛋白质或非编码RNA)或显示一个可再生的生化特征(例如,蛋白结合,或一个特定的染色质结构)。比较基因组研究表明,3 - 8%的基地正在净化(消极的)选择4,5,6,7,8因此可能功能,虽然其他的分析提出了更高的预期9,10,11。覆盖1%的基因组在试点阶段,编码项目注释60%的哺乳动物的进化约束的基地,但还发现了许多其他公认的功能性元素没有约束的证据2。现在更强大的DNA测序技术的出现使全基因组和更精确的分析和广泛的功能分析。
在这里,我们描述了1640年生产和初步分析数据集设计在整个人类基因组注释的功能元素。我们整合来自不同实验结果在细胞类型,相关实验涉及147种不同的细胞类型,和所有编码数据与其他资源,如候选人地区从全基因组关联研究(GWAS)和进化限制区域。在一起,这些努力揭示重要特征对人类基因组的组织和功能,总结如下。
•绝大多数(80.4%)的人类基因组至少参与一个生化RNA -和/或chromatin-associated事件至少在一个细胞类型。基因组的谎言接近监管事件:95%的基因组在8个碱基(kb) dna蛋白质相互作用(如化验绑定ChIP-seq图案或DNase我脚印),和99%在1.7 kb的至少一个生化事件的编码。
•Primate-specific元素以及元素没有检测到哺乳动物约束显示,总的来说,负选择的证据;因此,他们中的一些人预计将功能。
•分类基因组为七个染色质状态表示一组初始的399124个地区与promoter-like enhancer-like特性和70292个地区特性,以及成千上万的静止区域。高分辨率分析进一步细分基因组为成千上万的狭窄的状态具有不同的功能特性。
•可以关联定量RNA序列生产加工与染色质标记和启动子转录因子结合,表明启动子功能可以解释大部分的RNA表达的变化。
•许多非编码变异个体基因组序列躺在ENCODE-annotated功能区域;这个数字至少是那些躺在蛋白编码基因一样大。
•单核苷酸多态性(snp)与疾病相关的GWAS浓缩在非编码的功能元素,与多数居住在或接近ENCODE-defined蛋白编码基因以外的区域。在许多情况下,这种疾病表型可以关联到一个特定的细胞类型或转录因子。
编码数据生产和初步分析
自2007年以来,编码开发方法和执行大量的序列研究映射整个人类基因组的功能元素3。映射的元素(和方法使用)包括RNA转录区域(RNA-seq笼,RNA-PET和人工注释),蛋白质编码区域(质谱),transcription-factor-binding网站(ChIP-seq和DNase-seq),染色质结构(DNase-seq FAIRE-seq,组蛋白ChIP-seq和MNase-seq),和DNA甲基化网站(rrb试验)(箱1列表方法和缩写;补充表1节P,细节生产统计数据)3。在不同的实验室,比较和整合结果数据生产的努力集中在两组选择的细胞系,指定“一级”和“二级”(箱1)。捕捉范围更广泛的生物多样性,选择化验也执行第三层包括超过100细胞类型包括的主要细胞。所有可用的数据和协议描述http://www.encodeproject.org/和用户指南包括程控的细节的选择和限制是最近出版的3。
集成方法
为了一致性,数据生成和加工使用标准化的指导方针,和一些化验,新的质量控制措施的设计(见参考文献3,12和http://encodeproject.org/ENCODE/dataStandards.html;a . Kundaje个人沟通)。统一的数据处理方法为每个试验(见发达补充信息;a . Kundaje个人通信),大多数试验结果可以作为信号信息(每个基站估计整个基因组)和离散元素(地区计算确定为丰富信号)。广泛的处理管道开发生成每个表示(m·m·霍夫曼et al。在准备和a . Kundaje手稿,个人沟通)。此外,我们开发了不能再现的发现率(IDR)13测量提供一个健壮的和保守的估计的阈值两个排名列表从生物复制的结果不再同意(也就是说,不能复制的),我们应用这个定义的离散元素集。我们确认,排除在分析,大部分区域产生不可靠的信号可能出土文物(例如,multicopy地区)。在一起,这些地区占0.39%的基因组(见补充信息)。这个问题是不同的ENCODE-identified附带的海报元素及其基因组覆盖率。
转录和蛋白质编码区域
我们使用手动和自动注释产生全面的人类蛋白质编码和非编码rna以及假基因,称为GENCODE参考基因集14,15(补充表1,部分U)。这包含了20687个蛋白编码基因(GENCODE注释,v7),平均6.3或者拼接记录每个轨迹不同蛋白质编码记录(3.9)。总的来说,GENCODE-annotated蛋白编码基因的外显子覆盖2.94%的基因组蛋白质编码外显子或1.22%。从最外层蛋白编码基因跨越33.45%开始停止密码子,从启动子或39.54%聚(A)的网站。质谱数据的分析从K562和GM12878细胞株产生57自信确定独特的肽序列基因间区域相对于GENCODE注释。结合基因组转录无处不在的证据16,这些数据表明,额外的蛋白质编码基因仍有待发现。
此外,我们注释8801自动派生小RNA和9640手动策划长非编码RNA (lncRNA)位点17。lncRNAs比作其他编码数据表明lncRNAs生成蛋白质编码基因通过类似的途径17。GENCODE项目还注释11224假基因,其中863是转录和染色质与活跃18。
核糖核酸
我们测序RNA16从不同的细胞系和多个亚细胞分数来开发一个广泛的RNA表达的目录。使用保守的RNA的阈值来确定地区活动,62%的测序基因组基地可重复代表长(> 200个核苷酸)RNA分子或GENCODE外显子。这些基地,只有5.5%被GENCODE外显子解释道。大多数转录基地内或重叠带注释的基因边界(即intronic),只有31%的基地测序转录基因间16。
我们使用CAGE-seq (5′cap-targeted RNA隔离和测序)转录起始点的识别62403 (tss)高信心0.01 (IDR)层1和2细胞类型。其中,27362例(44%)在100个碱基对(bp)的5′末端GENCODE-annotated成绩单或之前报道全身的信使RNA。其余地区主要和3′外显子躺在区域(utr),和一些展览cell-type-restricted表达式;这些可能代表一开始网站的小说,cell-type-specific记录。
最后,我们看到了很大一部分的编码和非编码转录加工成稳态稳定比200个核苷酸短rna。这些前体包括转移核糖核酸、小核RNA和小核仁的微RNA, RNA(分别为tRNA, microRNA、核内小RNA和snoRNA)和这些加工产品的5′末端结合封顶5′末端标记16。
蛋白结合的区域
直接识别监管区域,我们绘制了绑定的位置119种不同的dna结合蛋白质和RNA聚合酶组件的数量在72年使用ChIP-seq细胞类型(表1,补充表1,部分N和裁判。19);87(73%)被sequence-specific转录因子。总的来说,636336个绑定地区占地231 megabases (Mb;8.1%)的基因组区域富集受dna结合蛋白在所有类型的细胞。我们评估每个此种网站已知dna结合蛋白的浓缩主题和小说主题的存在。总体来看,86%的DNA片段被sequence-specific转录因子包含一个强大的DNA结合主题,而且在大多数(55%)例已知的主题是最丰富(p . Kheradpour和m . >手稿准备)。
此种地区缺乏高同源或中度亲和力识别网站平均分数的排名低于21%的地区(Wilcoxon排名和识别序列P值< 10−16)。百分之八十二的低信号地区的高亲和性识别序列的其他因素。此外,当ChIP-seq山峰排名由他们与已知的识别序列的一致性,中位数DNase我可访问性是两个在底部20%的峰值高于上80%(基因组结构调整(GSC)20.P值< 10−16),与以前的观测一致21,22,23,24。我们推测低信号区域要么lower-affinity网站21或间接目标区域相关转录因子通过与其他因素的相互作用(参见参考文献25,26)。
我们组织所有的信息与每个转录factor-including ChIP-seq山峰,发现组蛋白的化学修饰图案和相关模式FactorBook (http://www.factorbook.org;ref。26),一种公共资源,将随着项目的进行更新。
DNase我过敏的网站和脚印
染色质易访问性特征是DNase我过敏是监管DNA区域的标志27,28。我们映射289万独特的重叠DNase我过敏的网站(dhs) 125年由DNase-seq细胞类型,绝大多数tss谎言远端29日。我们也映射480万网站25显示nucleosomal交联减少了做的细胞类型,其中许多配合国土安全部。此外,我们使用微球菌的核酸酶绘制核小体在GM12878入住率和K562细胞30.。
在一级和二级细胞类型中,我们发现了一个205109年的意思是国土安全部每细胞类型(在错误发现率(罗斯福)1%),包括平均1.0%的基因组序列在每个细胞类型,和总体的3.9%。平均98.5%的入住率的转录因子由编码映射ChIP-seq(集体,94.4%的所有110万个转录因子ChIP-seq山峰在K562细胞)内访问染色质DNase我定义的热点29日。然而,少量的因素,特别是heterochromatin-bound压制性复合物(例如,TRIM28-SETDB1-ZNF274复杂31日,32编码的TRIM28,SETDB1和ZNF274基因),似乎占据nucleosomal网站的一个重要部分。
利用基因组DNase我足迹33,34在41个细胞类型我们确定了840万种不同DNase我足迹(罗斯福1%)25。我们的新创主题发现DNase我足迹恢复∼90%的已知的转录因子主题,以及数以百计的小说的进化图案,许多显示高度cell-selective占用模式类似于主要的发展和组织的监管机构。
区域组蛋白修饰
我们检测染色体位置12个组蛋白修饰和变异46个细胞类型,包括一个完整的矩阵在一级和二级的八个修改。因为修改州可能跨越多个核小体,自己可以跨细胞群位置不同,我们使用一个连续信号测量的组蛋白修饰在下游分析中,而不是调用区域(m·m·霍夫曼et al。,在准备手稿;看到http://code.google.com/p/align2rawsignal/)。最强的峰样的组蛋白修饰,我们使用mac电脑35描述丰富的网站。表2描述了不同的组蛋白修饰,峰值特征,总结他们的角色(参了》)。
我们的数据显示,全球模式修改是高度可变的细胞类型,按照转录活性的变化。与以前的研究一致40,41,我们发现可以使用不同的组蛋白修饰信息集成系统的功能属性分配给基因组区域(见下文)。
DNA甲基化
胞嘧啶的甲基化,通常在CpG二核苷酸,参与基因表达的表观遗传调控。启动子甲基化通常是与镇压,而基因的甲基化与转录相关的活动42。我们使用了表示亚硫酸氢盐测序(rrb)剖面DNA甲基化定量82年平均每人120万论文认定的细胞系和组织非重复性的基因组论文认定(8.6%),包括论文认定基因间区域,近端启动子和基因内区(基因的身体)43,但应该注意的是,rrb方法优先目标CpG-rich岛屿。我们发现96%的论文认定表现出差异甲基化在至少一个细胞或组织化验(k .瓦利et al。、个人通信)和DNA甲基化水平与染色质可访问性。最不定地常常在基因甲基化论文认定发现身体和基因间区域,而不是在启动子和上游监管区域。此外,我们发现一个意想不到的对应unmethylated基因的CpG岛P300和绑定,一个组蛋白乙酰转移酶与增强子活动44。
因为rrb与单碱基序列分析解决,我们能够识别论文认定allele-specific甲基化与基因组印记一致,并确定这些位点表现出异常的甲基化在癌症细胞系(k .瓦利et al。个人沟通)。此外,我们发现可再生的胞嘧啶甲基化在成人CpG二核苷酸外组织45提供进一步的支持,这个非规范甲基化事件可能有重要的作用在人类生物学(k .瓦利et al。个人沟通)。
Chromosome-interacting地区
物理之间的相互作用不同的染色体区域,可以由数百个碱基被认为是重要的基因表达的调节46。我们使用两种互补染色体构象捕获(3 c)的技术来探测这些远程物理交互。
3 c-carbon副本(5 c)的方法47,48提供公正的检测与tss远程交互目标1%的基因组(44编码试点地区)在四个细胞类型(GM12878、K562 HeLa-S3和H1 hESC)49。我们发现了数以百计的统计学意义在每个远程交互后细胞类型占染色质聚合物和实验变异行为。对相互作用位点表明基因表达水平的相关性强TSS和特定功能增强剂等元素类。远端元素相互作用TSS的平均数为3.9,和TSS的平均数为2.5,与远端交互元素表示一个复杂的网络互联染色质。这种交织远程架构也发现了全基因组与paired-end标记使用染色质交互分析测序(ChIA-PET)50应用于识别交互染色质丰富的RNA聚合酶II (Pol II)芯片从五个细胞类型51。在K562细胞中,我们确定了127417年promoter-centred染色质交互使用ChIA-PET,其中98%是intra-chromosomal。而2324个基因的启动子区域参与了“单基因”enhancer-promoter交互,的19813个基因参与的多基因相互作用复合物生成几个megabases,包括promoter-promoter和enhancer-promoter交互51。
这些分析描述复杂的远程景观基因要素连接在数百个碱基数megabases范围,包括相互作用无关的基因(补充图1,部分Y)。此外,在5度结果,50 - 60%的远程交互发生在只有一个的四个细胞系,表明高度的组织特异性基因要素连接49。
总结ENCODE-identified元素
占所有这些元素,令人惊讶的是,相当大的一部分人类基因组,80.4%,是由至少一个ENCODE-identified元素(详细补充表1,部分Q)。最广泛的元素类代表不同的RNA类型,覆盖62%的基因组(虽然多数基因内含子内或附近)。地区第二大组蛋白修饰形式类高纯度(56.1%)。不包括RNA和广泛的组蛋白元素,基因组的44.2%。小比例的基因组被占领地区开放的染色质的转录因子(15.2%)或网站绑定(8.1%),有19.4%由至少一个国土安全部或转录因子在所有细胞系ChIP-seq高峰。用我们最保守的评估,8.5%的基地是由transcription-factor-binding-site主题(4.6%)或国土安全部足迹(5.7%)。然而,这仍高出4.5倍数量的蛋白质编码外显子,和两个高于pan-mammalian约束的预计金额。
考虑到编码项目没有化验所有细胞类型,或全部转录因子,特别是抽样一些专业或发育限制细胞谱系,这些比例必须低估总额的功能基础。然而,许多分析进行多个细胞类型,允许评估发现的新元素。国土安全部和CTCF-bound网站,新元素最初的数量增加迅速,陡坡的饱和曲线,然后放缓与越来越多的细胞类型(补充无花果1和2,部分R)。与当前数据,在最平的饱和曲线的一部分,每一个新的细胞类型补充说,平均而言,9500年国土安全部元素(在106个细胞类型)和500 CTCF-binding元素(在49个细胞类型),代表元素数量总数的0.45%。我们模仿饱和度国土安全部和CTCF-binding网站使用威布尔分布(r2> 0.999)和预测饱和约410万(标准错误(东南部)= 108000)和185100年(王新宏。= 18020)网站,分别,这表明我们已经发现大约一半的估计总国土安全部。这些代表一个下界估计,但加强观察,有更多的非编码DNA功能比编码序列或哺乳动物的进化约束基地。
选择的功能元素的影响
从比较基因组研究,至少有3 - 8%的基地正在净化(消极的)选择4,5,6,7,8,9,10,11,这表明这些基地可能潜在的功能。我们之前发现60%的哺乳动物进化限制基地在编码注释的试点项目,但也注意到,许多功能元素缺乏约束的证据2,结论证实52,53,54。现在的多样性和全基因组发生功能元素识别提供了一个前所未有的机会来检查进一步负面的力量选择对人体功能的序列。
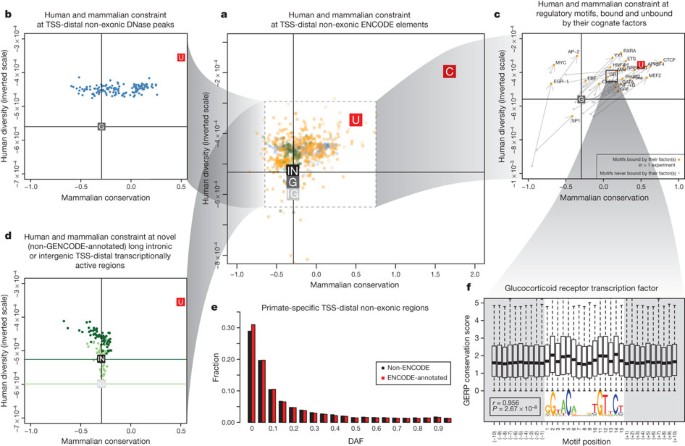
我们检查负选择使用两个措施,强调在人类基因组中不同时期的选择。第一个测量,跨物种,pan-mammalian约束(GERP-based分数;24的哺乳动物8),地址在哺乳动物的进化选择。第二个措施是intra-species约束估计数字的变异在人类发现使用数据从1000人基因工程55人类进化,包括选择。在图1,我们把这两种措施的约束不同类别的识别功能元素,不包括功能重叠的外显子和已知约束的推动者。每个图还显示了基因组背景水平和措施coding-gene约束进行比较。因为我们把人口多样性倒置的规模,元素,更受到消极的选择往往会躺在上面和右边区域的阴谋。
一个pan-mammalian约束水平(意味着GERP的分数;24的哺乳动物8,x轴)与多样性相比,负选择的衡量人口(平均期望杂,倒规模,y轴)编码数据集。平均每个点是一个数据集。右上的角落有最强的进化约束和多样性最低。编码(C)、UTR (U)基因组(G),基因间(IG)和intronic平均值显示为满广场。在每种情况下的垂直和水平十字准线显示代表水平中性期望的哺乳动物保护和人类人口的多样性,分别。分布在所有non-exonic编码元素大于2.5 kb tss。内部的虚线框显示部分情节已经放大了周围的外板,尽管外部情节的尺度提供确切的区域和尺寸放大。国土安全部网站传播(b)和RNA元素(d)左边的图所示。RNA元素要么是长小说intronic(深绿色)或长基因间(浅绿色)RNA。水平十字准线颜色相关的数据集d。c,转录因子主题传播实例地区受转录因子(橙色点)或在相应的主题匹配在灰色,绑定和释放点与一个箭头在每种情况下显示绑定网站通常更多的约束和更少的多样化。e派生的等位基因频率谱primate-specific元素,与外部编码元素变化黑色和红色的变化由编码元素。低频等位基因的增加相比,背景是指示性的负选择发生在变体编码注释的数据的集合。f、聚合哺乳动物约束分数的糖皮质激素受体(GR)转录因子在绑定网站主题,显示预期的主题相关的信息内容基地。这图的交互式版本可用的在线版本。
我DNase元素(图1 b)和绑定主题(图1 c),大多数的元素显示浓缩pan-mammalian约束和减少人口的多样性,尽管对一些细胞类型DNase我网站不似乎整体pan-mammalian约束。结合转录因子主题设置的有一种天然的控制转录因子主题以同样的顺序可能绑定但没有绑定的证据ChIP-seq实验所有情况下,结合主题显示更多的哺乳动物的约束和更高的抑制人类的多样性。
与先前的研究一致,我们不遵守全基因组的证据pan-mammalian选择新颖的RNA序列(图1 d)。也有大量的元素没有哺乳动物的约束,在17%和90%之间transcription-factor-binding地区以及国土安全部和做区域。先前的研究不能确定这些序列是活跃的生化反应,但是没有整体对生物体的影响,或者在lineage-specific选择。通过隔离优先序列插入到灵长类动物血统,这是唯一可行的全基因组规模的这些数据,我们能够专门研究这个问题。大多数primate-specific序列是由于逆转录转座子的活动,但是一个明显的比例非重复性的primate-specific序列。104343413年primate-specific基地(不含重复元素)、67769372年(65%)被发现在ENCODE-identified元素。检查227688个变异在这些primate-specific隔离区域显示所有类别的元素(RNA和监管)显示抑郁派生等位基因频率,符合最近的负选择发生在至少其中一些地区(图1 e)。另一种方法检查序列不清楚pan-mammalian约束下显示出相似的结果(l .病房和m >手稿提交)。这表明一个可观的比例所需的无约束元素lineage-specific元素生物的功能,符合长期最近进化的观点56,其余的可能是“中性”的元素2不是目前正在选择但仍可能影响细胞或大规模表型对健康没有影响。
转录因子的结合模式不统一,我们可以联系国米和intra-species措施负选择的主题的整体信息内容的位置。选择一些主题头寸高达蛋白质编码外显子(图1 f;l .病房和m . >手稿提交)。这些综合措施在图案显示绑定偏好人群中发现的网站也是一个网站相关的行为。通过开发一个网站的人口指标影响主题,我们发现高度约束绑定实例在哺乳动物能够缓冲个体变异的影响57。
编码数据集成与已知的基因特性
Promoter-anchored集成
许多编码分析直接或间接提供的信息促进剂的作用。关注的tss蛋白质编码记录,我们调查了不同的编码分析之间的关系,特别是测试假设RNA表达(输出)可以有效地预测模式的染色质修饰或转录因子绑定(输入)。与先前的报告相一致58,我们观察两个相对不同的启动子类型:(1)广泛,主要是(C + G)丰富,TATA-less推动者;(2)狭窄,TATA-box-containing推动者。组蛋白修饰这些启动子具有不同的模式,transcription-factor-binding网站选择性地富集在每个类(补充图1,部分Z)。
我们开发了预测模型探讨组蛋白修饰之间的交互和措施启动子的转录区分修改添加称为转录的结果(如H3K36me3和H3K79me2)和其他类别的组蛋白标记59。在我们的分析,最好的模型有两个组件:一个初始分类组件(开/关)和第二个定量模型组件。我们的模型显示,激活乙酰化标志(H3K27ac和H3K9ac)大致一样的激活甲基化标记(H3K4me3和H3K4me2) (图2一个)。虽然压抑的标志,如H3K27me3或H3K9me3,显示负相关个人和模型中,删除这些标记产生只有一小减少模型的性能。然而,对于每个细胞株启动子的一个子集,压抑的组蛋白标记(H3K27me3或H3K9me3)必须被用来预测他们的准确表达。我们还检查H3K79me2和H3K36me3标志之间的相互作用,这两个标记基因的身体,可能反映了招聘修饰酶的聚合酶亚型。如前所述,H3K79me2发生优先基因5′末端的身体和H3K36me3发生更多的3′,和我们的分析支持前面的模型H3K79me2 H3K36me3转变发生在第一个3′拼接的网站60。
一些先前的研究试图建立定性或定量模型的全基因组转录从转录因子水平,因为缺乏记录transcription-factor-binding地区和周围的缺乏协调一个细胞系。我们因此检查transcription-factor-binding信号的预测能力对启动子的表达水平(图2 b)。与组蛋白修饰的概要文件,大多数转录因子显示丰富绑定TSS附近的信号在一个狭窄的DNA区域,使用相对较高绑定信号启动子高CpG内容。大多数这种相关性可以通过查看总完成绑定没有特定的转录因子的转录因子。在一起,这些相关性模型表明,一组有限的染色质是都足以解释的转录,多种转录因子可能有广泛的角色通常在许多基因转录水平。重要的是要注意,这是一个天生的观察性研究的关联模式,符合各种机械模型和不同染色质之间的因果关系,转录因子和RNA化验。然而,它确实表明有足够的信息出现在基因的启动子区域来解释大部分的RNA表达的变化。
我们开发了预测模型用来模拟类似转录活动探索之间的关系水平的组蛋白修饰和外显子的交替在拼接记录。甚至占表达水平,H3K36me3有积极贡献外显子包容,而H3K79me2 (h . Tilgner负贡献等手稿做准备)。通过监测RNA数量在K562细胞的亚细胞分数,我们发现基本上所有拼接co-transcriptional61年,进一步支持染色质结构和连接之间的联系。
Transcription-factor-binding site-anchored集成
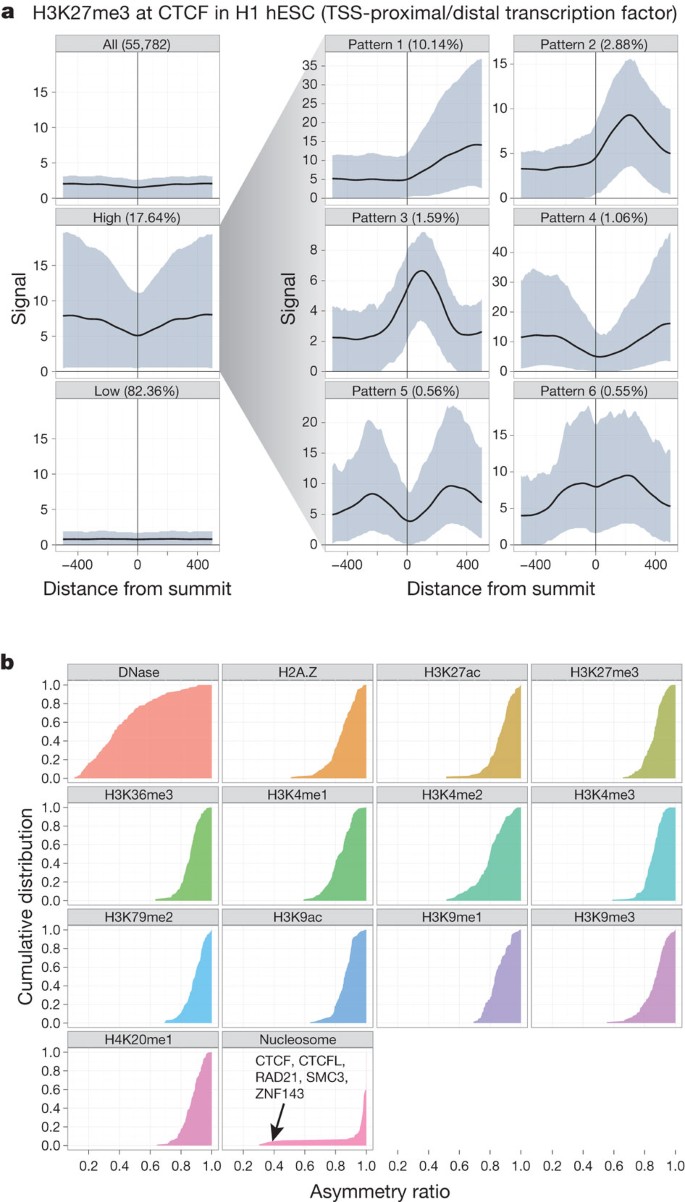
Transcription-factor-binding网站提供一个自然集中在探索染色质属性。转录因子通常是多功能的,可以结合各种基因位点的不同组合和染色质标记和核小体组织的模式。因此,而不是平均染色质马克概要文件在所有转录因子的结合位点,我们开发了一个聚类过程,称为集群聚合工具(CAGT),确定共享相似但不同的模式的结合位点子集染色质标记信号大小,形状和隐藏的方向性30.。例如,平均的压抑的组蛋白标记H3K27me3在H1为其所有55782 CTCF-binding网站显示信号差浓缩(图3)。然而,信号幅度分组资料后我们发现9840(17.6%)的一个子集CTCF-binding网站,表现出显著的侧翼H3K27me3信号。形状和方向分析进一步表明,主要的信号剖面H3K27me3 CTCF高峰会议是不对称的,符合一个边界的角色之间的一些CTCF网站活跃和polycomb-silenced域。提供了进一步的例子补充无花果5和6TAF1部分大肠的主要tss附近发现,转录的不对称网站面向的方向。然而,对于远端网站,比如那些受GATA1 CTCF,我们还观察到一个高比例的非对称组蛋白模式,虽然独立于主题方向性。事实上,所有transcription-factor-binding数据集在所有细胞系主要表现不对称模式(不对称比> 0.6)为所有染色质标记而不是DNase我信号(图3 b)。这表明大多数transcription-factor-bound染色质事件与结构化、定向的组蛋白修饰模式,启动子的方向并不是唯一来源定位在这些网站。
一个集群的结果聚合H3K27me3修改信号CTCF-binding网站(多功能蛋白质参与染色质结构)。前三个情节(左列)显示的信号行为组蛋白修饰在所有网站(上),然后分成高低信号组件。实线显示平均信号分布的相对位置与蓝色阴影区域限定第十和百分之九范围。高信号组件然后进一步分解成六个不同的形状类右侧(见参考。30.详情)。形状分解过程链。b总结DNase我的形状不对称,核小体组蛋白的化学修饰和信号通过绘制每个信号的不对称比所有transcription-factor-binding网站。所有组蛋白修饰主要以本研究显示不对称模式transcription-factor-binding网站。这图的交互式版本可用的在线版本。
我们还研究了核小体入住率相对于染色质标记的对称性transcription-factor-binding网站。tss左右,通常有强烈的非对称核小体入住率,通常占大多数的组蛋白修饰信号(例如,见补充图4,部分E)。然而,远离tss、更和谐。例如,CTCF-binding网站通常显示阵列定位准确的核小体两侧的高峰会议(补充图1部分E)62年。侧翼的染色质标记信号高、信号往往不对称,说明微分与组蛋白修饰标记(补充无花果2和3部分E),因此,我们在全基因组范围内确认转录因子可以形成壁垒的核小体和组蛋白修饰被安排在不同的配置62年,63年,64年,65年。这是参中进一步详细地探讨25,26和30.。
转录因子co-associations
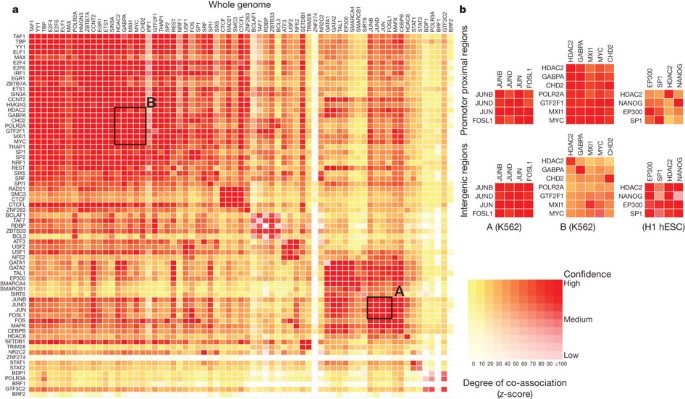
Transcription-factor-binding区域非随机分布在整个基因组,对其他功能(例如,启动子)和其他Transcription-factor-binding地区。在层1和2细胞系,我们发现3307对统计co-associated因素(P< 1×10−16GSC)涉及114(满分117分)的因素(97%)(图4)。这些包括预期关联,如小君和安全系数,和一些不太期望小说协会,如TCF7L2 HNF4-α和FOXA2 (ref。66年;给出了一个完整的清单补充表1,部分F)。当一个人认为单独启动子和基因间区域,这改变3201对(116因素,99%)启动子和1564对(108因素,92%)基因间区域,有一些关联更具体的对这些基因组上下文(例如,集群HDAC2, GABPA, CHD2, GTF2F1, MXI1 MYC基因启动子区域和SP1, EP300, HDAC2 NANOG在基因间区域(图4 b))。这些通用和上下文相关的协会导致的网络表示co-binding与许多有趣的特性,探讨了参考文献19,25和26。此外,我们还发现了一组区域受多种因素代表高入住率的转录因子(热)地区67年。
一个,重要的转录因子对co-associations使用GSC统计在K562细胞在整个基因组。颜色的强度代表协会的程度(从红色(强大),橙色,黄色(弱)),而颜色的深度代表了适合GSC20.模型(白色表明统计模型在哪里不合适)的关键。大多数转录因子与其他转录因子的联系,这些联系是依赖于基因组上下文,这意味着一旦基因组分为子近端和远端地区,co-association的总体水平下降,但更具体的关系了。b,三个类的行为。第一列显示一组关联的强度在启动子和远端地区独立的位置,而第二个列显示一组转录因子在promoter-proximal地区具有较强的关联。这些例子都是在K562细胞和突出显示数据在全基因组co-association矩阵(一个A和B)标签框,分别。第三列显示一组转录因子显示远端地区强大的协会(H1 hESC线)。这图的交互式版本可用的在线版本。
全基因组整合
识别功能基因组区域,我们下一个综合元素使用独立的基因组地标有识别力的训练方法,已知元素的一个子集的一个特定的类被用来训练模型被用来发现更多这类的实例,或者使用方法中只有数据编码化验使用没有任何注释的显性知识。
有识别力的培训中,我们使用一个三步过程预测潜在的增强剂,所描述的补充信息和裁判。67年。两种歧视模型聚集在一组∼13000年在K562细胞中假定的增强剂67年。在第二个方法中,两种方法论上不同的无偏的方法(见参考文献40,68年霍夫曼和m . m .et al。,准备手稿)聚集在一套整合的组蛋白修饰和chromatin-accessibility模式,可用于基因组片段的一级和二级细胞株,虽然单个位点在每个州每个细胞株是不同的。除了RNA聚合酶II和CTCF的转录因子数据没有显著改变这些模式。在这个阶段,我们故意排除RNA和甲基化分析,保留这些数据来验证分割。
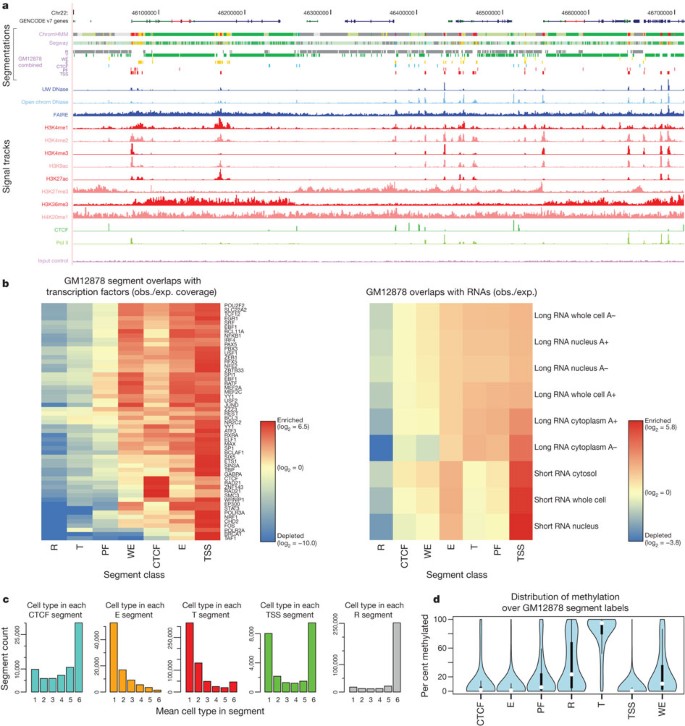
我们的集成的两种分割方法(m·m·霍夫曼et al。,在准备手稿)建立一套一致的七大类基因组,描述表3。标准的积极推动者,独特的核心启动子区域(TSS和PF状态),导致活跃基因的身体(T,转录状态),是重新发现了在这个模型(图5 a, b)。有三个“活跃”远端状态。我们暂时贴上两个作为增强剂(预测增强剂,E,并预测弱增强剂,我们)由于其发生地区开放的染色质H3K4me1高,尽管他们在H3K27ac等标志的水平不同,目前认为区分活跃的和不活跃的增强剂。其他活动状态(CTCF)高CTCF绑定,包括序列函数作为绝缘体在转染实验。剩下的压抑状态(R)总结序列分成不同类型的积极压抑或不活跃的,静止的染色质。我们发现在细胞类型CTCF-binding-associated状态是相对不变的,与个别地区经常占据CTCF的状态在所有六个细胞类型(图5度)。相反,E和T州有实质性的特异性行为,而TSS状态有一个双向行为具有类似数字cell-invariant和特异性的事件。重要的是要注意,共识总结类不捕获所有的细节中发现个人分割包含更多的州。
一个,说明地区两种分割方法(ChromHMM和赛格威)在一个密集的视图和组合分割显示每个州在GM12878细胞扩张,在一个压缩的GENCODE基因注释。注意,在这个级别的变焦和基因组浏览器分辨率,一些片段似乎尽管他们不重叠。分割类的名称和颜色方案表3。下分割显示每一个作为输入数据的归一化信号分割。开放的染色质信号DNase-seq华盛顿大学的集团(威斯康辛大学DNase)或编码开放染色质组织(Openchrom DNase)和做化验所示蓝色;信号从组蛋白修饰ChIP-seq红色;信号和转录因子ChIP-seq波尔二世与CTCF绿色。淡紫色ChIP-seq控制信号(输入控件)底部还包括作为输入分割。b,协会选择了转录因子(左)和RNA(右)中的元素组合分割状态(x轴)表示为一个观察/预期比率(obs. / exp。)为每个转录因子或RNA组合元素和分割类使用热图规模的关键除了每个热点图所示。c细胞系之间,变化的状态,显示事件的分布状态的六个细胞系基因组在特定位置:从独特的细胞系在所有六个细胞系无处不在的五个州(CTCF E T TSS和R)。d从rrb分布的甲基化水平在个人网站分析GM12878细胞在不同状态,显示预期hypomethylation tss和甲基化基因的身体状态(T)和压抑(R)的地区。
RNA的分布物种跨段截然不同,表明分段捕获潜在的生物活性。腺苷RNA基因体内大量丰富。在启动子周围,有短RNA物种之前确认为promoter-associated短RNA (图5 b)16,69年。同样,DNA甲基化显示显著区别部分,概括已知的主要生物学unmethylated积极推动者(TSS)其次是甲基化基因的身体42(T状态,图5 d)。两个enhancer-enriched状态显示不同的DNA甲基化模式,与不活跃增强器状态(通过H3K27ac / H3K4me1水平)显示高甲基化。这些国家也没有过多的RNA元素聚(A)反面methyl-cap RNA,化验的笼子序列,基因间的匹配控制相比,表明一个特定的转录模式与活性增强有关70年。转录因子也表现出不同的分布在段(图5 b)。惊人的模式是转录因子的浓度在TSS-associated状态。增强剂包含一组不同的转录因子。例如,在K562细胞,E为绑定状态是丰富的蛋白质编码的EP300,”丛书,FOSL1,GATA2,HDAC8,JUNB,JUND,NFE2,SMARCA4,SMARCB1,SIRT6和TAL1基因。我们测试了这些预测的一个子集增强剂在老鼠和鱼转基因模型(例子图6),超过一半的元素展示活动,通常在相应的组织类型。
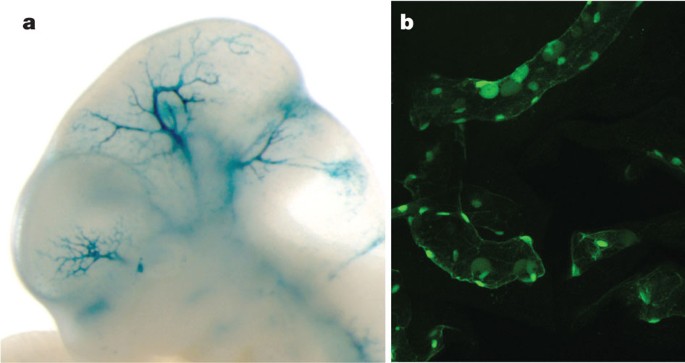
段(见随机抽样E状态表3)K562分割被克隆的鼠标,fish-based转基因增强剂化验。一个,代表LacZ-stained转基因胚胎天11.5 (E)小鼠胚胎获得构造hs2065 (GRCh37 EN167, chr10: 46052882 - 46055670)。高度可再生的染色观察血管中9的9胚胎产生的独立的转基因整合事件。b代表绿色荧光蛋白转基因记者青鳉鱼从基底构造hsp70启动子meganuclease-based转染。血液循环可再生的转基因表达的有核细胞和内皮细胞的墙被认为在81年从100年转基因测试的结构。
分割提供了一个线性确定整个基因组的功能状态,但不是一个特定的基因与远端区域协会。通过使用跨细胞系DNase我信号的变化,39%的E(增强剂相关的)州可能与提议的调节基因29日整合与地域邻近模式由5度决定的49或ChIA-PET。
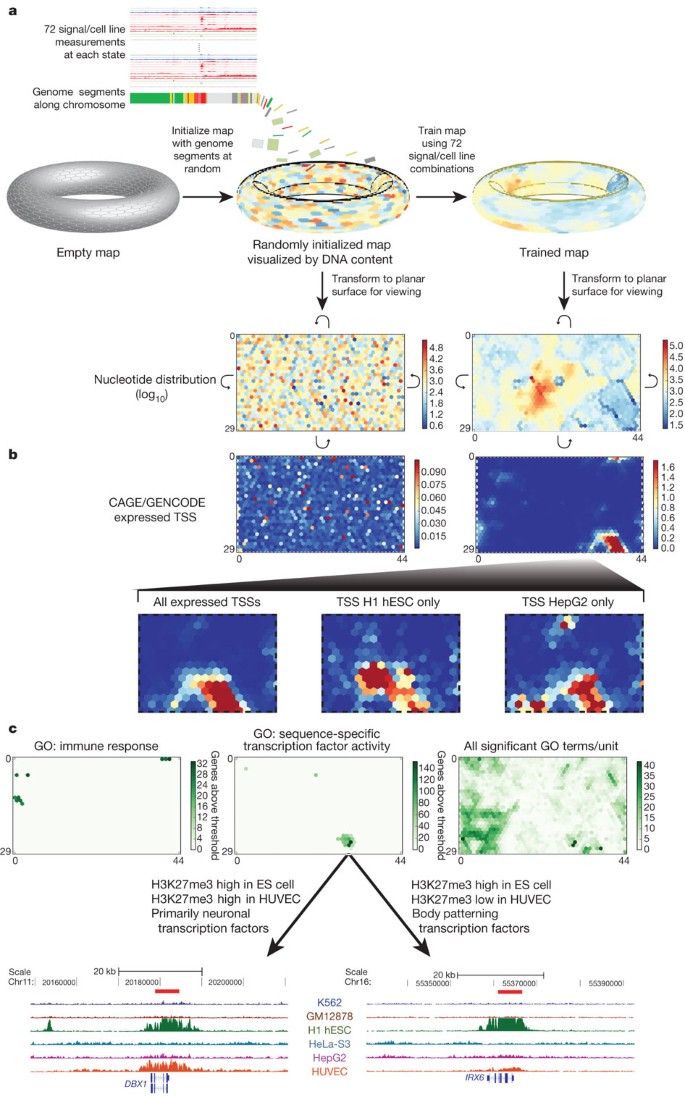
提供细粒度的区域分类,我们求助于自组织映射(SOM)集群基因组分割区域根据检测信号特征(图7)。分割区域最初随机分配到1350 -状态映射在一个二维环形空间(图7)。这张地图可以被可视化为二维矩形平面上可以绘制各种信号分布。例如,矩形的左下角图7显示了基因组的分布在初始随机地图。SOM被训练使用十二个不同ChIP-seq DNase-seq化验在之前6个细胞类型分析大规模分割(即超过72 -维空间)。训练后,SOM聚类又在二维可视化,现在显示的组织分布基因组片段(右下角的面板,图7)。单个数据集相关的基因组片段在每个SOM地图单位(六角形细胞)在同一框架可以可视化学习每个额外的数据分布在染色质状态的地图。图7 b显示笼/ TSS表达数据覆盖的随机初始化(左)和训练有素的地图(右)板。以这种方式训练有素的细胞类型特异的TSS SOM强调集群(底部面板图7 b),这表明有套组织相互区别的tss的微妙的组合编码染色质数据。许多ultra-fine-grained状态分类显示的SOM与特定的基因本体论(去)条款(右面板图7 c)。例如,左侧面板图7 c标识十SOM地图单位富含基因与基因组区域相关术语的免疫反应。中央面板标识一组不同的地图单位丰富的术语“sequence-specific转录因子的活动”。两个地图单位最丰富的词,表示最黑暗的绿色色素,含有基因高H3K27me3 H1为段,但在HUVECs H3K27me3水平不同。基因功能分析与本体工具(好了71年)显示,地图单位H3K27me3高水平在两种细胞类型丰富转录因子基因与已知的神经功能,而邻近的地图单元丰富体内基因参与模式。基因组浏览器底部的照片图7 c挑出一个例子地区的两个SOM地图单位说明H3K27me3信号的差异。总体来说,我们有228个不同的术语与特定领域跨一个或多个州(a . Mortazavi个人通信),并可以分配超过三分之一的基因注释单独的基础上其多细胞组蛋白模式。因此,SOM分析提供了一个细粒度的跨多个细胞染色质数据类型的映射,可以用来与染色质结构,其他数据类型以不同层次的分辨率(例如,单位的大型集群包含任何活跃的TSS subclusters由单位丰富的TSS活跃在只有一个细胞类型,或者个人地图单位显著富集为特定条款)。
一个- - - - - -cSOM的训练(一个)和分析的结果(b,c)所示。最初我们从ChromHMM任意放置基因组片段分割到环形地图表面,虽然SOM不使用ChromHMM国家分配(一个)。然后我们训练地图使用12种不同的信号ChIP-seq和六个细胞类型DNase-seq化验分析。每个单元的SOM表示由六角形细胞在一个平面的二维视图环形地图。弯曲的箭头表明,穿过二维视图的边缘带我们回到了相反的边缘。生成的地图可以覆盖任何类的编码或其他数据来查看数据的分布在这个高分辨率分割。在面板一个基因组的分布在未经训练的基地和训练地图(左和右,分别)所示为日志使用热图的颜色10值。btss的分布从笼实验GENCODE注释的平面表示的初始随机组织(左)或最后训练SOM(右)使用热量地图彩色根据附带的鳞片。的下半部分b扩大tss表达的不同分布的SOM(左)或tss专门表达细胞系两个例子中,H1 hESC(中心)和HepG2(右)。c基因本体论的协会(去)条款同样的训练有素的SOM在相同的表示。我们分配在20 kb基因的基因组片段SOM单位,单位,然后这组基因相关条款使用超几何分布在修正了多个测试。地图单位去显著相关术语的绿色,随着力量的颜色反映了越来越多的基因显著相关的条款去免疫反应(左)或sequence-specific转录因子的活动(中心)。在每种情况下,特定的SOM单位显示与这些术语。右边的面板显示了所有的分布在同一SOM显著相关条款,现在色彩的词数/ SOM单位。sequence-specific转录因子的活动,两个示例提取的基因组区域的底部面板c从邻近SOM单位。这些都是周围地区DBX1(从SOM单元26日,31岁的左面板)IRX630 (SOM单元27日,右面板)基因,分别,连同其H3K27me3 ChIP-seq信号的每个层1和2细胞类型。为DBX1,主要代表一组神经元与单位相关转录因子26日,31岁,有一个专制H3K27me3 H1为和HUVECs信号;为IRX6身体,代表一组模式相关的转录因子与SOM单位27日,30日,压抑的标志是主要局限于胚胎干细胞(ES)细胞。这图的交互式版本可用的在线版本。
这里给出的分类分析和细胞系研究必然是有限的,而且可能含有许多异构类的元素。尽管如此,健壮的分类,允许人类基因组的系统视图。
洞察人类基因组变异
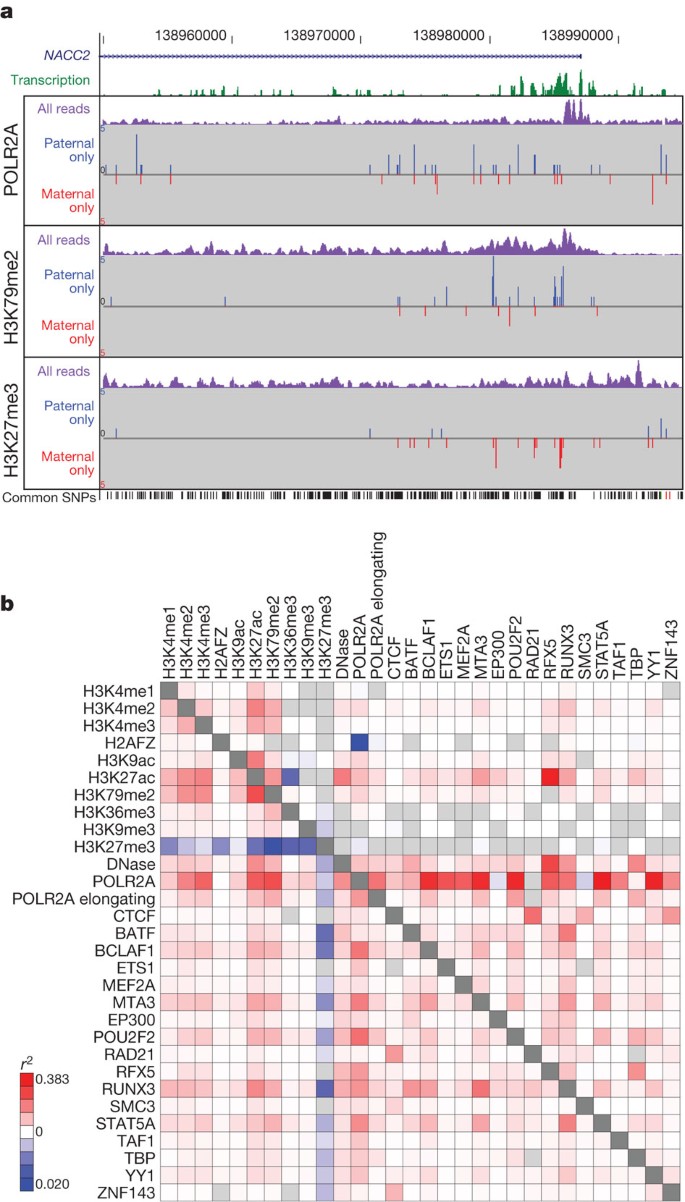
我们下一个序列变异的潜在影响进行了探讨编码功能元素。我们检查了allele-specific变异使用结果GM12878细胞来源于个体(NA12878) 1000基因组测序项目,连同她的父母。因为编码分析主要是基于三个设计允许每个GM12878数据集除以杂合的地点具体的贡献,产生聚合haplotypic信号从多个基因组网站。我们检查了193个编码化验allele-specific偏见使用1409992分阶段,杂合的SNPs和167096插入/删除(indels) (图8)。定位偏差对等位基因出现在参考基因组序列是避免使用一个序列在NA12878专门量身定做的变异和单体型(“个性化基因组”)72年。我们发现优惠的实例绑定到每个父母等位基因。例如,比较的结果POLR2A, H3K79me2和H3K27me3化验NACC2(图8)展示了一个强大的父亲的偏见H3K79me2 H3K27me3 POL2RA和强烈的母性偏见,表明微分父系和母系等位基因的活动。
一个,代表从GM12878细胞allele-specific信息选择化验的第一外显子NACC2基因(GRCh37 Chr9基因组区域:138950000 - 138995000)。转录信号以绿色显示,三个部分显示allele-specific数据三个数据集(POLR2A, H3K79me2 H3K27me3 ChIP-seq)。在每种情况下的紫色信号处理信号序列分析读取,而蓝色和红色信号显示顺序读取专门分配给父亲或母亲基因组的副本,分别。从dbSNP常见snp的集合,包括阶段性、杂合的snp用于提供作业,面板的底部所示。NACC2POLR2A有统计学意义的偏见和马克H3K79me2 transcription-associated和有很大的孕产妇专制马克H3K27me3偏见。b在单个基因,双向allele-specific信号的相关性(对角线以下)或个人ChromHMM段在整个基因组内选择DNase-seq和组蛋白修饰和转录因子ChIP-seq化验。相关性是彩色的程度根据热图表示从规模正相关(红色)到anti-correlation(蓝色)。这图的交互式版本可用的在线版本。
图8 b显示选择的相关性allele-specific信号在整个基因组。例如,我们发现一个强大的等位基因之间的相关性POL2RA BCLAF1绑定,以及H3K79me2和H3K27me3之间呈负相关,在基因(图8 b,低于对角线,左下)和染色体片段(右上角)。总的来说,我们发现积极的等位基因的相关性在193编码分析比负相关性更强、更频繁。这可能是由于优先捕获的等位基因和/或特定的组蛋白修饰和转录因子,分析在工程中使用。
罕见变异,个人基因组和体细胞变异
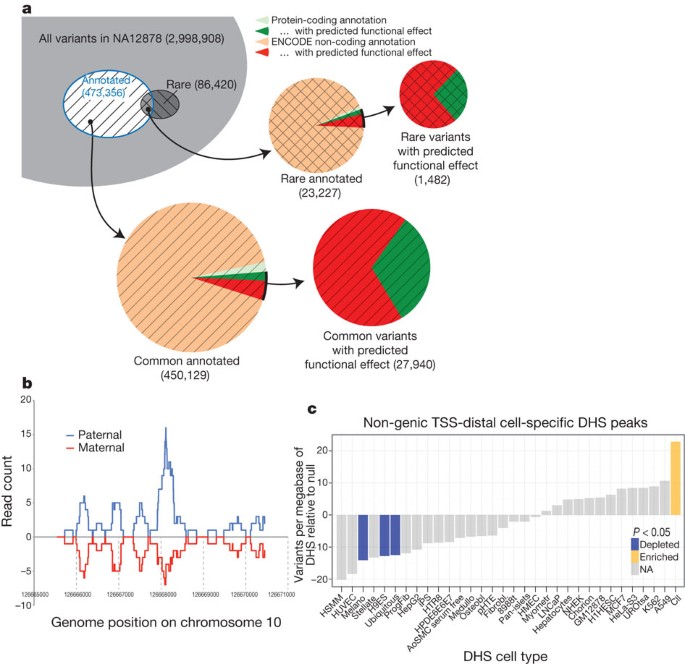
我们进一步研究了个体变异的潜在功能影响的环境中编码注释。我们NA12878变异分为常见和罕见的类,和这些这些重叠分区编码注释(图9和补充表1和2,部分K)。我们也预测潜在的功能作用:蛋白质编码基因,这些非同义snp或变异可能引起损失由框移函数,过早停止,或剪切位点中断;为其他地区,这些变异重叠transcription-factor-binding站点。我们发现类似数量的潜在功能变异影响蛋白质编码基因或影响其他编码注释,表明许多功能变体在个人基因组蛋白质编码基因的外显子之外。更详细的分析监管变体注释中描述ref。73年。
一个,分解一个基因组的变异(NA12878)的频率(常见或罕见的(也就是说,变异的179人没有出现在信号低测序飞行员1 1000人基因工程的欧洲面板55))和编码注释,包括蛋白质编码基因和非编码元素(GENCODE注释蛋白质编码基因,假基因和其他ncRNAs,以及从ChIP-seq transcription-factor-binding网站数据集,不包括广泛的注释(如组蛋白修饰、分割和RNA-seq)。注释状态由预测功能的效果,进一步细分为蛋白质编码区域非同义和错义突变和变异重叠转录因子为非编码元素图案注释。大部分变异注释具有预测功能的影响在非编码类别。b相对罕见事件之一,对齐到一个个人基因组序列(父亲和母亲板)显示不同的参考基因组的读出。在这种情况下,paternal-haplotype-specific CTCF峰值识别。c,从全基因组水平的体细胞变异发生在国土安全部的黑色素瘤样例独特不同的细胞系。彩色条显示的情况下大大丰富或压制的体细胞突变。编码的细节可以发现细胞类型http://encodeproject.org/ENCODE/cellTypes.html。这图的交互式版本可用的在线版本。
进一步研究NA12878基因组变异的潜在影响transcription-factor-binding地区,我们进行峰值使用个人二倍体基因组序列构造呼吁NA12878 (ref。72年)。我们从GM12878 ChIP-seq对齐序列分别对父系和母系的单。正如所料,更大一部分读取比参考基因组(见对齐补充信息,补充图1部分K),平均大约1%的transcription-factor-binding网站GM12878 haplotype-specific时尚细胞检测。例如,图9 bCTCF-binding网站显示没有检测到使用的参考序列,只是现在的单体型由于1-bp删除(见也补充图2,部分K)。DNA测序进一步降低成本,优化分析ENCODE-type数据应该使用个体或细胞的基因组序列分析。
大多数癌症基因组的分析到目前为止都聚焦于体细胞变异蛋白质编码区域的特征。我们交叉四个可用癌症全基因组数据集与编码注释(图9 c和补充图2,部分L)。总的来说,体细胞变异是相对减少编码注释区域,特别是对于特定细胞类型的元素匹配的假定的肿瘤来源(例如,皮肤黑色素细胞黑色素瘤)。检查在内含子元素的突变谱strand-specific突变情况下的任务可以显示有突变谱差异国土安全部和未经地区(0.06确切概率法,补充图3,部分L)。体细胞突变的抑制是符合这些元素在肿瘤细胞内的重要功能角色,强调潜在的替代组检查癌症的目标。
常见变异与疾病有关
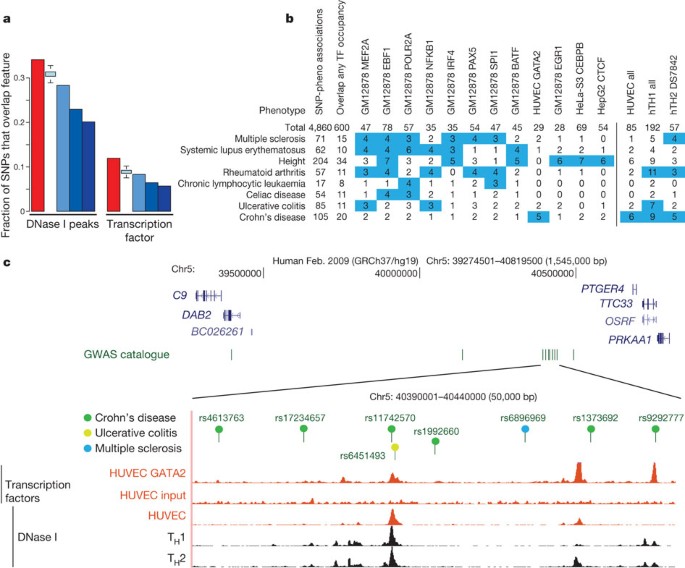
近年来,GWAS极大地扩展我们的知识与人类疾病相关的基因位点的风险和其他表型。这些研究是一系列的输出单核苷酸多态性(GWAS SNPs)与表型相关,虽然不一定是功能变体。值得注意的是,88%的单核苷酸多态性intronic或基因间的关联74年。我们检查了4860 SNP-phenotype协会4492个snp策划国家人类基因组研究所(NHGRI) GWAS目录74年。我们发现12%的这些snp transcription-factor-occupied重叠区域而34%重叠国土安全部(图10)。两个数字反映重要的充实与1000人基因工程snp的整体比例(分别为6%和23%)。即使占偏差引入了snp的选择标准的基因序列,GWAS snp显示持续较高的重叠编码注释(图10,请参阅补充信息)。此外,密度分区后基因组的不同类型的功能元素,GWAS snp都丰富除了function-rich分区中的所有基因单核苷酸多态性,并在function-poor分区(见减少补充图1,部分M)。GWAS snp特别丰富的分割类与增强子和tss在几个细胞类型(见补充图2,部分M)。
一个,重叠的铅SNP NHGRI GWAS SNP目录(2011年6月)和国土安全部(左)或transcription-factor-binding网站(右)红酒吧与各种控制SNP在蓝色。控制SNP集(从左到右):单核苷酸多态性的Illumina公司2.5芯片为例,一种广泛使用的GWAS SNP输入面板;从1000人基因工程单核苷酸多态性;24个人基因组snp提取(见个人基因组变异跟踪http://main.genome-browser.bx.psu.edu(ref。80年),所有显示为蓝色的酒吧。此外,进一步控制使用1000随机的基因SNP面板中,匹配的单核苷酸多态性与每个NHGRI目录SNP等位基因频率和距离最近的TSS(浅蓝色酒吧边界四分位范围)的1.5倍。国土安全部和transcription-factor-binding地区,更大比例的重叠GWAS-implicated snp发现相比于任何控件集。b,总重叠的表型选择transcription-factor-binding网站(左矩阵)或国土安全部在选定的细胞系(右矩阵),计算表型之间的重叠和细胞系/因素。值在蓝色方块通过实证P值阈值≤0.01(基于相同的分析随机选择之间的重叠,GWAS-matched SNPs和这些表观遗传特性),至少有三个重叠的一个计数。的P值的总数phenotype-transcription关联的因素是< 0.001。c,几个单核苷酸多态性与克罗恩病相关基因和其他炎性疾病,驻留在一个大沙漠5号染色体上,连同一些表观遗传特性的函数的说明。苏格兰民族党(rs11742570)密切相关,克罗恩病重叠GATA2 transcription-factor-binding HUVECs信号决定。这个区域也是DNase我在HUVECs高度敏感,辅助TH1和TH2细胞。这图的交互式版本可用的在线版本。
检查综合编码注释的SOM(见上图),我们发现19 SOM地图单位显示重要的浓缩GWAS snp,包括许多SOM单位之前与特定的基因功能,如免疫反应区域。因此,相当大比例的单核苷酸多态性中确定初始GWAS扫描功能或躺在编码注释的长度(∼平均500个基点),代表合理的候选功能变体。扩大的可行集功能性单核苷酸多态性与合理的连锁不平衡,多达71%的GWAS SNP有潜在病因SNP重叠DNase我网站,和31%的SNP位点有候选人,重叠一个转录因子结合位点被(参见参考文献73年,75年)。
GWAS目录提供了丰富的功能分类的精确的表型被研究。这些表型分类与编码注释和非随机相关有显著的对应关系之间的表型和细胞类型的身份或转录因子编码中使用试验(图10 b)。例如,五个单核苷酸多态性与克罗恩病相关重叠GATA2-binding网站(P价值0.003通过随机排列或0.001 GWAS-matched snp通过实证方法比较;看到补充信息),14个位于国土安全部相关免疫细胞类型中找到。5号染色体上的一个著名的例子是一个基因沙漠p13.1包含八个单核苷酸多态性与炎症性疾病相关。数接近或在国土安全部辅助1型(TH1)和TH2细胞以及峰值的绑定HUVECs转录因子(图10 c)。后者细胞系并不是免疫,但入住率因素检测到可能会有一个代理绑定的一个更相关的因素,如GATA3、T细胞。也在这一地区影响表达水平的遗传变异PTGER4(ref。76年),EP4前列腺素受体编码。因此,编码数据加强假设基因变异在5 p13.1调节侧翼基因的表达,而且提供的特定假设一个叫因素的变异影响入住率allele-specific方式,从而影响对克罗恩病的易感性。
与编码细胞表型的联系加强了论点,至少一些GWAS铅snp功能或者非常接近功能变体。每个铅SNP和编码注释之间的联系仍然是一个可靠的假设一个特定的功能元素类或细胞类型和未来实验探索。补充表1 - 3、14885节,列出所有成对协会在整个编码注释。附带的文件有一个更详细的检查常见变异与其他监管信息19,25,29日,73年,75年,77年。
结束语
功能元素的前所未有的数量确定在本研究提供了一个宝贵的资源,科学界以及显著增强我们对人类基因组的理解。我们的分析揭示了许多小说方面的基因表达和调控等的组织信息,附带插图的论文(见http://www.encodeproject.org/ENCODE/pubs.html收集编码出版物)。然而,仍然有许多具体细节,特别是关于机械的过程,生成这些元素和他们怎么没功能,这需要额外的实验来说明。
报道的大传播我们的最高分辨率,最保守的基地与GENCODE蛋白质编码基因外显子(2.9%)或特定的DNA结合蛋白(8.5%)最广泛,最一般的标志覆盖基因组(大约80%),与许多层次between-presents光谱发现的元素具有不同的功能特性的编码。总共99%的基因组中已知的基地是在1.7 kb的编码元素,而95%的基地是在结合转录因子8 kb的主题或DNase我足迹。有趣的是,即使使用最保守的估计,基地的分数可能参与直接的基因调控,虽然不完整,显著高于归因于蛋白质编码外显子(1.2%),提高的可能性更多信息在人类基因组中基因调控的可能是重要的比生化功能。许多监管元素并不限制在哺乳动物进化,而到目前为止最可靠的迹象之一的重要生化事件有机体。因此,我们的数据提供了同源指标暗示可能的功能元素。
第一次重要的是,我们有足够的统计能力评估的影响- primate-specific元素上的选择,和所有编码类显示负选择这些unique-to-primate元素的证据。此外,即使最保守的估计的功能元素(假定的DNA /蛋白结合区域的8.5%),假设我们已经取样的一半元素从转录因子和程控多样性,有人估计,至少20%(17%来自蛋白质绑定和2.9%的蛋白质编码基因外显子)基因组的参与这些特定功能,可能图更高。
编码注释的广泛覆盖提高了我们对常见疾病有遗传因素的理解,罕见的遗传疾病,癌症,如图所示,我们链接的能力否则匿名协会功能元素。编码和类似的研究提供一个第一步解释其他genome-beyond蛋白质编码genes-thereby增加常见疾病基因研究和可测试的假设。这些信息证明执行全基因组测序(而非外显子组,1.2%的基因组)罕见疾病和调查体细胞变异在非编码的功能元素,例如,在癌症。此外,GWAS分析通常将疾病snp在很大区域,比较编码非编码的功能元素可以帮助确定假定的因果变异除了精致精细定位位置的技术78年。结合与allele-specific编码数据信息来源于个人基因组序列提供了特定的有关基因变异的影响。事实上,我们相信,一个重要的目标是使用功能等数据,来自这个项目分配每个基因变异可能影响人类的表型。
到目前为止,编码抽样119 1800种已知的转录的转录因子和通用组件机械有限数量的细胞类型,和13的60多个目前已知的组蛋白或DNA在147细胞类型的修改。DNase我做和广泛的RNA化验在亚细胞分离一直在进行许多细胞类型,但总体来说这些数据反映出一个小比例的潜在功能在人类基因组中编码的信息。未来一个重要的目标将是扩大这个数据集附加因素,修改和细胞,补充这一领域的其他相关项目(例如,表观基因学的项目,http://www.roadmapepigenomics.org/人类表观基因组的国际财团,http://www.ihec-epigenomes.org/)。这些项目将为人类基因组构成的基础资源,允许更深入的解释基因的组织和监管信息和监管的机制,从而为人类健康和疾病提供重要的见解。可以在线探索通过ENCODE-related联合发表论文自然编码explorer (http://www.nature.com/ENCODE),一个特别设计的可视化工具,允许用户访问相关论文和研究主题,讨论了多个文件通过主题组织的线程。
方法总结
全部细节的方法,请参阅补充信息。
引用
-
1
编码项目财团。编码(百科全书的DNA元素)项目。科学306年636 - 640 (2004)
-
2
伯尼,大肠et al。识别和分析的功能元素1%的人类基因组编码的试点项目。自然447年799 - 816 (2007)
-
3
编码项目财团。用户指南DNA序列元素的百科全书(编码)。公共科学图书馆杂志。9e1001046 (2011)
-
4
老鼠基因组测序协作组。初始的小鼠基因组测序和比较分析。自然420年520 - 562 (2002)
-
5
Chiaromonte, f . et al .人类基因组DNA的分享下选择估计从human-mouse基因组比对。冷泉哈布。计算机协会。定量,杂志。68年245 - 254 (2003)
-
6
库珀、通用汽车等分布和强度的约束在哺乳动物基因组序列。基因组Res。15901 - 913 (2005)
-
7
帕克,s . C。汉森,L。,Abaan, H. O., Tullius, T. D. & Margulies, E. H. Local DNA topography correlates with functional noncoding regions of the human genome.科学324年389 - 392 (2009)
-
8
Lindblad-Toh, k等。高分辨率的地图使用29哺乳动物人类进化的约束。自然478年476 - 482 (2011)
-
9
野鸡,m & Mattick j . s .提高人类序列估计的功能。基因组Res。171245 - 1253 (2007)
-
10
桥,c·p & Hardison r c功能人类基因组的一部分是什么?基因组Res。211769 - 1776 (2011)
-
11
就是为了,s等。广泛分布的非编码净化选择在人类基因组中。Proc。《科学。美国104年12410 - 12415 (2007)
-
12
Landt编著,s . g . et al . ChIP-seq modENCODE编码和财团所使用的指导原则和实践。基因组Res。http://dx.doi.org/10.1101/gr.136184.111(2012)
-
13
李,问。,Brown, J. B., Huang, H. & Bickel, P. J. Measuring reproducibility of high-throughput experiments.安。达成。统计。51752 - 1779 (2011)
-
14
哈罗,j . et al。GENCODE:人类基因组注释编码项目的引用。基因组Res。http://dx.doi.org/10.1101/gr.135350.111(2012)
-
15
Howald, c . et al。结合RT-PCR-seq和RNA-seq目录编码在人类基因组中基因的所有元素。基因组Res。http://dx.doi.org/10.1101/gr.134478.111(2012)
-
16
Djebali, s . et al .景观在人类细胞中转录。自然http://dx.doi.org/10.1038/nature11233(这个问题)
-
17
Derrien, t . et al。人类长非编码rna的GENCODE v7目录:分析他们的基因结构、进化,和表达式。基因组Res。http://dx.doi.org/10.1101/gr.132159.111(2012)
-
18
裴,b . et al . GENCODE假基因资源。基因组医学杂志。13R51 (2012)
-
19
格斯坦,m . b . et al .架构来源于人类的监管网络编码数据。自然http://dx.doi.org/10.1038/nature11245(这个问题)
-
20.
Bickel, p . J。博伊尔,N。,Brown, J. B., Huang, H. Y. & Zhang, N. R. Subsampling methods for genomic inference.安。达成。统计。41660 - 1697 (2010)
-
21
卡普兰,t . et al .定量模型的机制,控制全基因组转录因子结合在早期的模式果蝇发展。公共科学图书馆麝猫。7e1001290 (2011)
-
22
李x y . et al .染色质的作用指导广泛的可访问性,重叠的模式果蝇转录因子绑定。基因组医学杂志。12R34 (2011)
-
23
Pique-Regi, r . et al .准确推断转录因子结合的DNA序列和染色质易访问性数据。基因组Res。21447 - 455 (2011)
-
24
张,y et al .初级序列和表观遗传因素在活的有机体内入住率GATA1的基因组DNA。核酸Res。377024 - 7038 (2009)
-
25
Neph, s . et al。人类监管词汇编码转录因子的脚印。自然http://dx.doi.org/10.1038/nature11212(这个问题)
-
26
Whitfield, t . w . et al .功能分析人类启动子的转录因子结合位点。基因组医学杂志。13R50 (2012)
-
27
Garrard总值,d s &, w . t .核酸酶在染色质高度敏感网站。为基础。学生物化学启。57159 - 197 (1988)
-
28
莫斯科f . d .染色质重塑作为指导在哺乳动物转录调控网络。j .细胞。物化学。88年684 - 694 (2003)
-
29日
瑟曼,r . e . et al。访问染色质景观的人类基因组。自然http://dx.doi.org/10.1038/nature11232(这个问题)
-
30.
Kundaje, a . et al .无处不在的染色质环境的异质性和不对称监管元素。基因组Res。http://dx.doi.org/10.1101/gr.136366.111(2012)
-
31日
舒尔茨,d . C。,Ayyanathan, K., Negorev, D., Maul, G. G. & Rauscher, F. J., III SETDB1: a novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins.Dev的基因。16919 - 932 (2002)
-
32
Frietze, S。,O’Geen, H., Blahnik, K. R., Jin, V. X. & Farnham, P. J. ZNF274 recruits the histone methyltransferase SETDB1 to the 3′ ends of ZNF genes.《公共科学图书馆•综合》5e15082 (2010)
-
33
博伊尔,a . p . et al .高分辨率全基因组在活的有机体内不同的转录因子在人类细胞的碳足迹。基因组Res。21456 - 464 (2011)
-
34
Hesselberth, j . r . et al .全局映射protein-DNA交互在活的有机体内通过数字基因组的碳足迹。自然方法6283 - 289 (2009)
-
35
张y等。基于模型分析ChIP-Seq (mac)。基因组医学杂志。9R137 (2008)
-
36
Kouzarides, t .染色质修饰和它们的功能。细胞128年693 - 705 (2007)
-
37
李,B。,Carey, M. & Workman, J. L. The role of chromatin during transcription.细胞128年707 - 719 (2007)
-
38
亲爱的,g . C。,H一个wkins, R. D. & Ren, B. Predictive chromatin signatures in the mammalian genome.嗡嗡声。摩尔,麝猫。18R195-R201 (2009)
-
39
周,诉W。,Goren, A. & Bernstein, B. E. Charting histone modifications and the functional organization of mammalian genomes.自然启麝猫。12,7 - 18 (2011)
-
40
恩斯特,j . et al .染色质状态的映射和分析动力学在9个人类细胞类型。自然473年43-49 (2011)
-
41
亲爱的,G。,Wang, W. & Ren, B. Discovery and annotation of functional chromatin signatures in the human genome.公共科学图书馆第一版。医学杂志。5e1000566 (2009)
-
42
球,m . p . et al。目标和公司战略揭示gene-body甲基化签名在人类细胞。生物科技自然》。27361 - 368 (2009)
-
43
迈斯纳,a等。公司DNA甲基化的地图多能和分化细胞。自然454年766 - 770 (2008)
-
44
Ogryzko, V V。,Schiltz, R. L., Russanova, V., Howard, B. H. & Nakatani, Y. The transcriptional coactivators p300 and CBP are histone acetyltransferases.细胞87年953 - 959 (1996)
-
45
李斯特,r . et al。人类DNA methylomes基地分辨率显示广泛的外遗传性差异。自然462年315 - 322 (2009)
-
46
德克,j .基因调控在第三维度。科学319年1793 - 1794 (2008)
-
47
Dostie, j . et al .染色体构象捕获碳副本(5 c):一个大规模并行解决方案映射基因组之间的交互元素。基因组Res。161299 - 1309 (2006)
-
48
Lajoie, b R。,van Berkum, N. L., Sanyal, A. & Dekker, J. My5C: web tools for chromosome conformation capture studies.自然方法6690 - 691 (2009)
-
49
Sanyal,。,Lajoie, B., Jain, G. & Dekker, J. The long-range interaction landscape of gene promoters.自然http://dx.doi.org/10.1038/nature11279(这个问题)
-
50
Fullwood, m . j . et al。一个人类染色质interactome oestrogen-receptor-alpha-bound。自然462年58 - 64 (2009)
-
51
李,g . et al .广泛promoter-centered染色质交互提供一个拓扑转录调控的依据。细胞148年84 - 98 (2012)
-
52
Borneman, a . r . et al .散度相关的转录因子结合位点在酵母的物种。科学317年815 - 819 (2007)
-
53
奥多姆,d . t . et al .组织转录调控人类和小鼠之间的显著差异。自然麝猫。39730 - 732 (2007)
-
54
施密特,d . et al . Five-vertebrate ChIP-seq揭示转录因子的进化动态绑定。科学328年1036 - 1040 (2010)
-
55
从人口规模的测序人类基因组变异的地图。自然467年1061 - 1073 (2010)
-
56
国王,m . c & Wilson, a . c .进化在人类和黑猩猩的两个层次。科学188年107 - 116 (1975)
-
57
Spivakov, m . et al。分析转录因子结合位点的变异果蝇和人类。基因组医学杂志。13R49 (2012)
-
58
Sandelin, a . et al。哺乳动物的RNA聚合酶II核心启动子:从全基因组研究的见解。自然启麝猫。8424 - 436 (2007)
-
59
盾,x等。建模基因表达在各种细胞上下文中使用染色质特性。基因组医学杂志。13R53 (2012)
-
60
发怒,j . T。,Plocik, A. M., Guthrie, C. & Yamamoto, K. R. Reciprocal intronic and exonic histone modification regions in humans.自然结构。摩尔。杂志。171495 - 1499 (2010)
-
61年
Tilgner, h . et al .深度测序的亚细胞RNA分数显示拼接主要co-transcriptional在人类基因组中但lncRNAs效率低下。基因组Res。http://dx.doi.org/10.1101/gr.134445.111(2012)
-
62年
傅,Y。,Sinha, M., Peterson, C. L. & Weng, Z. The insulator binding protein CTCF positions 20 nucleosomes around its binding sites across the human genome.公共科学图书馆麝猫。4e1000138 (2008)
-
63年
科恩伯格,r . d . &路博,l .统计分布的核小体:非随机随机位置的机制。核酸Res。166677 - 6690 (1988)
-
64年
周末,d . e . et al .核小体定位在人类基因组的动态监管。细胞132年887 - 898 (2008)
-
65年
Valouev, a . et al,核小体组织在人类细胞主要的决定因素。自然474年516 - 520 (2011)
-
66年
Frietze, et al。细胞特定类型绑定模式显示,TCF7L2可以拴在基因组与GATA3协会。基因组医学杂志。13R52 (2012)
-
67年
Yip刘贤等。人类基因组区域分类的基础上根据模拟结合位点100多transcription-related因素。基因组医学杂志。13R48 (2012)
-
68年
霍夫曼,m . m . et al .无监督模式在人类发现通过基因组染色质结构分割。自然方法9473 - 476 (2012)
-
69年
Kapranov, p . et al . RNA地图揭示新的RNA类和一个可能的转录功能普遍。科学316年1484 - 1488 (2007)
-
70年
科赫,f . et al .转录起始平台和GTF招聘组织增强剂和促进剂。自然结构。摩尔。杂志。18956 - 963 (2011)
-
71年
麦克莱恩,c . y . et al .伟大的改善功能的解释基因区域。生物科技自然》。28495 - 501 (2010)
-
72年
Rozowsky j . et al . AlleleSeq:分析allele-specific表达式和绑定在一个网络框架。摩尔。系统。医学杂志。7522 (2011)
-
73年
博伊尔,a . p . et al .注释功能使用RegulomeDB个人基因组的变化。基因组Res。http://dx.doi.org/10.1101/gr.137323.112(2012)
-
74年
Hindorff,洛杉矶等。潜在的病原学的和功能影响人类疾病的全基因组关联位点和特征。Proc。《科学。美国106年9362 - 9367 (2009)
-
75年
肖布,m·A。,Boyle, A. P., Kundaje, A., Batzoglou, S. & Snyder, M. Linking disease associations with regulatory information in the human genome.基因组Res。http://dx.doi.org/10.1101/gr.136127.111(2012)
-
76年
Libioulle, c . et al .小说克罗恩病轨迹被全基因组关联映射到一个基因沙漠p13.1 5日和调节PTGER4的表情。公共科学图书馆麝猫。3e58 (2007)
-
77年
Vernot et al。个人和人口基因组学人类的监管变化。基因组Res。http://dx.doi.org/10.1101/gr.134890.111(2012)
-
78年
Harismendy, o . et al . 9 p21基因变异与冠状动脉疾病损害interferon-γ信号响应。自然470年264 - 268 (2011)
-
79年
程,et al。理解转录调控转录因子绑定数据的综合分析。基因组Res。http://dx.doi.org/10.1101/gr.136838.111(2012)
-
80年
舒斯特尔,s . c . et al。完成(并从南部非洲班图人的基因组。自然463年943 - 947 (2010)
确认
我们感谢我们实验室的其他成员和机构促成了这个项目的实验和分析组件。我们感谢d . Leja援助与生产数据。的财团资助NHGRI如下:生产补助:U54HG004570 (b . e . Bernstein);U01HG004695 (e .伯尼);U54HG004563 (g . e . Crawford);U54HG004557 (t . r . Gingeras);U54HG004555 (t·j·哈伯德);U41HG004568 (w·j·肯特);U54HG004576 (r . m . Myers);U54HG004558 (m . Snyder); U54HG004592 (J. A. Stamatoyannopoulos). Pilot grants: R01HG003143 (J. Dekker); RC2HG005591 and R01HG003700 (M. C. Giddings); R01HG004456-03 (Y. Ruan); U01HG004571 (S. A. Tenenbaum); U01HG004561 (Z. Weng); RC2HG005679 (K. P. White). This project was supported in part by American Recovery and Reinvestment Act (ARRA) funds from the NHGRI through grants U54HG004570, U54HG004563, U41HG004568, U54HG004592, R01HG003143, RC2HG005591, R01HG003541, U01HG004561, RC2HG005679 and R01HG003988 (L. Pennacchio). In addition, work from NHGRI Groups was supported by the Intramural Research Program of the NHGRI (L. Elnitski, ZIAHG200323; E. H. Margulies, ZIAHG200341). Research in the Pennachio laboratory was performed at Lawrence Berkeley National Laboratory and at the United States Department of Energy Joint Genome Institute, Department of Energy Contract DE-AC02-05CH11231, University of California.
作者信息
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
补充信息
补充信息1
这个文件包含补充文本和数据、方法和引用——查看详细内容列表。(PDF 1875 kb)
补充表第一章U
这个数据5显示了GENCODE基因注释的统计数据。(XLS 20 kb)
41586 _2012_bfnature11247_moesm288_esm.mov
这个视频展示了一块人类DNA的瞬时表达式将青鳉鱼胚胎的增强剂。预测来自人类细胞系k562,红细胞前体派生系。青鳉的表达式是在成熟的红细胞(这些是有核细胞在鱼)。表达绿色荧光蛋白。视频是在Wittbrodt海德堡大学的实验室。(MOV 930 kb)
补充电影1
这个视频展示了一块人类DNA的瞬时表达式将青鳉鱼胚胎的增强剂。预测来自人类细胞系k562,红细胞前体派生系。青鳉的表达式是在成熟的红细胞(这些是有核细胞在鱼)。表达绿色荧光蛋白。视频是在Wittbrodt海德堡大学的实验室。(MOV 930 kb)
41586 _2012_bfnature11247_moesm289_esm.mov
这个视频展示了一块人类DNA的瞬时表达式将青鳉鱼胚胎的增强剂。预测来自人类细胞系k562,红细胞前体派生系。青鳉的表达式是在成熟的红细胞(这些是有核细胞在鱼)。表达绿色荧光蛋白。视频是在Wittbrodt海德堡大学的实验室。(MOV 637 kb)
补充电影2
这个视频展示了一块人类DNA的瞬时表达式将青鳉鱼胚胎的增强剂。预测来自人类细胞系k562,红细胞前体派生系。青鳉的表达式是在成熟的红细胞(这些是有核细胞在鱼)。表达绿色荧光蛋白。视频是在Wittbrodt海德堡大学的实验室。(MOV 637 kb)
补充信息2
这个文件包含E1-E6补充数据,k1 k2, L1-L3, M1-M2, R1-R2, Y1和Z1,补充表E1-E2 k1 k2和L1和额外的引用。(PDF 2511 kb)
补充表第一章F
这个数据文件显示了TF公司联系。(XLS 2170 kb)
补充表第一章米
这个数据文件显示GWAS SNP表型关联在TF和国土安全部编码注释。(XLS 609 kb)
补充表2-section米
这个文件包含GWAS SNP对智慧协会在国土安全部编码注释。(TXT 907 kb)
补充表3-section米
这个文件包含GWAS SNP对智慧协会在TF编码注释。(TXT 2395 kb)
补充表第一章N
这个数据文件显示了编码TF详细分类。(XLS 34 kb)
补充表第一章P
这个数据文件显示了编码数据生产总结。(XLS 38 kb)
补充表第一章问
这个数据文件显示了编码元素数量和长度的数据类型。(XLS 323 kb)
权利和权限
本文根据分布Creative Commons Attribution-Non-Commercial-Share都许可证(http://creativecommons.org/licenses/by-nc-sa/3.0/)。
关于这篇文章
引用这篇文章
邓纳姆,我。,Kundaje, A., Aldred, S.et al。一个集成的百科全书在人类基因组中DNA的元素。自然489年,57 - 74 (2012)。https://doi.org/10.1038/nature11247
收到了:
接受:
发表:
发行日期:
进一步的阅读
针对异常的RAS /皇家空军/ MEK / ERK信号对癌症治疗
细胞(2020)
微rna - 425 - 5 - p抑制肺癌细胞生长在体外和体内的表达下调TFIIB-Related因素2
技术在癌症研究和治疗(2020)
临床意义的原发性开角型青光眼遗传学的最新进展
眼睛(2020)
细胞分化:50年来我们学到了什么?
理论生物学杂志》上(2020)
大麻素CB2受体转录因子NRF2特定调制的小胶质细胞
细胞和分子神经生物学(2020)