文摘
进化甲型流感病毒与人体免疫系统相互作用,表现为病毒血凝素抗原漂移”,是一个最好的描述分子进化中的模式。然而,人们很少知道公司该病原体的进化动力学。同样,基因组如何处理与全球流感流行病学、中/ H3N2和A / H1N1亚型co-circulate,了解甚少。通过分析1302年完整的病毒基因组样本从温带种群在两个半球,我们表明,甲型流感病毒的基因组演化特征是一个复杂的相互作用频繁的重组和周期性选择性清洁工。/ H3N2和A / H1N1亚型展览不同的进化动力学,与不同的血统在A / H1N1病毒的传播,表明弱抗原漂移。这些结果表明sink-source病毒生态学模型从持久的流感新血统的水库,我们假设位于热带地区,人口在温带地区。
主要
流感是一种最重要的人类呼吸道感染,负责每年250000到500000人死亡1。三种类型的流感病毒,A型是最致命的、与季节(冬季)流行病在温带地区,热带地区传播的更加持久2,和偶尔的大规模的全球大流行的特点是发病率和死亡率增加。
自1918年全球大流行亚型H1N1甲型流感病毒引起的,五个基因组片段内保持一个完整的进化历史humans-those编码核衣壳蛋白(NP),基质蛋白(M1和M2)和非结构蛋白(NS1 NS2),和两个编码聚合酶蛋白(PB2和PA)3,4。相比之下,新血凝素(HA)和神经氨酸酶(NA)表面糖蛋白,PB1聚合酶,所获得的人类流感病毒通过与禽流感病毒的重组。这些收购恰逢全球大流行;HA亚型H2和NA亚型N2出现在1957年,HA H3亚型出现在1968年,一个新的PB1段在1957年和1968年收购。虽然A / H1N1病毒重新出现在1977年(ref。5)和继续流通,季节性流感病毒1968年以来由/ H3N2病毒6,不时抗原进化的特征7。
尽管最近完整的基因组序列数据的可用性8,许多方面的甲型流感病毒的进化和流行病学动态保持不透明。特别是,没有严格的病毒多样性跨越时间的测量,在空间和亚型。此外,大多数演化动力学的研究都聚焦于单一领域,没有探索它们之间的交互。虽然比较研究表明,重组频繁出现在/ H3N2(参考文献9,10)之间的内部和/ H2N2和A / H1N1亚型11,12,13,在禽流感病毒14的全面重组,以及它如何与抗原进化,还未确定。在天平的流行病学,虽然很明显,一个/ H3N2和A / H1N1经验振荡季节性动态,确定这种周期性的部队,以及他们如何不同空间,是未知的。
来帮助解决这些问题,我们详细研究了甲型流感病毒的基因组的进化动态和流行病学鳞片。使用一个数据集的1302 / H3N2和a / H1N1隔离取样12-yr一段,说明大量在温带地区的北部(美国纽约州)和南部(新西兰)半球,我们量化中甲型流感病毒亚型的基因多样性,基因组片段和地理位置。
动态的流感基因多样性
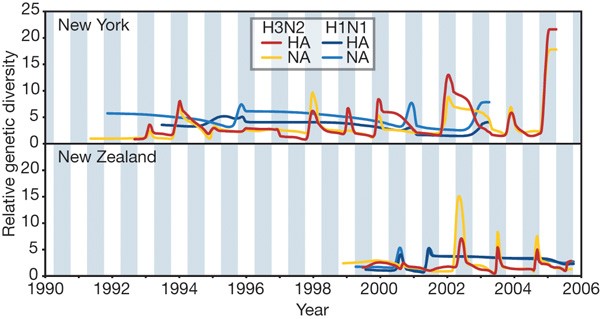
遗传多样性的变化模式在纽约州病毒分离和新西兰清楚地揭示了季节性甲型流感在个别温带种群动力学(图1)。对于better-sampled / H3N2亚型,暴露我们的模式coalescent-based年度系列的分析遗传多样性的山峰还夹杂着强烈的遗传瓶颈大多数流感季节的结束。正如预期的那样,遗传多样性的纽约州流行高峰在北半球冬季,但新西兰的孩子却抵消了大约6个月,对应于南半球的冬天。观察(一个类似的模式从澳大利亚123个基因组序列添加时,表明这些模式是常见的其他温带的人群,看到的补充信息)。此外,遗传多样性在新西兰/ H3N2通常是低于在纽约州,可能反映了小主人人口在新西兰(大多数隔离取样从坎特伯雷,南岛)。宿主人口规模的差异也许可以解释为什么一个在新西兰/ H3N2多样有时低于A / H1N1在纽约州,即使临床流行病学/ H3N2通常占主导地位。绝对数量的遗传多样性,即使在季节性高峰,小于其他快速发展的病毒感染更少的人15,16,这表明强烈的自然选择,除了定期的瓶颈,减少了在任何时候co-circulates的多样性水平。模拟表明,遗传多样性的重建是健壮的采样协议(补充信息)。
更值得注意的是,在这两种人群,甲型H1N1流感季节表现出高度峰值定义多样性通常配合弱峰定义/ H3N2多样性(也就是说,疫情的措施“尖峰”/ H3N2和A / H1N1是负相关;Wilcoxon符号秩检验,W= 348,n= 32,P< 0.002;补充无花果1和2)。这意味着一个进化亚型之间的交互;例如,A / H1N1疫情是由群体免疫抑制/ H3N2占主导地位时,或者/ H3N2胜过了A / H1N1,也许由于更大的复制的健身。符合我们观察,A / H1N1只占主导地位在赛季后异常温和的H3N2流感流行17,感染亚型可以防止再感染和其他以连续的流行病18。此外,自1977年以来,A / H1N1流行展览死亡率低于/ H3N2流感(参考文献19和20.)和空间同步21,22,23。在两个半球,A / H1N1看起来也不太容易描述/ H3N2的季节性遗传瓶颈,这表明基因不同A / H1N1血统能更好地共存。这可能表明抗原选择行为效力较低A / H1N1,表现为较低的氨基酸固定在HA1 (ref。17),以便发生选择性营业额的血统更慢17,24。
持久性病毒多样性之间流行高峰/ H3N2和A / H1N1有两种解释:链感染在每个人口生存和inter-epidemic间隔,或遗传多样性是导入到温带人口每年。系统发育强烈重证据反对前者,因为很少有直接的系统发育关系甲型流感病毒在连续采样季节从特定位置,如预期原位进化是司空见惯25,26。此外,高风险的随机灭绝使连续重复生存的小型连锁店之间的感染流行不太可能。此外,纽约州的日期段血统的1999 - 2000赛季再次出现在2002 - 2003赛季,之后的主要瓶颈/ H1N1-dominant 2000 - 2001赛季,强烈建议多样性是维持在一个水库。
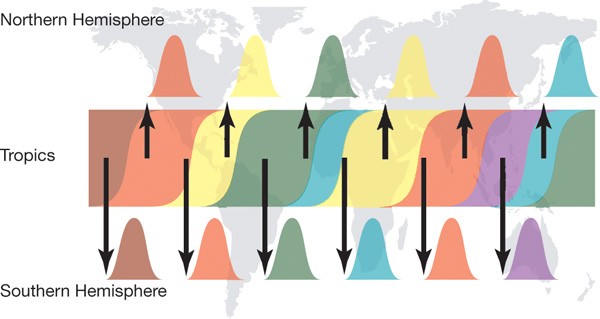
推断的进化动力学流感在纽约州和新西兰在北半球和南半球其他类似的人口让我们提出一个“源库”模式的全球生态甲型流感病毒(图2)。Continual-but主要unidirectional-gene流从常见的人口(或链接的源网络人群24,27)提供点燃每一个流行的病毒数量的北半球和南半球。尽管中国南方已被建议作为甲型流感病毒的中心28,有可能是热带地区通常表示人口因为扩展的病毒传播的理想来源2。这种模式的一个必要的后果是,selection-driven抗原漂移源人口将更有效,长期有效的大小由高背景感染率和维护的缺失严重人口瓶颈与温带地区2。因此,抗原变化的观察在温带种群内的选择是一个次要的影响,和基因流从源人口。这解释了存在频繁的积极选择的矛盾在全球范围内跨季节29日,30.,31日但几乎没有证据表明抗原漂移在个别季节性流感疫情的规模这些温带人群研究到目前为止26,32。为了测试这个假说,它将必须获得更多流感病毒样本热带地区,如东南亚。
基因多样性和重组
确定人类流感的全基因组进化动力学,我们推断出每个部分的人口遗传历史/ H3N2病毒取样的纽约州(最大的数据集)。这一分析表明,在任何给定的时间,个人部分可以相对遗传多样性上有本质上的区别,因此在系统发育历史(补充图3)。
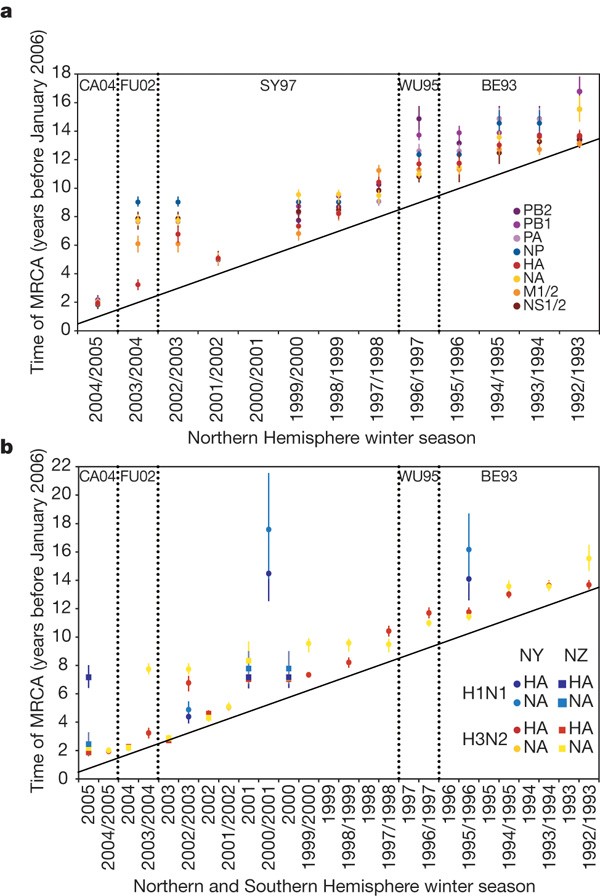
确定这些基因多样性的差异的原因,我们估计时间最近的共同祖先(TMRCA)每个流感季节的每一段(图3;估计后验密度最高的间隔时间所示补充图4)。大多数TMRCAs远远在赛季开始前,他们采样,这样多个血统存在多个流行波谷,符合我们源库模型。TMRCAs也不同年段和,反映基因组重组的互动过程,自然选择和基因流。例如,NP的TMRCAs段通常高于HA段,表明遗传多样性持续更长时间在前。这些变化模式的多样性提供了一个独特的洞察全基因组进化过程。特别是,2001 - 2002赛季的特点是减少TMRCAs整个/ H3N2基因组(占主导地位的A / H1N1流感季节后),所有片段有大约相同的TMRCA,强烈建议全基因组选择扫描。在接下来的2002 - 2003赛季,遗传多样性重新出现在基因组规模,因此,所有部分都TMRCAs约会不晚于1999 - 2000。随后,2003 - 2004年,HA段单独TMRCA减少,表明它接受选择性扫描,重组维护其它领域的多样性。另一个全基因组选择扫描是反映在严格TMRCAs 2004 - 2005。
一个,b,时间最近的共同祖先(TMRCA)的每一个基因片断/ H3N2分离循环每一季在纽约州(一个)和血凝素和神经氨酸酶基因片段/ H3N2和A / H1N1隔离的循环在纽约州和新西兰(b)。所代表的值意味着TMRCAs估计后验密度最高,95%的间隔采样获得使用贝叶斯模型分析整个树。显式的后验概率的任何季节是否有TMRCA比前的每一个季节了补充表1。每个季节的对角线穿过1月1日(7月1日新西兰),近似季节性中点。重大变化的时间尺度抗原(HA)类型在美国也描绘。在北半球,单个流感季节跨越了两年,而在南半球它们包含在一个日历年度。
分析A / H1N1病毒的血凝素和神经氨酸酶在纽约州和新西兰揭示了一个类似的模式的多样性变化(图3 b)。然而,TMRCAs往往远远大于那些观察/ H3N2同年,提供进一步的证据表明,更大的变化持续流行季节中A / H1N1甲型H3N2。这可能是由于有效的人口规模较小或较弱的免疫选择a / H1N1,这样自然选择是有效地修复有利变异少。
重组的发生和自然选择也正值HA抗原性的变化(图3)。最早的抗原转变我们的数据是北京/ 1993 (BE93)——和武汉之间/ 1995 (WU95)——病毒7。这是与急剧增加的TMRCA PB2在1996 - 1997赛季,由于再现家族现在直到1994 - 1995年,加上重组。同样,WU95悉尼/ 1997——就像病毒(SY97)转变的同时,显著减少M1/2以外的所有片段的遗传多样性,重组已分离的这部分的基因组,而获得新的HA血统的时候SY97福建/ 2002——就像病毒(FU02)抗原的变化33。最后,全基因组选择的2004 - 2005年正值FU02加州/ 2004 (CA04)——就像病毒抗原转变。
进化之间的交互部分
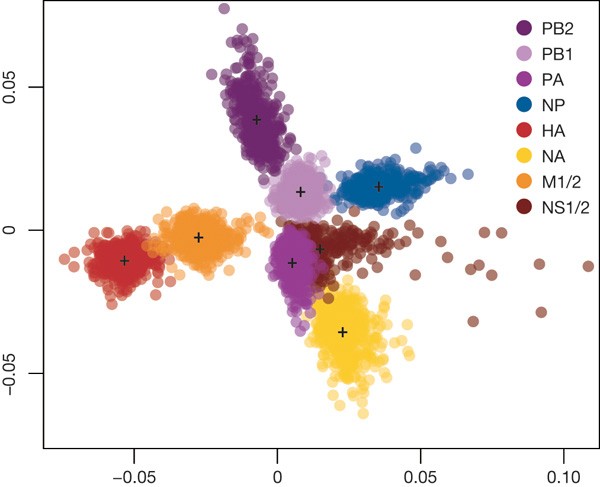
我们用多元统计总结的系统发育历史的差异片段/从纽约州H3N2病毒(图4和补充图12)。每个云点相同的颜色对应于一个特定的流感病毒基因组,云内的传播分代表统计系统的不确定性这段历史。显然,历史片段之间的差异大于每个部分的估计误差。最突出的结果是不同位置的哈,特别是与NA,即使表面糖蛋白编码。这些差异在进化历史兼容频繁的重组,这表明HA段一直放置在新的基因组背景,可能极大地影响病毒健身。
一个多维定标块样品每一部分的500棵树之间的距离从纽约州/ H3N2病毒取样。树与树之间的距离计算的相关系数向量的1 - TMRCAs每个季节。每个云点相同的颜色代表了统计的不确定性的系统发育历史个体基因组片段(每段的质心分布表示)。考虑到每个部分之间以成对的方式比较揭示出了一个类似的模式(补充图12)。
相比之下,有明显的相似系统发育历史的HA和M1/2段,兼容HA结构相互作用的观测在病毒装配和M1, M2离子通道平衡pH值对最优融合34,35,36,37,38。系统发育历史上还有一个相似在NS和NP段,和一定程度上的PB2, PB1和PA聚合酶复杂部分,建议他们受到不同程度的物理联系。阐明这些联系的生理基础模式无疑会提供信息关于大流行性流感病毒株的出现。
进一步洞察段提供的交互分析的进化率/ H3N2隔离从纽约州(补充图5)。我们确认主要比例的区别这些片段表达病毒粒子表面(高)和那些只有内部函数(低利率);例外是NS段,只有NS2蛋白的一个小病毒粒子组件。如预期鉴于免疫逃避它的角色,HA蛋白展品进化变化的总体率最高(平均为5.72×103核苷酸替换每个网站每年每年每网站(潜艇),95%最高的后验密度区间5.17,6.28×103潜艇每年每网站),以及产生的替代率最高,相对利率表示的第一和第二和第三个密码子的位置。更令人吃惊的是,M1/2 NS1/2和NA段进化速度重叠的HA(平均为5.20×103潜艇每年每站点(95%最高的后验密度区间4.36,6.07×103潜艇每年每网站),5.39×103(4.48,6.35×103)和5.41×103(4.81,5.99×103),分别),尽管产生变化的利率较低。相比之下,构成病毒聚合酶的三个部分,以及rna结合NP,明显表现出更低的利率在所有网站和在第一和第二密码子位置,揭示强选择性约束对氨基酸变异。
因为NA和HA段有不同的系统发育历史,他们的相似之处整体进化速度不太可能完全由于遗传连锁。因此,NA也可能受到相对强劲的积极的选择,对抗原变异或功能兼容公顷39。免疫介导的选择也可以解释在M1/2和NS1/2增加利率。M2离子通道的细胞外的领域是一个关键的目标发展的通用流感疫苗40,而M1蛋白是病毒衣壳的主要成分和细胞免疫反应的潜在目标41。同样,NS1编码多效性的,非结构蛋白会使double-stranded-RNA-induced抗病毒反应42,或者,也许同时共同进化出与PB1段观察过去两大流行30.,43。
结论
我们的分析表明,甲型流感病毒的进化动力学是由快速突变之间的复杂的相互作用,频繁的重组,广泛的基因流,自然选择(偶尔生成全基因组选择清洁工),功能之间的交互部分,和全球流行病动力学。虽然在HA支持进化的增加率模型,这种蛋白质受到强大的免疫逃避,选择在其他virion-associated进化变化的高蛋白质(有不同的系统发育历史公顷)表明,他们是同样的积极选择。这种适应的一个重要组件可能需要优化的功能兼容性段;例如,一个新的HA变体可能不会增加健身,除非它与功能兼容的部分。这种现象可以解释为什么血统导致FU02抗原类型没有主导病毒人口直到几年首次出现后和co-incident HA重组事件33。
同样引人注目的是基因重组的高度及其与周期选择性清洁工。因此,至关重要的上下文中考虑抗原漂移的过程中频繁的重组和全基因组上位相互作用。重组抗原小说哈变异成不同基因背景的地方,其中一小部分可能恢复,甚至增加,病毒复制的健身中可能丢失由于HA的变化。虽然不太可能重组率本身之间的不同部分,很可能,大多数涉及某些段组合的重组deleterious-such涉及HA和M1/2似乎相对紧密联系,通过净化被选择,导致系统发育历史发现的相似点和不同点。
除了全基因组的相互作用,有必要考虑的复杂的空间流行病学动态流感如果我们要充分理解抗原进化。我们观察到一致的两个种群动力学模式说明在北半球和南半球温带地区,一起跨多个流行病毒性血统的持久性。要解决这些矛盾倾向的观察,我们建议连续水库或源人口的存在,在强有力的选择抗原变化发生。这种复杂性必然意味着任何个人血统的流感病毒的长期成功不仅依赖其抗原特性也在其复制的能力,其遗传性和环境因素,也许背后的季节性流感在温带地区。在这种情况下,重要的是要确定抗原的准确原因进化在a / H1N1收益减少的速度,这样多个血统co-circulate在全球范围内,尽管这个亚型偶尔季节性流感的主要原因。
方法总结
完整的基因组序列/ H3N2和A / H1N1流感病毒从纽约州,美国和新西兰,从NCBI流感整理数据库作为流感基因组测序项目的一部分8,44。表示每个基因片段的序列比对,病毒亚型和取样部位聚集。演化动力学为每个数据集使用建立了贝叶斯估计马尔可夫链蒙特卡罗(密度)的方法45,46病毒样本包含的确切的一天。获得每个模型分析导致边缘后验估计:核苷酸替代率(补充图5);相对进化速度第一和第二和第三个密码子的位置(补充图5);和种群遗传多样性的动态时间(见图1和补充图4)。此外,估计后验分布的家谱相关序列数据推断同时为每个对齐。从这些分布,我们获得的边际后估计每个基因组的TMRCA在每个流感季节段(图3 a, b)。node-height分布差异的每个组后的家谱可视化使用多维标度(图4)。每个系谱分布的节点高度和拓扑结构进行了综述使用最大进化枝支持方法(补充图3)。
网上的方法
检查之间的交互的人类流感病毒H1N1和H3N2亚型和北半球和南半球的季节性波动,我们编制了血凝素和神经氨酸酶基因(只编码区域)A / H1N1在纽约州(81 1995年和2003年之间的隔离取样)和新西兰(127年2000年和2005年之间的隔离取样),以及A / H3N2在纽约州(687 1993年和2005年之间的隔离取样)和新西兰(407隔离在2000年和2005年之间)。强烈季节性流感在这两个地方,很少有报告病例以外的各自的冬天(世界卫生组织“FluNet”数据监测系统;http://gamapserver.who.int/GlobalAtlas/home.asp)。详细检查全基因组进化过程,我们进行了等效分析每一部分的687隔离/ H3N2采样的纽约州。对于每个部分,编码蛋白质的区域提取和对齐。与多个基因片段(M1/2和NS1/2),这两个基因串接在一起,重叠的基码(见只包含一次补充信息)。在所有情况下,定位明确,导致以下数据集:PB2(2277核苷酸),PB1(2271核苷酸),PA(2148核苷酸),公顷(1698核苷酸),NP(1494核苷酸),NA(1407核苷酸),M1/2(979核苷酸)和NS1/2(835核苷酸)。病毒分离株的完整列表,连同他们的基因库加入数字,是可用的补充信息。
我们使用一个灵活的统计模型来研究遗传多样性的季节变化在采样时间跨度47。这种方法,基于合并48,允许整个遗传多样性的人口估计从一个小样本的基因序列。估计随着时间的推移发生变化的多样性可以获得对于一个给定的家谱47。个人估计从许多不同的家谱结合,获得与家谱和参数值根据它们的相对概率抽样,给定的序列数据45,46。在缺乏自然选择,获得的遗传多样性指标反映了有效的感染数量随着时间的变化(Neτ,在那里τ平均一代时间)。因为强烈的自然选择曾被证实对甲型流感病毒,我们解释这些情节相对遗传多样性的措施。野兽包中实现的获得方法的优点在于,它不是条件单一,和潜在的代表性,估计潜在的家谱。它提供边际替代过程的所有参数的后验估计包括整体的分子进化速率,和《纽约时报》的所有节点genealogy-a时间表由已知的通知日期(天)的采样序列以及它们之间的基因变化45,49。的确,已经采样序列的使用在许多季节允许波动动力学有限的重建最早的日期(见样例补充信息)。
对这些分析中,我们使用一个nucleotide-substitution模型进化适应的不同利率和限制在不同的密码子的位置50。这让我们来估计总体每个基因的分子进化速率,和相对的第一和第二密码子的三和弦的测量位置约束对氨基酸变化与产生比强烈相关的核苷酸替换50。
我们调查的影响之间的重组部分通过比较相关的系统发育树独立采样段。每棵树,我们发现最近共同祖先的样品从每个季节和绘制边缘后验概率分布的时间节点(图3)。此外,我们估计的相关系数1 - TMRCAs内部和之间的一个样本的500棵树获得后验分布的密度分析每个基因片段。这些关系是绘制使用多维尺度降维技术(图4)和双向补充图12。
每段对齐,我们使用两个独立运行的野兽100000000步抽样树木每10000步骤和参数。比较两个运行确认都聚集,抽样分布相同,,然后被组合(删除10%作为“老化”)。这里所有的结果总结了从剩下的18000棵取样或参数值。
引用
- 1
谁。简报211号。流感。。<http://www.who.int/mediacentre/factsheets/fs211/> (2003)
- 2
Viboud C。,Alonso, W. J. & Simonsen, L. Influenza in tropical regions.科学硕士。3e89 (2006)
- 3
里德,a . H。,Taubenberger, J. K. & Fanning, T. G. Evidence of an absence: the genetic origins of the 1918 pandemic influenza virus.启Microbiol性质。2909 - 914 (2004)
- 4
陶本伯杰,j . k . et al . 1918年流感病毒聚合酶基因的特征。自然437年889 - 893 (2005)
- 5
徐先生,C。,von Hoyningen, V. & Rott, R. Genetic relatedness between the new 1977 epidemic strains (H1N1) of influenza and human influenza strains isolated between 1947 and 1957 (H1N1).病毒学89年613 - 617 (1978)
- 6
陶本伯杰,j·k·& Morens d . m . 1918年流感:母亲的大流行。紧急情况。感染。说。12第15 - 22 (2006)
- 7
史密斯,d . j . et al。流感病毒的抗原和遗传进化的映射。科学305年371 - 376 (2004)
- 8
Ghedin、大肠等。人类流感的大规模测序揭示病毒基因组进化的动态特性。自然437年1162 - 1166 (2005)
- 9
林德斯特伦,美国大肠et al。整个基因组的系统发育分析来自日本的甲型流感病毒(H3N2): 6内部基因的遗传重组的证据。j .性研究。72年8021 - 8031 (1998)
- 10
Schweiger B。,Bruns, L. & Meixenberger, K. Reassortment between human A(H3N2) viruses is an important evolutionary mechanism.疫苗246683 - 6690 (2006)
- 11
格里高利,诉et al。出现甲型流感H1N2重组病毒在人口2001人。病毒学300年1 - 7 (2002)
- 12
林德斯特伦,美国E。,Cox, N. J. & Klimov, A. Genetic analysis of human H2N2 and early H3N2 influenza viruses, 1957–1972: evidence for genetic divergence and multiple reassortment events.病毒学328年101 - 119 (2004)
- 13
年轻,j . f . & Palese p .进化的人类流感病毒在自然界:重组导致H1N1病毒的遗传变异。Proc。《科学。美国76年6547 - 6551 (1979)
- 14
Macken, c。,Webby,R. J. & Bruno, W. J. Genotype turnover by reassortment of replication complex genes from avian influenza A virus.j .性研究。87年2803 - 2815 (2006)
- 15
卡灵顿,C。福斯特,J。Pybus, O。,Bennett, S. & Holmes, E. Invasion and maintenance of dengue virus type 2 and type 4 in the Americas.j .性研究。79年14680 - 14687 (2005)
- 16
Lemey, P。,兰姆伯特,一个。&Pybus, O. HIV evolutionary dynamics within and among hosts.艾滋病牧师。8125 - 140 (2006)
- 17
狼,我y。,Viboud C。,Holmes, E. C., Koonin, E. V. & Lipman, D. J. Long intervals of stasis punctuated by bursts of positive selection in the seasonal evolution of influenza A virus.医学杂志。直接。134 (2006)
- 18
Sonoguchi, T。Naito, H。Hara, M。,Takeuchi, Y. & Fukumi, H. Cross-subtype protection in humans during sequential, overlapping, and/or concurrent epidemics caused by H3N2 and H1N1 influenza viruses.j .感染。说。151年81 - 88 (1985)
- 19
个性,M。,W一个tanabe, A. & Aizawa, H. Differences in clinical features between influenza A H1N1, A H3N2, and B in adult patients.Respirology8231 - 233 (2003)
- 20.
赖特,p F。,Thompson, J. & Karzon, D. T. Differing virulence of H1N1 and H3N2 influenza strains.点。j .论文。112年814 - 819 (1980)
- 21
干草,a·J。,Gregory, V., Douglas, A. R. & Lin, Y. P. The evolution of human influenza viruses.菲尔。反式。r . Soc。Lond。B356年1861 - 1870 (2001)
- 22
Finkelman et al。全球模式在季节性H3N2流感活动,A / H1N1,和B从1997年到2005年:病毒共存和纬向梯度。《公共科学图书馆•综合》2e1296 (2007)
- 23
Viboud, c . et al .同步,波浪,和空间层次结构在流感的传播。科学312年447 - 451 (2006)
- 24
弗格森:M。,Galvani, A. P. & Bush, R. M. Ecological and immunological determinants of influenza evolution.自然422年428 - 433 (2003)
- 25
纳尔逊,我m。,Simonsen, L., Viboud, C., Miller, M. A. & Holmes, E. C. Phylogenetic analysis reveals the global circulation pattern of seasonal influenza A viruses.公共科学图书馆Pathog。3e131 (2007)
- 26
纳尔逊,麻省理工学院et al .随机过程是短期内甲型流感病毒进化的关键决定因素。公共科学图书馆Pathog。2e125 (2006)
- 27
阿隆索,w . j . et al .季节性流感在巴西:行波从亚马逊到亚热带。点。j .论文。165年1434 - 1442 (2007)
- 28
Shortridge k . f .中国流感中心吗?下巴。地中海。j .(英格兰)。110年637 - 641 (1997)
- 29日
布什,r . M。,惠誉,w . M。,Bender, C. A. & Cox, N. J. Positive selection on the H3 hemagglutinin gene of human influenza virus A.摩尔。杂志。另一个星球。161457 - 1465 (1999)
- 30.
惠誉,w . M。,Leiter, J. M., Li, X. Q. & Palese, P. Positive Darwinian evolution in human influenza A viruses.Proc。《科学。美国88年4270 - 4274 (1991)
- 31日
施,a . C。,Hsiao, T. C., Ho, M. S. & Li, W. H. Simultaneous amino acid substitutions at antigenic sites drive influenza A hemagglutinin evolution.Proc。《科学。美国104年6283 - 6288 (2007)
- 32
Lavenu, a . et al。详细分析流感病毒的遗传进化过程中一个流行。论文。感染。134年514 - 520 (2006)
- 33
福尔摩斯,e·c·等。人类流感病毒全基因组分析揭示了多个持久化血统和重组最近H3N2病毒。公共科学图书馆杂志。3e300 (2005)
- 34
阿里,一个。,Avalos, R. T., Ponimaskin, E. & Nayak, D. P. Influenza virus assembly: effect of influenza virus glycoproteins on the membrane association of M1 protein.j .性研究。74年8709 - 8719 (2000)
- 35
酒吧间招待员,S。阿里,。,Hui, E. K., Adhikary, L. & Nayak, D. P. Transport of viral proteins to the apical membranes and interaction of matrix protein with glycoproteins in the assembly of influenza viruses.病毒Res。77年61 - 69 (2001)
- 36
Enami, m & Enami k .流感病毒血凝素和神经氨酸酶膜糖蛋白刺激关联矩阵的蛋白质。j .性研究。70年6653 - 6657 (1996)
- 37
Ohuchi, m . et al .救援向量来禽病病毒的血凝素的生物活性形式acidotropic代理和coexpressed M2蛋白质。j .性研究。68年920 - 926 (1994)
- 38
竹内,k和羊肉,r . a .流感病毒M2蛋白质离子通道活动稳定的原生形式家禽瘟疫病毒血凝素在细胞内的运输。j .性研究。68年911 - 919 (1994)
- 39
Mitnaul l . j . et al .平衡血凝素和神经氨酸酶活动是有效的甲型流感病毒复制的关键。j .性研究。74年6015 - 6020 (2000)
- 40
格,W。,Mozdzanowska, K. & Zharikova, D. Prospects for universal influenza virus vaccine.紧急情况。感染。说。12569 - 574 (2006)
- 41
Berkhoff e . g . et al .功能约束的甲型流感病毒抗原表位限制逃避细胞毒性T淋巴细胞。j .性研究。79年11239 - 11246 (2005)
- 42
克鲁格,r . M。元,W。,Noah, D. L. & Latham, A. G. Intracellular warfare between human influenza viruses and human cells: the roles of the viral NS1 protein.病毒学309年181 - 189 (2003)
- 43
路德维格。,Schultz, U., Mandler, J., Fitch, W. M. & Scholtissek, C. Phylogenetic relationship of the nonstructural (NS) genes of influenza A viruses.病毒学183年566 - 577 (1991)
- 44
流感病毒基因组测序项目<http://www.niaid.nih.gov/dmid/genomes/mscs/influenza.htm> (2007)
- 45
德拉蒙德,a·J。,Nicholls, G. K., Rodrigo, A. G. & Solomon, W. Estimating mutation parameters, population history and genealogy simultaneously from temporally spaced sequence data.遗传学161年1307 - 1320 (2002)
- 46
德拉蒙德,a·j·兰姆伯特& a野兽:贝叶斯进化分析抽样树。BMC另一个星球。医学杂志。7214 (2007)
- 47
德拉蒙德,a·J。兰姆伯特,。,夏皮罗,B。&Pybus, O. G. Bayesian coalescent inference of past population dynamics from molecular sequences.摩尔。杂志。另一个星球。221185 - 1192 (2005)
- 48
金曼,j .合并。随机过程。达成。13235 - 248 (1982)
- 49
兰姆伯特,估计分子进化的速度:合并non-contemporaneous序列最大似然的发展史。生物信息学16395 - 399 (2000)
- 50
夏皮罗,B。,兰姆伯特,一个。&德拉蒙德,a·J。Choosing appropriate substitution models for the phylogenetic analysis of protein-coding sequences.摩尔。杂志。另一个星球。237号到9号(2006)
确认
这项研究的部分支持由美国国立卫生研究院的校内研究项目,Fogarty国际中心,国家过敏症和传染病研究所和国家综合医学科学研究所。境和O.G.P.受到英国伦敦皇家学会的支持。境是一个跨学科的一部分人类和禽流感研究中心(ICHAIR)。
作者的贡献境和E.C.H.构思。境和O.G.P.进行的基因组序列分析和开发方法。E.C.H.,A.R. and O.G.P. wrote the paper. M.I.N., C.V. and J.K.T. provided epidemiological and virological data. All authors interpreted the results and commented on the paper.
作者信息
从属关系
相应的作者
权利和权限
关于这篇文章
引用这篇文章
兰姆伯特,一个。Pybus, O。,Nelson, M.et al。人类流感病毒的基因组和流行病学动态。自然453年,615 - 619 (2008)。https://doi.org/10.1038/nature06945
收到了:
接受:
发表:
发行日期:
进一步的阅读
基因流行病学和积极监测调查汉坦病毒的暴发
感染细胞和微生物学前沿(2021)
SARS-CoV-2基因组从俄克拉何马州,美国
遗传学前沿(2021)
在硅片的预测序列分析流感疫苗的有效性
疫苗(2021)
证据的强度增加国际米兰还是季节性流感,昆士兰,澳大利亚,2009还是2019
流感及其他呼吸道病毒(2021)
自适应进化的证据在季节性冠状病毒的受体结合域OC43和229 e
eLife(2021)