文摘
流感病毒heterotrimeric聚合酶复杂(PA、PB1 PB2)是参与病毒复制的许多方面和与宿主因素进行交互1在宿主特异性,从而拥有一个角色2,3。从1918年人类流感病毒聚合酶蛋白序列不同于禽流感共识在只有少量的氨基酸序列,与假设一致的是,他们来自禽源前不久大流行。然而,当鸟类序列相比,1918年的聚合酶基因的核苷酸序列的差异大于预期,表明进化距离已知禽流感毒株。这里我们提出和系统发育分析的完整基因组序列的1918年流感病毒4,5,6,7,8并提出,1918年病毒不是一个重组病毒(如1957年和1968年的大流行9,10),但更有可能的是一个完全avian-like病毒适应人类。这些数据支持之前的系统发育研究表明,1918年病毒是来自一个禽源11。共有十个氨基酸的变化聚合酶蛋白持续1918年和随后的人类流感病毒序列有别于禽流感病毒序列。值得注意的是,许多相同的变化被发现在最近循环,高致病性H5N1病毒引起的疾病和死亡在人类和害怕是一个新的流感大流行的前兆。确定的序列变化可能是重要的在人类流感病毒的适应性。
主要
A型流感病毒引起人类和家畜年度爆发。定期,新的毒株出现在人类导致全球大流行。严重的“西班牙”流感大流行的数亿1918 - 1919感染,并导致了约5000万人的死亡12。我们以前使用的系统发育分析来帮助理解大流行性病毒的起源8,11;功能理解1918年病毒的致病性研究正在进行中6,13,14,15,16,17。最近的数据表明,病毒结构轴承1918血凝素基因致病在老鼠模型中,但尚未观察到的遗传基础映射6,13,14,15,16,17。这个项目的总体目标是理解的起源和不寻常的1918年流感病毒的毒性。
流感病毒聚合酶功能作为heterotrimer PB2形成,PB1和PA蛋白(见参考。1审查)。一个额外的小最近开放阅读框,编码的肽(PB1-F2)认为在病毒诱导细胞死亡中发挥作用18。目前尚不清楚如何聚合酶复杂必须改变以适应一个新的主持人3。单个氨基酸的变化PB2 E627K,显示(1)为哺乳动物的适应是很重要的2,3,(2)区分高致病性禽流感(HPAI)在小鼠的H5N1病毒19,(3)出现在单一致命的人类感染高致病性禽流感H7N7爆发2003年在荷兰(ref。20.),在最近的一些H5N1分离人类在越南和泰国,在中国野生鸟类21,22,23。
开放阅读框的部分序列1 (PB2)段2 (PB1)和段胡尔汀3 (PA) / / 1/1918,和理论的四个翻译确定阅读框架,所示补充图1 a - c。1918年PB2蛋白从鸟类的共识序列(包含五个更改表1)。其中,A199S区域映射为PB1绑定网站,和L475M变化是在核本地化的信号24,25,26。其他三个变化残留567年、627年和702年发生在网站没有已知的功能域。
1918 PB1蛋白质由七个残留物(不同于禽流感共识所示之一表1;另请参阅补充图2)。这些,K54R重叠绑定域名互补(c) RNA, RNA病毒(v)。变化残留375年、383年和473年之间的所有发生在四个守恒的聚合酶图案的cRNA绑定域名27,在残留变化576年、645年和654年发生在vRNA绑定域名28。
七变化被发现在1918 PA蛋白与禽流感共识(4所示表1,其他三个C241Y K312R和I322V)。C241Y变化发生在一个核定位信号,但其他六个变化(残留55岁,100,312,322,382和552年)发生在网站之外的功能域24,25,26。
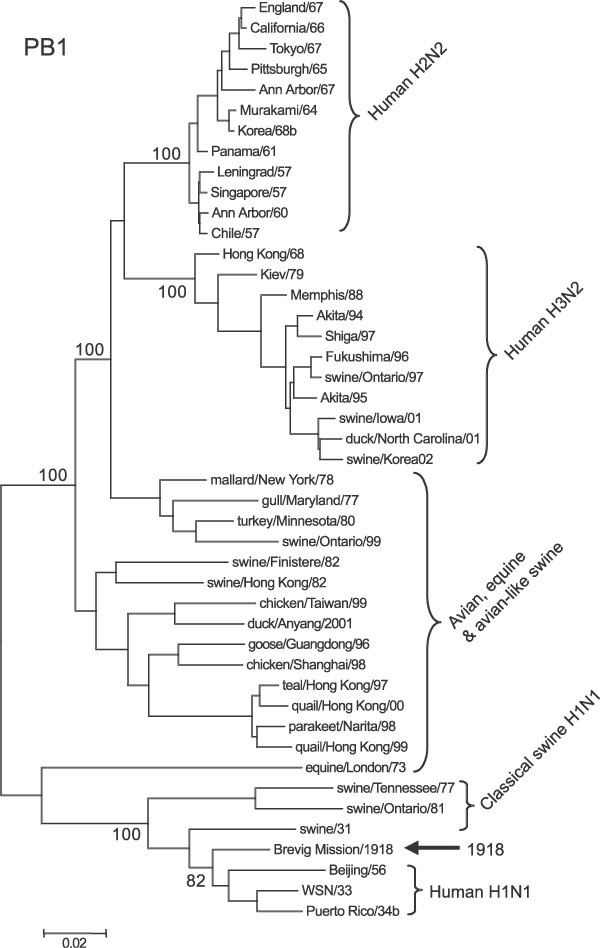
代表三个聚合酶基因的系统发育分析无花果所示1- - - - - -3。1918年人类大流行性流感病毒聚合酶基因比较代表禽流感基因对于过渡/颠换(T我/Tv)比、同义和非同义(S / N)比在四倍简并地点和数量的差异(ref中定义。11)。T我/Tv比率对于大多数比较使用1918病毒基因和代表北美或欧亚禽流感基因序列2和4之间的值。这个范围是比较类似于观察各种鸟类基因,除了PB1基因。PB1,对比1918年的病毒基因与禽流感病毒PB1基因总是接近2,而比较各种鸟类基因彼此在6 - 10的范围。有更少的颠换比较禽流感PB1基因比对比PB1禽流感PB1和1918年人类病毒,可能反映了颠换更经常导致非同义的变化。
S / N比率对于大多数比较使用1918病毒基因序列和代表北美或欧亚禽流感基因通常产生值的范围7 - 16为PA和PB2基因,对于大多数鸟类和禽流感一样PA和PB2基因比较。就像T我/Tv比率,S / N比率偏高,PB1基因(大部分比较了比率在第16 - 25的范围),由于小数量的非同义禽流感PB1基因的变化比较。这些发现可能反映了更为保守的PB1在鸟类的进化。
的一个子集的差异发生在网站四倍退化(也就是说,一个替换任何基本不会导致一个氨基酸替换)。因为这些站点不受选择压力在蛋白质水平,基地在许多四倍简并替换站点可以迅速积累。如果流感病毒基因在鸟类进化足够长的时间达到进化停滞,所提出的上述较高的S / N比率,有人预测,在许多网站的四倍简并度是可行的,所有四个基地将会出现在鸟类进化枝除非RNA二级结构的约束限制的积累的变化。事实上,当鸟类序列从地理上不同的血统(北美和欧洲)比较,差异百分比在退化网站产生四倍值27 - 38%范围内。相比之下,计算每分差四倍简并的网站比较1918年的病毒PA, PB1和PB2基因序列与禽流感序列产生持续高值(范围41 - 51%)三个基因。与其他1918个基因11,这表明,1918年病毒进化的捐赠来源隔离与禽流感病毒目前在数据库中表示。
强调avian-like大自然1918年的流感病毒聚合酶蛋白,19日总氨基酸的变化从鸟类的共识,只有10个氨基酸位置(2232总密码子)始终区分人类1918年和随后的聚合酶蛋白PB2, PB1和PA禽流感同行(这些变化被定义为从禽流感病毒序列在1918年保持没有变化在随后的人类病毒)(表1)。很可能在人类适应这些变化有重要的作用。七这些十改变以前在禽流感和人类流感病毒聚合酶之间的对齐3。下面是对比1918年病毒变化和最近的H5N1病毒分离株,为了评估可能的平行进化的例子在禽流感病毒对人类的适应。
PB2蛋白,5变化区分人类隔离和禽流感序列(表1)。253可用PB2序列从人类H1N1, H2N2 H3N2隔离,这五个变化是几乎完全保留了,除了这两个最近的H3N2隔离禽流感赖氨酸残留在702位置。只有少量的禽流感病毒分离株表现出任何这五种变化,它是有趣的,几乎所有这些隔离来自高致病性禽流感H5N1病毒或H7N7病毒,或从人类的感染H9N2谱系少量在中国在1990年代末(ref。29日)。只有5个可用的282年禽流感PB2序列有Ser残留在199位置,四个来自香港的1997年H5N1病毒分离株。A199S变化也发现了5个18 H5N1菌株从人类分离(所有5个从1997年香港爆发)。的禽流感病毒,36 336有一个参数残留在702位置,其中30 H9N2隔离来自中国1996 - 2000左右,和5是H5N1病毒分离株从香港在1997年和2001年。可用的18 1997年H5N1菌株从人类分离,三个有K702R改变。
也许最有趣的是,1918年的病毒和随后的人类隔离有赖氨酸残留在627位置。这种残渣与主机适应2,3,曾被证明是至关重要的1997年老鼠感染了高致病性的H5N1病毒19。禽流感的隔离,19 345有赖氨酸残基位置627,18岁的高致病性禽流感H5N1病毒或H7N7禽流感病毒。16这些最近被H5N1分离的特点是死亡的野生水禽青海湖2005年在中国西部(ref。21)。在人类H5N1病毒分离株,11个37 E627K变化:/香港/ 483/1997 /香港/ 485/1997,四,六隔离在2004年从越南(ref。22),从2004年的泰国和三分之二的隔离(ref。23)。E627K突变被认为在六个H5N1病毒分离株2004年从泰国虎,也出现在H7N7病毒负责单一人类病死率高致病性禽流感期间H7N7爆发2003年在荷兰(ref。20.)。这不是在同生鸡隔离。
在475的位置,只有一个355年的禽流感分离有一个遇到渣(H5N1高致病性禽流感病毒从2004)。同样,只有一个345年的禽流感病毒有一个Asn残留在567位置。没有一个人类H5N1病毒分离株L475M或D567N变化。没有可用的H5N1病毒或H7N7序列有多个的human-adaptive PB2变化确定为1918年的病毒。
PA蛋白显示了类似的模式:四个残留始终在1918年和随后的人类隔离和不同鸟类的共识序列(表1)。三个其他变化(C241Y K312R和I322V)区分1918年H1N1和H2N2人类隔离,但大多数H3N2隔离有禽流感氨基酸在这些位置。295年可用的序列从人类H1N1 H2N2 H3N2隔离,都有Asn在位置55(/网络/ 33除外),阿拉巴马州在100年和552年Ser位置的位置。只有5 295人隔离有禽流感Glu残留在382位置。值得注意的是,这五个隔离的小进化枝最近的H3N2隔离,有许多不寻常的变化从典型的人类H3N2病毒30.。当禽流感序列分析,没有一个(209序列)Asn在552位置55或Ser位置。只有8 209年禽流感PA蛋白序列显示V100A变化:最近六H6N2隔离从鸡在加州,2002年和两个高致病性禽流感H5N1鸭分离株来自中国。209年禽流感的序列,五有一个Asp残留在382位置,其中包括两名高致病性禽流感H5N2隔离从1994年的墨西哥鸡。
PB1基因片段取代重组在1957年和1968年大流行9。我们比较PB1蛋白从1918年人类流感病毒与avian-derived PB1段从1957年和1968年大流行。人类H1N1 H2N2和H3N2病毒源自于1918年,1957年和1968年大流行,分别为每一个拥有独特的派生avian-like PB1基因片段,所以我们试图找出任何可能阐明人类适应平行变化。三位人类大流行性PB1蛋白质不同鸟类共识只有4 - 7残留物(补充图2)。只有一个共享这些变化在大流行性流感隔离:N375S变化。这种变化丝氨酸残基还发现在猪和马流感隔离。除了少数例外,所有人类流感PB1蛋白质有爵士在这个网站。230年人类流感序列,只有两个H1N1隔离(/ FM / 47 /北京/ 1956),上述小进化枝的H3N2隔离禽流感Asn残渣30.。相比之下,虽然这渣保持在几乎所有哺乳动物的隔离,它是变量之间的禽流感PB1蛋白质。293年禽流感分离,66%有共识Asn残留在375位置,18%有Ser残渣和12%有刺残渣。
这里给出的数据突出的标志保护PB1禽流感病毒的蛋白质。PB1函数作为一个依赖RNA的RNA聚合酶,所以它是合理的假设,其酶的功能是最佳的守恒形式。在人类,PB1蛋白质经验线性随时间变化。事实上,PB1在人类获得∼每年0.4氨基酸的变化。等有强烈的人类病毒抗原的选择,它是可能的,尽管PB1中的观察到的变化是有选择地对抗原性有益,他们温和有害酶的功能。如此复杂的健身权衡被认为是在RNA病毒进化。支持这一假说,最近的一项研究检查禽流感和人类流感病毒聚合酶的组合表明,最有效的流感转录活动在体外被认为与一个avian-derived PB1,即使PB2, PA和NP蛋白是来自人类的病毒3。收购的禽流感PB1重组可能会提供一个新病毒复制的优势,可能解释为什么这两个过去的两大流行流感病毒和1918年都有非常avian-like PB1蛋白质。
1957年和1968年大流行流感病毒禽流感/人类重组2 - 3禽流感基因片段的可行性以循环,人适应病毒9,10。不像1957年和1968年大流行,然而,1918年的病毒很可能不是一个人类/禽重组病毒,而是avian-like病毒适应人类在托托8,11。氨基酸置换率的基础上,在人类流感病毒聚合酶基因,可能这些片段流传在人类流感病毒早在1900年。然而,证明1918年的病毒没有保留从以前循环人类流感病毒基因片段需要发现一个样本的1918年以前病毒档案材料。供体来源,尽管avian-like蛋白质水平,可能来自禽流感病毒目前还没有代表的一个子集序列数据库,可能是进化的隔离。
氨基酸的变化这一事实确认在1918年的分析也在高致病性禽流感的H5N1毒株和H7N7禽流感病毒导致人类的死亡是有趣的,并表明这些变化可能促进人类细胞,增加致病性病毒复制。有可能是1918年的高pathogencity病毒相关的成为一个人适应禽流感病毒。这些变化可能反映了一个平行进化的过程为禽流感病毒变异adaptational压力,并表明禽流感病毒的遗传基础适应人类可以映射。
方法
RNA分离、扩增和测序
RNA是隔绝冰冻1918人肺组织使用试剂盒(英杰公司)根据制造商的指示。每个片段反向转录、放大和测序至少两次。逆转录聚合酶链反应(rt - pcr)、隔离的产品和测序曾被描述4。引物列表和引物序列可按照客户要求定制。独立产生RNA复制rt - pcr反应的准备了相同的序列结果。2280 -核苷酸的完整编码序列PB2放大在33个重叠的碎片。2274 -核苷酸编码序列的PB1放大在33个重叠的碎片。PA的2151 -核苷酸编码序列是放大在32个重叠的碎片。PCR产物的大小不等,从77 - 138个基点。
系统发育分析
系统发育分析的三种聚合酶基因都是使用标准的方法。我们生成树使用neighbour-joining (NJ)算法,与不同比例的距离测量使用大型版本2.1。角色演变分析与MacClade程序后吝啬分析利用PAUP 4.0 beta版本,使用ACTRAN作为优化方法。树木也使用最大似然生成中点加油。所有算法生成类似的树,主要代表人类演化支,古典猪和avian-like病毒(NJ树无花果所示1- - - - - -3;完整的数据集可以在请求)。聚合酶片段序列用于分析得到从基因库和流感序列数据库(ISD)。(见补充表1的序列使用。)83年PB2基因序列被使用,所有这些都是长度。91年PB1基因序列被使用,三是不完整的。105年PA基因序列,六是不完整。
引用
Fodor,大肠& Brownlee g . g .流感(ed。波特,c . w .) 1至29(爱思唯尔,阿姆斯特丹,2002)
苏巴拉奥,e·K。,London, W. & Murphy, B. R. A single amino acid in the PB2 gene of influenza A virus is a determinant of host range.j .性研究。67年1761 - 1764 (1993)
Naffakh, N。,Massin, P., Escriou, N., Crescenzo-Chaigne, B. & van der Werf, S. Genetic analysis of the compatibility between polymerase proteins from human and avian strains of influenza A viruses.j .性研究。81年1283 - 1291 (2000)
里德,a . H。,Fanning, T. G., Hultin, J. V. & Taubenberger, J. K. Origin and evolution of the 1918 “Spanish” influenza virus hemagglutinin gene.Proc。《科学。美国96年1651 - 1656 (1999)
里德,a . H。,Fanning, T. G., Janczewski, T. A. & Taubenberger, J. K. Characterization of the 1918 “Spanish” influenza virus neuraminidase gene.Proc。《科学。美国97年6785 - 6790 (2000)
巴斯勒,c . f . et al . 1918年大流行流感病毒非结构基因序列(NS)段和表征轴承1918 NS基因重组病毒。Proc。《科学。美国98年2746 - 2751 (2001)
里德,a . H。,Fanning, T. G., Janczewski, T. A., McCall, S. & Taubenberger, J. K. Characterization of the 1918 “Spanish” influenza virus matrix gene segment.j .性研究。76年10717 - 10723 (2002)
里德,a . H。,Fanning, T. G., Janczewski, T. A., Lourens, R. & Taubenberger, J. K. Novel origin of the 1918 pandemic influenza virus nucleoprotein gene segment.j .性研究。78年12462 - 12470 (2004)
Kawaoka Y。,Krauss, S. & Webster, R. G. Avian-to-human transmission of the PB1 gene of influenza A viruses in the 1957 and 1968 pandemics.j .性研究。63年4603 - 4608 (1989)
徐先生,C。罗德,W。,Von Hoyningen, V. & Rott, R. On the origin of the human influenza virus subtypes H2N2 and H3N2.病毒学87年13-20 (1978)
里德,a . H。,陶本伯杰,J。K. & Fanning, T. G. Evidence of an absence: the genetic origins of the 1918 pandemic influenza virus.启Microbiol性质。2909 - 914 (2004)
约翰逊:p &米勒,j .更新账户:全球死亡率的1918 - 1920年的“西班牙”流感大流行。公牛。嘘。地中海。76年105 - 115 (2002)
角膜,g·k . et al .细胞转录分析甲型流感病毒感染肺上皮细胞:非结构化的角色NS1蛋白的逃避宿主先天防御和其潜在的贡献大流行性流感。Proc。《科学。美国99年10736 - 10741 (2002)
汤佩使t . m . et al。现有抗病毒药物对流感病毒有效从1918年大流感病毒的基因。Proc。《科学。美国99年13849 - 13854 (2002)
汤佩使,t . m . et al .致病性和免疫原性的流感病毒与1918年大流感病毒的基因。Proc。《科学。美国101年3166 - 3171 (2004)
卡什,j . c . et al。全球宿主免疫反应:血凝素和神经氨酸酶基因的贡献从1918年西班牙流感病毒的发病机理。j .性研究。78年9499 - 9511 (2004)
Kobasa, d . et al。增强毒性的甲型流感病毒与1918年大流感病毒的血凝素。自然431年703 - 707 (2004)
陈威等。一种新型甲型流感病毒线粒体蛋白诱导细胞死亡。自然医学。71306 - 1312 (2001)
山中,k . et al . PB2氨基酸在627位置影响复制的效率,但并不是细胞趋向性,香港H5N1流感病毒在小鼠体内。病毒学320年258 - 266 (2004)
Fouchier, r . A . et al。禽流感病毒(H7N7)与人类结膜炎和急性呼吸窘迫综合征的致命病例。Proc。《科学。美国101年1356 - 1361 (2004)
陈,h . et al .禽流感:H5N1病毒爆发迁徙水鸟。自然436年191 - 192 (2005)
李,k . s . et al。《创世纪》的高致病性H5N1和潜在的大流行流感病毒在亚洲东部。自然430年209 - 213 (2004)
Puthavathana, p . et al .分子表征人类流感H5N1病毒完整基因组的隔离来自泰国。j .性研究。86年423 - 433 (2005)
丰田章男,T。,Adyshev, D. M., Kobayashi, M., Iwata, A. & Ishihama, A. Molecular assembly of the influenza virus RNA polymerase: determination of the subunit–subunit contact sites.j .性研究。77年2149 - 2157 (1996)
Masunaga所说,K。,Mizumoto, K., Kato, H., Ishihama, A. & Toyoda, T. Molecular mapping of influenza virus RNA polymerase by site-specific antibodies.病毒学256年130 - 141 (1999)
Ohtsu Y。,Honda, Y., Sakata, Y., Kato, H. & Toyoda, T. Fine mapping of the subunit binding sites of influenza virus RNA polymerase.Microbiol。Immunol。46167 - 175 (2002)
Biswas s . k . & Nayak d . p .突变的守恒的主题分析甲型流感病毒聚合酶碱性蛋白1。j .性研究。68年1819 - 1826 (1994)
冈萨雷斯,s & Ortin j .不同地区的流感病毒聚合酶PB1亚基承认vRNA和cRNA模板。EMBO J。183767 - 3775 (1999)
郭,y . j . et al .致病性的表征的新成立的H9N2流感病毒在亚洲血统。病毒学267年279 - 288 (2000)
福尔摩斯,e·c·等。人类流感病毒全基因组分析揭示了多个持久化血统和重组最近H3N2病毒。公共科学图书馆杂志。3e300 (2005)
确认
本报告中所描述的研究是使用严格的安全措施来保护实验室工作人员,这种病毒的环境和公众。本研究的目的是提供了解流感大流行毒株的基础形式,并帮助确定未来流感大流行的风险。本研究部分支持格兰特J.K.T.来自美国国立卫生研究院,校内基金从军事病理研究所的。本文所包含的观点是作者的私人看法和不被视为官方或反映的观点美国军队或美国国防部。作者贡献J.K.T.计划项目,A.H.R.,R.M.L.,R.W. and G.J. generated the sequence data. J.K.T., A.H.R. and T.G.F. performed data analysis. J.K.T. wrote the manuscript.
作者信息
从属关系
相应的作者
道德声明
相互竞争的利益
PB2的编码序列,PB1和PA基因已经被沉积在基因库加入数字DQ208309 DQ208310 DQ208311,分别。可在再版和权限信息npg.nature.com/reprintsandpermissions。作者声明没有竞争的经济利益。
权利和权限
关于这篇文章
引用这篇文章
陶本伯杰,J。里德,。,Lourens, R.et al。描述1918年的流感病毒聚合酶基因。自然437年,889 - 893 (2005)。https://doi.org/10.1038/nature04230
收到了:
接受:
发行日期:
DOI:https://doi.org/10.1038/nature04230
进一步的阅读
调查活禽市场的低致病性禽流感病毒在广西,中国南部,2016 - 2019
科学报告(2021)
古代DNA分析
自然评论方法引物(2021)
COVID-19疾病X和未来循环经济转型:重新设计大流行防备,以防止全球灾难
循环经济和可持续发展(2021)
1918年流感大流行的分析给提示想象可能的冠状病毒病级- 2019 (COVID-19) ?
转化医学杂志》(2020)
排名的哺乳动物pathogenicity-related PB2突变的禽流感病毒
科学报告(2020)